Servicios Personalizados
Revista
Articulo
Indicadores
Links relacionados
Compartir
Vive Revista de Salud
versión impresa ISSN 2664-3243
Vive Rev. Salud v.3 n.9 La Paz dic. 2020
INVESTIGACIÓN
https://doi.org/10.33996/revistavive.v3i9.54
Detección de mutaciones del gen 23S de Helicobacter pylori implicadas en la resistencia a claritromicina
Detection of Helicobacter pylori 23S gene involved in clarithromycin resistance
Detecção de mutações no gene 23S do Helicobacter pylori envolvidas na resistência à claritromicina
Nathalie Campos Murillo1![]() ,Paola Orellana Bravo1
,Paola Orellana Bravo1![]() , Patricia Noguera Cárdenas2
, Patricia Noguera Cárdenas2![]() , Carlos Andrade Tacuri1
, Carlos Andrade Tacuri1![]()
NC:Docente Investigador desde octubre 2009 hasta la presente fecha. Subdecana de la Unidad Académica de Ciencias Agropecuarias desde diciembre 2016 hasta noviembre 2018. Coordinadora del Laboratorio de Biotecnología del Centro de Investigación, Innovación y Transferencia de Tecnología mayo de 2018 hasta septiembre de 2019, en la Universidad Católica de Cuenca. Ecuador. ncampos@ucacue.edu.ec
PO: Médica General por la Universidad Nacional de Chimborazo (2011). Especialista en Medicina de Emergencias y Desastres por la Universidad Central del Ecuador (2018). Estudiante de la maestría en Gerencia de Instituciones de salud en la Universidad Técnica Particular de Loja. Médico tratante del servicio de Emergencia del Hospital General Riobamba IESS. Instituto Ecuatoriano de Seguridad Social – Hospital General Riobamba. Ecuador. porellana@ucacue.edu.ec
PN: Química Farmacéutica en Clínica Santa Fe, desde julio 2012 hasta la presente fecha. Laboratorista en Centro de Salud B-Sucua desde marzo 2017 hasta la actualidad. Docente de la Universidad Católica de Cuenca de octubre 2018 hasta marzo 2019. Especialista en Atención Primaria de Salud. Centro De Salud B SUCUA IESS, Ecuador. patricianoguera.c@hotmail.com
CA: Docente Investigador desde octubre 2011 hasta la presente fecha. Perito en Medicina Humana, Subespecialidad Genética 2016. Coordinador del Laboratorio de Genética y Biología Molecular del Centro de Investigación, Innovación y Transferencia de Tecnología (CIITT) de la Universidad Católica de Cuenca. Ecuador. candradet@ucacue.edu.ec
Recibido 12 de octubre 2020 | Arbitrado y aceptado 09 de noviembre 2020 | Publicado en 22 de diciembre 2020
Resumen
Introducción: Uno de los principales factores que influyen en el tratamiento para la erradicación de Helicobacter pylori es la resistencia a antibióticos, la cual difiere entre países e incluso regiones de un país. Entre los antibióticos más usados para el tratamiento de la infección se encuentra la claritromicina, se ha demostrado que el gen 23S ARNr está involucrado en la resistencia a este antibiótico, como resultado de mutaciones puntuales. Objetivo: Detectar las mutaciones presentes en el gen 23S ARNr que codifican la resistencia a la claritromicina en Helicobacter pylori a través de un método no invasivo y rápido. Materiales y métodos: A partir de muestras de heces de 76 pacientes con síntomas gastrointestinales asociados a la bacteria, se aisló y purificó el ADN bacteriano, se identificó el gen 23S ARNr mediante seminested PCR. Para la detección de mutaciones puntuales en el gen se realizó la RFLP, utilizando las enzimas HhaI que detecta la mutación T2717C y MboII que identifica la mutación A2142C/G. Resultados: Un total de 45 pacientes resultaron positivos a Helicobacter pylori lo cual corresponde al 59,2%. La mutación T2717C analizada con la enzima HhaI se presentó en el 2,2% de la muestra de estudio, no se obtuvo resultados positivos para la enzima MboII. Conclusiones: A través de la Seminested PCR se identificó al gen 23S ARNr de Helicobacter pylori, PCR-RFLP es un método fiable para detectar la presencia de mutaciones causantes de resistencias a antibióticos, útil antes de elegir el tratamiento erradicador contra las infecciones por Helicobacter pylori.
Palabras clave: Helicobacter pylori; claritromicna; PCR-RFLP; mutaciones; Gen 23S ARN
Abstract
Introduction: One of the main factors that influence the treatment for the eradication of Helicobacter pylori is resistance to antibiotics, which differs between countries and even regions of a country. Clarithromycin is among the most widely used antibiotics for the treatment of infection. The 23S rRNA gene has been shown to be involved in resistance to this antibiotic, as a result of point mutations. Objective: To detect the mutations present in the 23S rRNA gene that encode resistance to clarithromycin in Helicobacter pylori through a non-invasive and rapid method. Materials and methods: From stool samples of 76 patients with gastrointestinal symptoms associated with the bacteria, bacterial DNA was isolated and purified, the 23S rRNA gene was identified by seminested PCR. For the detection of point mutations in the gene, RFLP was performed, using the enzymes HhaI that detects the T2717C mutation and MboII that identifies the A2142C / G mutation. Results: A total of 45 patients were positive for Helicobacter pylori, which corresponds to 59.2%. The T2717C mutation analyzed with the HhaI enzyme was present in 2.2% of the study sample, no positive results were obtained for the MboII enzyme. Conclusions: The 23S rRNA gene of Helicobacter pylori was identified through Seminested PCR, PCR-RFLP is a reliable method to detect the presence of mutations causing resistance to antibiotics, useful before choosing the eradication treatment against Helicobacter pylori infections.
Key words: Helicobacter pylori; clarithromycin; PCR-RFLP; mutations; Gen 23S RNA
Resumo
Introdução: Um dos principais fatores que influenciam no tratamento para erradicação do Helicobacter pylori é a resistência aos antibióticos, que difere entre países e até mesmo regiões de um país. A claritromicina está entre os antibióticos mais amplamente utilizados para o tratamento de infecções.O gene 23S rRNA demonstrou estar envolvido na resistência a esse antibiótico, como resultado de mutações pontuais. Objetivo: Detectar as mutações presentes no gene 23S rRNA que codificam resistência à claritromicina no Helicobacter pylori, por meio de um método não invasivo e rápido. Materiais e métodos: A partir de amostras de fezes de 76 pacientes com sintomas gastrointestinais associados à bactéria, o DNA bacteriano foi isolado e purificado, o gene 23S rRNA foi identificado por PCR seminestado. Para a detecção de mutações pontuais no gene, foi realizado RFLP, utilizando as enzimas HhaI que detecta a mutação T2717C e MboII que identifica a mutação A2142C / G. Resultados: Um total de 45 pacientes foram positivos para Helicobacter pylori, o que corresponde a 59,2%. A mutação T2717C analisada com a enzima HhaI estava presente em 2,2% da amostra do estudo, nenhum resultado positivo foi obtido para a enzima MboII. Conclusões: Por meio da PCR seminestada, foi identificado o gene rRNA 23S do Helicobacter pylori, o PCR-RFLP é um método confiável para detectar a presença de mutações que causam resistência a antibióticos, útil antes de escolher o tratamento de erradicação contra infecções por Helicobacter pylori.
Palavras-Chave: Helicobacter pylori; claritromicina; PCR-RFLP; mutações; RNA Gen 23S
INTRODUCCIÓN
Helicobacter pylori (H. Pylori) es una bacteria capaz de colonizar la mucosa gástrica, causa inflamación crónica del revestimiento interno del estómago (gastritis), provoca dentro de la mucosa gastroduodenal cambios estructurales y funcionales, por tanto las personas infectadas desarrollan una inflamación gástrica crónica generalmente asintomática, sin embargo en otras personas se desarrolla úlcera péptica; también es un factor de riesgo frente al adenocarcinoma y cáncer gástrico (1) . Además, se ha demostrado una relación entre la diabetes, infecciones y elevados niveles de hemoglobina (2) , la colonización del estómago por H. pylori es la más común de las infecciones bacterianas crónicas en el ser humano, afectando alrededor del 50% de la población mundial (3) .
Entre los antibióticos más usados se encuentran amoxicilina, tetraciclina, metronidazol y claritromicina, siendo este último el antibiótico más frecuentemente usado, sin embargo, esta bacteria ha presentado resistencia a los antibióticos comúnmente utilizados, provocando así disminución en la rapidez de su erradicación (4) . La resistencia primaria varía entre 10% en los países industrializados y 70% en los países en vías de desarrollo (5) . La mala utilización de estas drogas contribuye sin duda al incremento en la velocidad de resistencia, algunos estudios han caracterizado el gen 23S ARNr el cual se cree que está asociado a la resistencia a la claritromicina en H. Pylori (6) .
La claritromicina al unirse directamente al ARN ribosomal (ARNr) de la subunidad mayor del ribosoma actúa inhibiendo la síntesis de proteínas, algunos análisis genéticos han demostrado que debido a la aparición de diversas mutaciones puntuales en el gen del 23S ARNr se desarrolla la resistencia (7) . Dichas mutaciones en el gen 23S ARNr presentan una disminución de la capacidad de unión del antibiótico al ribosoma, en la posición 2142 del gen se produce una mutación puntual que reemplaza una base de guanina por una base de adenina (A2142G) creando un sitio de restricción que va a ser reconocido por la endonucleasa MboII, produciéndose así mediante dicha enzima dos fragmentos de 700 pares de bases. Algunos estudios realizados en mayor detalle han señalado que las cepas que portan la mutación A2142G son las que presentan mayor resistencia a Claritromicina (5, 8) .
A más del cambio de una adenina por guanina en las posiciones 2142 y 2143, han reportado una sustitución de adenina a citosina en la posición 2142 y una transición de citosina a timina en la posición 2717 creando un sitio de restricción reconocido por la endonucleasa HhaI (9) . Durante los últimos años varias técnicas desarrolladas permiten detectar el ADN de H. pylori directamente de biopsias gástricas; sin embargo, también se puede detectar en muestras como heces, saliva o agua. La gran parte de estas técnicas se fundamentan en la Reacción en Cadena de la Polimerasa (PCR) convencional o clásica, además existen variaciones como la PCR-RFLP, nested o semi-nested PCR, en Tiempo Real, entre otras; cuyo objetivo es la detección de genes específicos en la bacteria y a su vez de los mecanismos de resistencia principalmente a la claritromicina en donde se amplifica un fragmento de 1,4 Kpb que corresponde al dominio V de la región 23S ARNr para detectar las mutaciones mediante la digestión con las enzimas MboII y HhaI (10,11).
En un consultorio privado en la ciudad de Cuenca donde se realizó el estudio, presenta afluencia de alrededor de 200 pacientes mensuales de los cuales 20 a 30 de ellos presentan H. pylori, se observa un incremento significativo del número de pacientes con enfermedades Gastroduodenales desencadenadas por la bacteria, sin que exista información detallada del por qué esta bacteria no ha sido erradicada en los pacientes que acuden a la institución antes mencionada o si es que dichos pacientes no han recibido ningún tipo de tratamiento, en tal virtud, es necesario plantear una propuesta de investigación que nos permita detectar la mutación causante de la resistencia a la claritromicina, antibiótico usado en la erradicación del H. pylori mediante una técnica no invasiva para el paciente que permita la rápida detección de las mutaciones.
Con la información generada en el presente estudio se aportará con un método de detección de H. pylori y de mutaciones en el gen 23S ARNr causantes de la resistencia a los antibióticos de erradicación de la bacteria, este estudio a su vez sugiere que esta prueba puede ser muy útil para el médico antes de escoger el tratamiento erradicador para los pacientes que padecen de enfermedades asociadas a H. pylori, además de tratarse de un método no invasivo, puesto que se va a realizar los análisis en muestras de heces fecales.
MATERIALES Y MÉTODOS
Se recogió un total de 76 muestras de heces de pacientes adultos con un rango de edad entre 22 a 52 años, de los cuales 36 fueron hombres y 40 mujeres, quienes presentaban síntomas gastrointestinales.
La extracción de ADN bacterial en las muestras frescas de heces fecales se realizó mediante el Kit QIAamp Fast DNA Stool, este kit está diseñado para la rápida y total purificación de ADN de 180mg hasta 220mg de heces ya sean muestras frescas o congeladas, este procedimiento se realizó acorde a las instrucciones del fabricante.
Se detectó la presencia de H. pylori a través del método de amplificación seminested PCR del gen 23S ARNr, las mezclas para las reacciones, el protocolo de amplificación y las condiciones para la electroforesis se detallan a continuación, así como las variantes realizadas:
La primera reacción se llevó a cabo con un set primers o cebadores con secuencia de nucleótidos conocida del gen 23S ARNr de H. pylori, Primer 1: HP1 forward (5-CCACAGCGATGTGGTCTCAG-3; con número de accesión U2720 de 1820 a 1840), y Primer 2: HP2 reverse (5-TGTGTAGCTACCCAGCGATGCTC-3 con número de accesión U2720 de 2811 a 2790). La mezcla de la reacción de amplificación acorde a las instrucciones del fabricante: GoTaq Hot Start Green Master Mix de Promega, el volumen de cada reacción de 25µl: GoTaq Hot Start Green Master Mix 2X, upstream primer 10µM, downstream primer 10µM, eluido de ADN purificado de las muestras de heces 2µl y Nuclease-Free Water. La reacción se llevó a cabo en el Termociclador Piko de Finnzymes instruments el mismo que permite un volumen de reacción final entre 5-20µl, por lo tanto se ajustó la mezcla de la reacción de amplificación.
La segunda reacción se desarrolló bajo la misma mezcla y volumen final de reacción así como las condiciones de PCR descritas anteriormente, se empleó 1µl, 1.5µl, 2µl y 2.5µl del producto obtenido de la primera reacción de PCR y un segundo juego de primers o cebadores, Primer 1: HP4 forward (5-GTCGGTTAAATACCGACCTG-3; con número de accesión U27270 de 2028 a 2048), y Primer 2: HP2 reverse (5-TGTGTAGCTACCCAGCGATGCTC-3) utilizado en la primera reacción.
El protocolo de amplificación de PCR que se llevó a cabo en el termociclador Piko, está conformado por una desnaturalización inicial de 2 minutos a 95°C, a continuación 30 ciclos cada uno de los cuales consistía en: 45 segundos a 95°C para la desnaturalización, 45 segundos a 65°C para la hibridación y 45 segundos a 72°C para la extensión, y una elongación final a 72 ° C durante 4 minutos.
Los productos de la seminested PCR fueron analizados por electroforesis, para lo cual se preparó un gel de agarosa al 1.5%, utilizando 2.25gr de agarosa en 150ml de TAE Buffer 1X y 7,5µl de Bromuro de Etidio como agente intercalante. Una vez listo el gel de agarosa con las especificaciones anteriores, se procedió a preparar las muestras con el tampón de carga Blue Juice, tomando 8µl del tampón con 10µl del producto de cada reacción de PCR.
El análisis de polimorfismos de longitud de fragmentos de restricción (RFLP), se llevó a cabo con dos enzimas: HhaI que detecta la mutación T2717C y MboII que detecta la mutación A2142C/G; la mezcla de la reacción se llevó a cabo con los amplicones obtenidos de la seminested PCR, debido a que son productos relativamente pequeños comparados con el ADN total además de la pureza del producto.
Digestión con la enzima de restricción HhaI y Digestión con la enzima de restricción MboII, Se elaboró una nueva mezcla de reacción para cada enzima, compuesta de la siguiente manera: agua estéril desionizada 14µl, RE 10X Buffer 5µl, Acetylated BSA 0.5µl, producto de PCR 10µl; mezclamos por pipeteo y añadimos la enzima de restricción HhaI 0.5 µl, ó, MboII 0.5µl, obteniendo un volumen final de 40µl. Se mezcló por pipeteo y centrifugamos durante 20 segundos, incubamos a 37°C durante 1 hora. Acabado el tiempo de incubación se adicionó 8µl de loading buffer (Blue Juice) y depositamos en el gel de agarosa para el análisis.
RESULTADOS
La primera reacción de PCR se llevó a cabo a partir de ADN genómico de Helicobacter pylori extraído a partir de muestras de heces, se utilizó los primers HP1 y HP2 para la amplificación del gen 23S ARNr. En la Figura 1 se utilizó el mismo ladder o marcador de peso molecular en la escala de 1000bp; y, se obtuvo un primer amplicón de 993bp, acorde a la revisión bibliográfica. De igual manera todas las imágenes presentan: Ladder; C-: Control negativo (Cepa ATCC 43300 de Staphylococcus aureus); C+: Control positivo (Cepa ATCC 26695 de Helicobacter pylori).
TABLAS Y FIGURAS
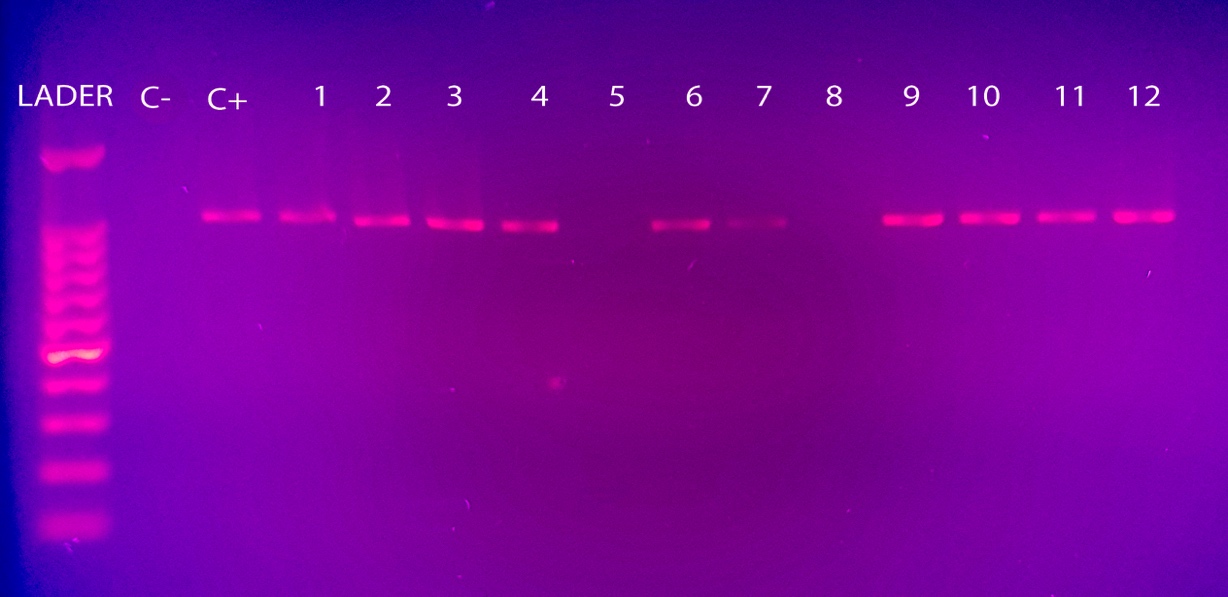
Figura 1. Electroforesis en gel de agarosa al 1,5% de la amplificación por PCR del gen 23S ARNr.
La segunda reacción de PCR (seminested) se llevó a cabo a partir del producto de la primera reacción, se utilizó los primers HP4 y HP2 para la amplificación del gen 23S ARNr. En la Figura 2, se utilizó el mismo ladder o marcador de peso molecular en la escala de 1000bp; y, se obtuvo un segundo amplicón de 783bp, acorde a la revisión bibliográfica. De igual manera todas las imágenes presentan: Ladder; C-: Control negativo (Cepa ATCC 43300 de Staphylococcus aureus); C+: Control positivo (Cepa ATCC 26695 de Helicobacter pylori).
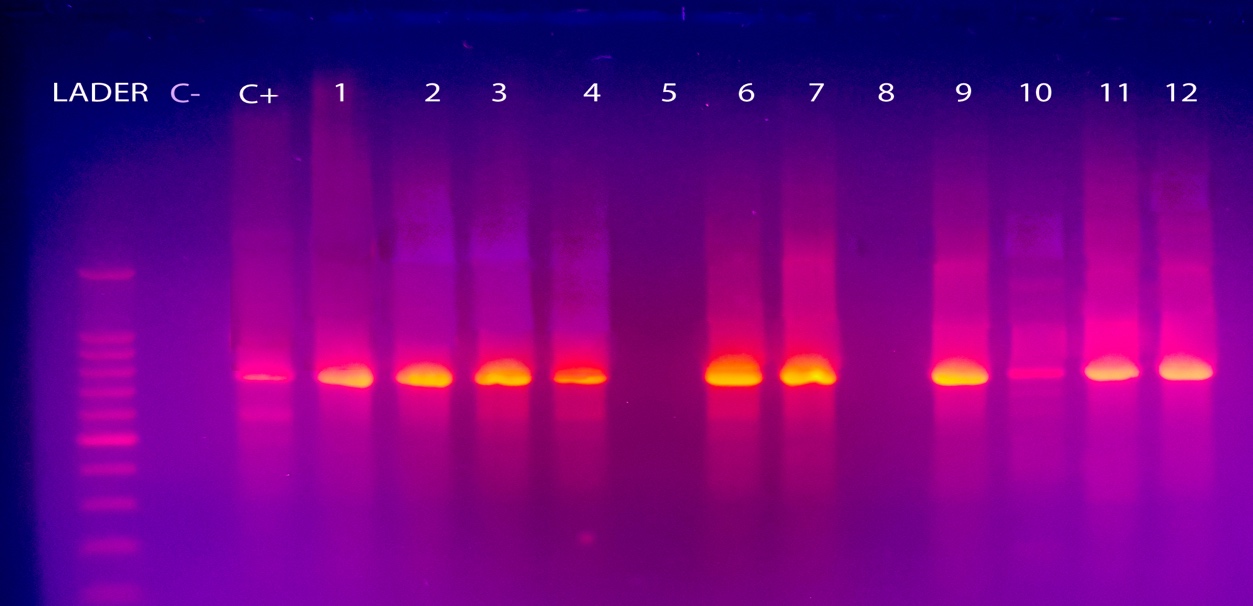
Figura 2. Electroforesis en gel de agarosa al 1,5% de la amplificación por Seminested PCR del gen 23S ARNr.
El análisis de polimorfismos de longitud de fragmentos de restricción (RFLP), se llevó a cabo con el producto de la seminested PCR, se utilizó la enzima HhaI. En la Figura 3 se presenta: Ladder; C-: Control negativo (Cepa ATCC 43300 de Staphylococcus aureus), se realizó repeticiones de la muestra 40 que resulto positiva para el análisis de restricción, observándose los diferentes tamaños generados debido a los cortes que genera la enzima HhaI. De las 45 muestras analizadas solamente una es resistente a la Claritromicina, presentando la mutación T2717C.
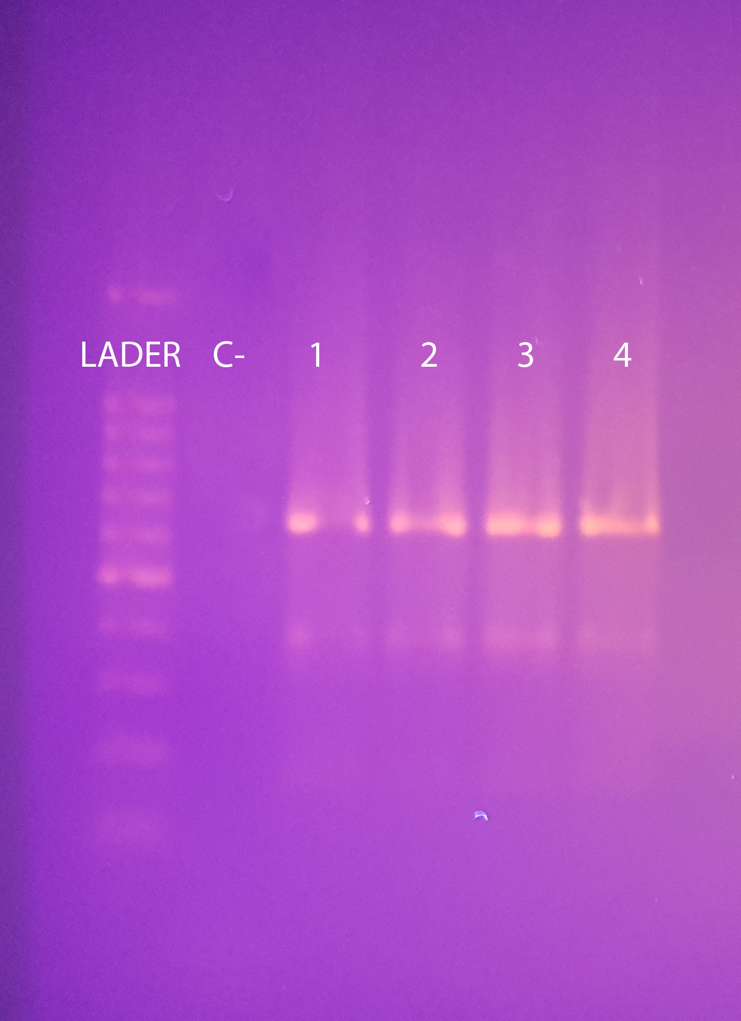
Figura 3. Electroforesis en gel de agarosa al 1,5% del RFLP con la enzima de restricción HhaI.
DISCUSIÓN
La prevalencia de Helicobacter pylori en la población del presente estudio fue del 59,2% misma que se presenta acorde a la prevalencia mundial determinada por la OMS la cual es mayor al 50%, cuya frecuencia aumenta en países en vías de desarrollo; sin embargo, en cuanto al género no se observó un gran rango de diferencia ya que en varones se presentó en un 44,44% y en mujeres en un 55,6%.
Según Malaty (12) , las manifestaciones clínicas aparecen en la edad adulta en la mayoría de los casos, acorde a su estudio la frecuencia más alta de H. pylori reportada en la presente investigación fue en una media de 35,88 años de edad.
La prueba de cassette Certest para la detección de antígenos en heces brinda además de una sensibilidad mayor al 94% una especificidad mayor al 99%, de acuerdo a los estudios realizados por la casa comercial BIOTEC (13) , ya que entre los estudios realizados no se detectó reacciones cruzadas con patógenos gastrointestinales ocasionalmente presentados en heces, lo cual aporta una gran ventaja para su aplicación en este tipo de estudios donde se propone técnicas no invasivas para el diagnóstico de H. pylori y posteriores aplicaciones a nivel molecular.
Actualmente existen diversas técnicas de PCR para la detección de H. pylori a partir de heces fecales, la seminested PCR reportada por Fontana et al. (14) mostró especificidad para H. pylori ya que la cepa bacteriana utilizada como control negativo no fue amplificada para el gen 23S ARNr. La amplificación de este gen directamente de heces fecales por seminested PCR se correlacionó en su totalidad con los resultados positivos del test Certest.
De acuerdo al Informe del Consenso de Florencia Maastricht-III de marzo del 2005, las recomendaciones de opciones de tratamiento de la infección por H. pylori se basan en una terapia de primera línea sea esta triple o cuádruple conformada por amoxicilina, claritromicina y metronidazol además de IBP o bismuto, para escoger la terapia adecuada se debe basar en la resistencia antimicrobiana la misma que es la principal razón del fracaso del tratamiento.
Según la Organización Mundial de Gastroenterología, la resistencia mundial a los antibióticos de del 2 - 25% para la claritromicina, del 50 - 80% para metronidazol y del 0 - 1% para la amoxicilina; si bien la resistencia a metronidazol es alta es menos relevante a nivel clínico; por tanto la terapia podría fracasar debido a la resistencia a la claritromicina. Si bien las tasas de resistencia son elevadas, las mismas varían de acuerdo a la población de estudio; pese a estos altos índices de resistencia microbiana no siempre se puede detectar antes de administrar la terapia antibiótica debido a los requerimientos especiales para cultivar la bacteria (4) .
Por tal motivo se han desarrollado nuevas técnicas como la PCR-RFLP descrita por Agudo et al. (10) y Fontana et al. (14) , posterior a la seminested PCR para amplificar el gen 23S ARNr, se sometió al análisis de longitud de los fragmentos de restricción generados por las enzimas HhaI y MboII, obteniendose únicamente el 1% de mutación para la enzima HhaI con lo cual determinamos la presencia de la mutación T2717C la cual genera resistencia a la claritromicina.
Estos resultados concuerdan con los de Fontana et al. (14) en una población italiana, en donde reportan únicamente 2 muestras positivas de H. pylori resistente a la claritromicina de 125 muestras de estudio; sin embargo, difiere de los estudios de Agudo et al. (10) en una población española, donde obtuvo 34 cepas resistentes a la claritromicina de 42 cepas de estudio; en otro estudio realizado por Álvarez et al. (15) en una población colombiana, reporta resultados similares, de un total de 88 cepas de estudio, el 2,2% presentó resistencia a la claritromicina.
Acorde a los estudios analizados sugieren que la resistencia a la claritromicina varía de una población a otra, por tanto es necesario conocer las características de las cepas que se encuentran en la población, así como los mecanismos de resistencia lo cual apunte a mantener antecedentes recopilados para una vigilancia permanente en los tratamientos de erradicación de H. pylori.
CONCLUSIONES
La bacteria Helicobacter pylori es un patógeno de gran importancia para la salud ya que infecta aproximadamente al 50% de la población mundial, las condiciones microbiológicas para su cultivo son exigentes y demandan gran cantidad de tiempo, y los métodos de diagnóstico más usados son de carácter invasivo por la fiabilidad y rapidez de los resultados.
El presente estudio se centró en la detección de las mutaciones del gen 23S ARNr que codifican la resistencia a la claritromicina a través de un método no invasivo y rápido. Se obtuvieron resultados interesantes, cuyos principales hallazgos de este estudio son:
Se identificó la presencia de Helicobacter pylori en muestras de heces de pacientes infectados a través de primers específicos mediante la primera reacción de PCR, se utilizó cepas control positiva y negativa para valorar la especificidad y sensibilidad de la técnica.
A través de Seminested PCR se identificó al gen 23S ARNr de Helicobacter pylori utilizando un nuevo primer para la segunda reacción, el uso de esta técnica de PCR puede reducir el uso de la endoscopia gastroduodenal la cual no es admitida por algunos pacientes como los pediátricos.
Se analizó la presencia de mutaciones en el gen 23S ARNr de Helicobacter pylori mediante RFLP con las enzimas de restricción: MboII que reconoce la mutación A2142C/G y la HhaI para la mutación T2717C; en este estudio se encontró la mutación T2717C en el 1% de la muestra de estudio. Este método permite direccionar la terapia antibiótica para la erradicación de la bacteria.
Es indispensable el conocimiento de las características de las cepas de H. pylori que aquejan a nuestra población, con el objetivo de recopilar información que facilite desde el punto de vista clínico y epidemiológico conseguir un buen resultado en las terapias de erradicación, y a su vez permita la posibilidad de la detección de forma rápida de la resistencia antibiótica.
- Conflicto de intereses: los autores declaramos no presentar conflicto de intereses.
- Financiación: autofinanciamiento.
- Agradecimiento: agradecemos a la Universidad Católica de Cuenca por permitir el uso de las instalaciones así como el apoyo para la realización del presente trabajo.
- Investigación realizada considerando los tratados bioéticos: esta investigación fue realizada bajo consentimiento informado, apegados a los tratados bioéticos, no se realizó ningún método invasivo para la toma de muestras.
REFERENCIAS BIBLIOGRÁFICAS
1. Murray PR. Microbiología médica básica. 2018. [ Links ]
2. Pareja Cruz A, Navarrete Mejía PJ, Parodi García JF. Seroprevalencia de infección por Helicobacter pylori en población adulta de Lima, Perú 2017. Horiz Médico 2017;17:55–8. https://doi.org/10.24265/horizmed.2017.v17n2.09 [ Links ]
3. Duquense A, Rodríguez Y, Orellana A. Caracterización clínico-epidemiológica-endoscópica-anatomopatológica y microbiológica de pacientes con gastritis. Policlínico 19 de Abril. 2012-2016. Panor Cuba y Salud 2014;9:42–7. [ Links ]
4. Harris PR, Calderón-Guerrero OG, Vera-Chamorro JF, Lucero Y. Guidelines on the Diagnosis, Prevention and Treatment of Helicobacter pylori. Rev Chil Pediatría 2020;91:1–19. https://doi.org/10.32641/rchped.vi91i5.2579 [ Links ]
5. Vallejos MC, Cerda AO, Valenzuela VM, Toledo AH. Resistencia antimicrobiana en Helicobacter pylori: aspectos clínicos y moleculares TT - Antimicrobial resistance of Helicobacter pylori: Clinical and molecular aspects. Rev Med Chil 2003;131:1313–20. https://doi.org/10.4067/S0034-98872003001100014 [ Links ]
6. Kwon DH, Hulten K, Kato M, Kim JJ, Lee M, El-Zaatari FAK, et al. DNA sequence analysis of rdxA and frxA from 12 pairs of metronidazole-sensitive and -resistant clinical Helicobacter pylori isolates. Antimicrob Agents Chemother 2001;45:2609–15. https://doi.org/10.1128/AAC.45.9.2609-2615.2001 [ Links ]
7. Sakinc T, Baars B, Wüppenhorst N, Kist M, Huebner J, Opferkuch W. Influence of a 23S ribosomal RNA mutation in Helicobacter pylori strains on the in vitro synergistic effect of clarithromycin and amoxicillin. BMC Res Notes 2012;5:2–5. https://doi.org/10.1186/1756-0500-5-603 [ Links ]
8. Bluemel B, Goelz H, Goldmann B, Grüger J, Hamel H, Loley K, et al. Antimicrobial resistance of Helicobacter pylori in Germany, 2015 to 2018. Clin Microbiol Infect 2020;26:235–9. https://doi.org/10.1016/j.cmi.2019.06.007 [ Links ]
9. Acosta CP, Hurtado FA, Trespalacios AA. Determinación de mutaciones de un solo nucleótido enel gen 23S rRNA de Helicobacter pylori relacionadas conresistencia a claritromicina en una población del departamento del Cauca, Colombia. Biomedica 2014;34:156–62. https://doi.org/10.7705/biomedica.v34i0.1649 [ Links ]
10. Agudo S, Pérez-Pérez G, Alarcón T, López-Brea M. Rapid detection of clarithromycin resistant Helicobacter pylori strains in Spanish patients by polymerase chain reaction-restriction fragment length polymorphism. Rev Esp Quimioter 2011;24:32–6 [ Links ]
11. Xuan SH, Wu LP, Zhou YG, Xiao MB. Detection of clarithromycin-resistant Helicobacter pylori in clinical specimens by molecular methods: A review. J Glob Antimicrob Resist 2016;4:35–41. https://doi.org/10.1016/j.jgar.2016.01.002 [ Links ]
12. Malaty HM. Epidemiology of Helicobacter pylori infection. Best Pract Res Clin Gastroenterol 2007;21:205–14. https://doi.org/10.1016/j.bpg.2006.10.005. [ Links ]
13. Certest Biotec S.L. CERTEST H. pylori. Certest Biotec SL. 2008:1–12 [ Links ]
14. Fontana C, Favaro M, Pietroiusti A, Pistoia ES, Galante A, Favalli C. Detection of clarithromycin-resistant Helicobacter pylori in stool samples. J Clin Microbiol 2003;41:3636–40. https://doi.org/10.1128/JCM.41.8.3636-3640.2003 [ Links ]
15. Álvarez A, Moncayo JI, Santacruz JJ, Corredor LF, Reinosa E, Martínez JW, et al. Resistencia a metronidazol y claritromicina en aislamientos de. Rev Med Chil 2009;137:1309–14 [ Links ]














