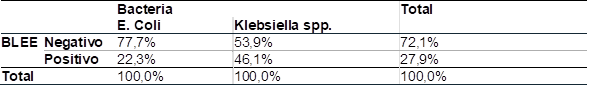ARTICULO DE INVESTIGACIÓN
Frecuencia de E.coli y Klebsiella spp productoras de betalactamasas en cultivos procesados en un laboratorio clínico
Frequency of E. coli and betalactamase-producing Klebsiella spp in cultures processed in a clinical laboratory
Frequência de E. coli e Klebsiella spp produtoras de betalactamase em culturas processadas em laboratório clínico
1Universidad Católica de Cuenca. Cuenca, Ecuador
RESUMEN
Introducción: la resistencia antibiótica en bacterias patógenas como Escherichia coli y Klebsiella spp. productoras de betalactamasas, han surgido como un problema global de salud pública. Su presencia, se asocia con infecciones intrahospitalarias y comunitarias, aumentando la morbilidad y la mortalidad de los pacientes. Objetivo: determinar la frecuencia de E.coli y Klebsiella spp productoras de betalactamasas en cultivos procesados en un laboratorio clínico.
Métodos: se realizó un estudio descriptivo de diseño documental. La muestra estuvo constituida por un total de 1465 resultados de cultivos positivos para Escherichia coli o Klebsiella spp. en el periodo 2022. Para la recolección de la información, se tuvo acceso a la base de datos anonimizada del laboratorio en una hoja de Excel para su posterior análisis. Los datos fueron tabulados en SPSS versión 25. Resultados: el análisis de bacterias productoras de BLEE mostró una positividad del 22,3% en E. coli y 46,1% en Klebsiella spp. E. coli presentó mayor frecuencia de negativos (77,7%) en comparación con Klebsiella spp. La presencia de E. coli fue más común en muestras de orina (90,6%) y en otras muestras como esputo y heridas cutáneas (21,3%). Se evaluaron 8 antibióticos, y se destacó la alta sensibilidad para amikacina (AK) (99,6% y 98,0%) y elevada resistencia ampicilina (AM) (91,5% y 100%) en ambas especies. Ciprofloxacino (CIP) y Trimetropin/Sulfametoxazol (STX) mostraron relativa frecuencia mayor de resistencia. Conclusión: los resultados muestran una alta frecuencia de bacterias productoras de BLEE en E. coli y Klebsiella spp., con una mayor prevalencia en Klebsiella spp. Además, la resistencia a AM, CIP y STX destaca la importancia de una gestión adecuada de la resistencia antimicrobiana.
Palabras clave: Escherichia coli; Enteropatógena; Klebsiella; Farmacorresistencia Microbiana
ABSTRACT
Introduction: antibiotic resistance in pathogenic bacteria such as Escherichia coli and Klebsiella spp. producing beta-lactamases has emerged as a global public health problem. Their presence has been associated with both hospital-acquired and community-acquired infections, leading to increased morbidity and mortality in patients. Objective: to determine the frequency of betalactamase-producing E. coli and Klebsiella spp. in cultures processed in a clinical laboratory. Methods: a descriptive documentary design study was conducted. The sample consisted of a total of 1465 positive culture results for Escherichia coli or Klebsiella spp. in the year 2022. Data collection involved accessing the laboratory's anonymized database in an Excel sheet for subsequent analysis. The data were tabulated in SPSS version 25. Results: the analysis of ESBL-producing bacteria showed a positivity of 22.3% in E. coli and 46.1% in Klebsiella spp. E. coli showed a higher frequency of negatives (77.7%) compared to Klebsiella spp. The presence of E. coli was more common in urine samples (90.6%) and in other samples such as sputum and skin wounds (21.3%). Eight antibiotics were evaluated, with high sensitivity noted for amikacin (AK) (99.6% and 98.0%) and high resistance for ampicillin (AM) (91.5% and 100%) in both species. Ciprofloxacin (CIP) and Trimethoprim/Sulfamethoxazole (STX) showed a relatively higher frequency of resistance. Conclusion: the results show a high frequency of ESBL-producing bacteria in E. coli and Klebsiella spp., with a higher prevalence in Klebsiella spp. Furthermore, the resistance to AM, CIP, and STX highlights the importance of proper management of antimicrobial resistance.
Key words: Enteropathogenic; Escherichia coli; Klebsiella; Drug Resistance
RESUMO
Introdução: a resistência antibiótica em bactérias patogênicas como Escherichia coli e Klebsiella spp., produtoras de beta-lactamases, emergiu como um problema de saúde pública global. Sua presença tem sido associada a infecções hospitalares e comunitárias, aumentando a morbidade e a mortalidade dos pacientes. Objetivo: determinar a frequência de E. coli e Klebsiella spp. produtoras de betalactamase em culturas processadas em laboratório clínico.
Métodos: foi realizado um estudo descritivo de design documental. A amostra consistiu em um total de 1465 resultados de cultura positiva para Escherichia coli ou Klebsiella spp. no ano de 2022. A coleta de dados envolveu o acesso ao banco de dados anonimizado do laboratório em uma planilha do Excel para análise subsequente. Os dados foram tabulados na versão 25 do SPSS. Resultados: a análise de bactérias produtoras de BLEE mostrou uma positividade de 22,3% em E. coli e 46,1% em Klebsiella spp. E. coli apresentou uma frequência maior de resultados negativos (77,7%) em comparação com Klebsiella spp. A presença de E. coli foi mais comum em amostras de urina (90,6%) e em outras amostras, como escarro e feridas na pele (21,3%). Foram avaliados oito antibióticos, com alta sensibilidade observada para amicacina (AK) (99,6% e 98,0%) e alta resistência para ampicilina (AM) (91,5% e 100%) em ambas as espécies. Ciprofloxacina (CIP) e Trimetoprima/Sulfametoxazol (STX) mostraram uma frequência relativamente maior de resistência. Conclusão: os resultados mostram uma alta frequência de bactérias produtoras de BLEE em E. coli e Klebsiella spp., com uma maior prevalência em Klebsiella spp. Além disso, a resistência a AM, CIP e STX destaca a importância da adequada gestão da resistência antimicrobiana.
Palavras-Chave: Escherichia coli; Enteropatogênica; Klebsiella; Resistência Microbiana a Medicamentos
INTRODUCCIÓN
La efectividad de los medicamentos antimicrobianos es crucial para la medicina contemporánea. No obstante, se ha observado un preocupante incremento en las tasas de infecciones resistentes en diversos microorganismos en todas las regiones a nivel mundial (1). Un estudio realizado por Murray et al., estimó que hubo aproximadamente 4.95 millones de muertes asociadas a la resistencia antimicrobiana en el año 2019, y entre los principales patógenos responsables a estas muertes se encontraron las bacterias Escherichia coli (E. coli) y Klebsiella spp) (2).
Estas bacterias gram negativas son reconocidas por su capacidad para producir betalactamasas, enzimas que desactivan los antibióticos betalactámicos. Estos microorganismos son responsables de infecciones comunes, como las del tracto urinario, respiratorias, hematológicas e intraabdominales, siendo de suma importancia entender la magnitud del problema de resistencia bacteriana (3,4).
Por lo tanto, se han llevado a cabo varios estudios para comprender mejor la prevalencia y resistencia de estas bacterias; un estudio realizado en Egipto reveló que el 60% de las cepas aisladas de E. coli y Klebsiella spp mostraron ser productoras de carbapenemasas, y aproximadamente el 93,8% de ellas presentaron una alta resistencia a los antibióticos β-lactámicos. Asimismo, un estudio en Tailandia encontró que del total de 362 aislados de estos microorganismos, un 87,3% poseía algún gen productor de betalactamasas. En contraste, una investigación llevada a cabo en Hong Kong encontró solamente un 9,8% de cepas con resistencia a β-lactámicos (5-7). El tratamiento de las infecciones causadas por E. coli y Klebsiella spp representa un desafío terapéutico para los médicos debido a los cambios en la incidencia y prevalencia de estas bacterias, los cuales van de la mano con su resistencia a diversos tipos de antibióticos.
Además, estudios realizados en España durante los años 2011-2015, investigaron la sensibilidad antimicrobiana y la presencia de β-lactamasas en bacilos gramnegativos. Los resultados revelaron que E. coli y Klebsiella spp eran los microorganismos más prevalentes con genes de resistencia a los betalactámicos (8). Por otro lado, una revisión sistemática que incluyó 49 estudios realizados en Irán encontró que el 24% de las cepas de E. coli y el 5% de las cepas de Klebsiella spp presentaron resistencia a los antibióticos (9). Estos hallazgos resaltan la importancia de comprender la resistencia bacteriana a nivel global, permitiendo desarrollar estrategias de tratamiento, implementar medidas de control de infecciones y desarrollar políticas de uso racional de antibióticos, ya que actualmente es un problema significativo de salud pública.
Es por eso, que nos surgió el interés de conocer la frecuencia de E.coli y Klebsiella spp productoras de betalactamasas en cultivos procesados en el laboratorio clínico Solidario de la ciudad de Machala en el año 2022, pudiendo estos hallazgos permitir comprender y abordar el problema de la resistencia antimicrobiana en nuestro entorno como a su vez, proporcionar una base inicial para tomar medidas efectivas en la prevención y tratamiento de las infecciones bacterianas.
Además, la presente investigación también se enfocó en analizar en qué tipo de muestras se encuentran estas bacterias productoras de betalactamasas como el nivel de resistencia bacteriana de las mismas.
MATERIALES Y MÉTODOS
Se realizó un estudio descriptivo de diseño documental. En este estudio, se recogieron de la base de datos bioinformática del laboratorio clínico Solidario del área de Microbiología durante el año 2022, los resultados de las muestras en las que hayan sido aisladas cepas de E. coli y Klebsiella spp.
Posteriormente, se extrajeron los datos sobre la resistencia a los diferentes antibióticos (sensible, intermedio y resistente), el tipo de muestra y se los relacionó con los datos de E. coli y Klebsiella spp productoras o no de betalactamasas. Para analizar la información, se utilizó estadística descriptiva en base a porcentajes utilizando tablas cruzadas tabuladas en el programa SPSS versión 25.
Esta investigación fue fundamentada según los principios éticos establecidos en la Declaración de Helsiki Adedum en Taiwán, se proporcionó la protección y privacidad a los datos de los pacientes, de los cuales se obtuvo la información sobre las muestras de estudio; los datos fueron manejados de manera confidencial y no se usaron con otros fines más que solo el investigativo.
RESULTADOS
Al analizar la frecuencia de bacterias productoras de BLEE, se encontró que un 22,3% de E. Coli y un 46,1% en Klebsiella spp. eran positivas. La diferencia entre las dos bacterias, se evidenció en la frecuencia de negativos encontrándose mayormente en E. Coli (77,7%) en relación a Klebsiella spp (53,9%) (Tabla 1).
Tabla 1 Frecuencia de E.Coli y Klebsiella spp. productoras de betalactamasas
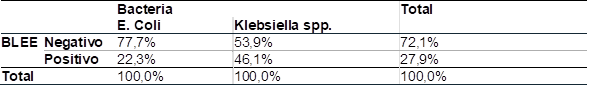
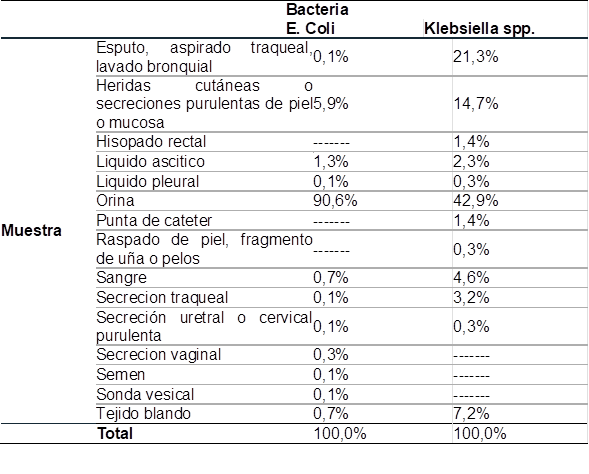
Según el tipo de muestra, la presencia de E. Coli se vio en mayor frecuencia en orina con un 90,6% y un 42,9% en el caso de Klebsiella spp. Se encontró un porcentaje considerable en esputo, aspirado traqueal, lavo bronquial y en heridas cutáneas o secreciones purulentas de piel o mucosa del 21,3% y 14,7% respectivamente en relación a Klebsiella spp. En el resto de muestras hubo un porcentaje bajo de presencia (Tabla 2).
Tabla 2 Frecuencia de E.Coli y Klebsiella spp. productoras de BLEE según tipo de muestra
Se seleccionaron ocho antibióticos que son utilizados durante la presencia y persistencia de E. Coli y Klebsiella spp. Al analizar los datos, se observó una elevada sensibilidad para amikacina en las dos especies bacterianas 99,6% y 98,0% mientras que para ampicilina hubo una elevada resistencia de E. Coli y Klebsiella spp con un 91,5% y 100% respectivamente. En relación a ciprofloxacino y trimetropin/sulfametoxal hubo una relativa frecuencia mayor de resistencia en comparación con los antibióticos amoxi-ácido clavulánico, gentamicina, nitrofurantoina y fosfomicina (Tabla 3).
Tabla 3 Perfiles de resistencia en E.Coli y Klebsiella spp. productoras de betalactamasas
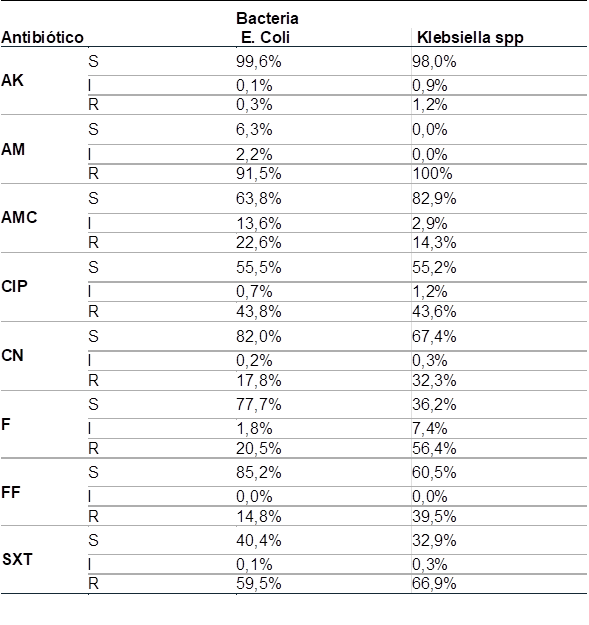
S: sensible, I: intermedio, R: resistente, AK: amikacina, AM: ampicilina, AMC: amoxi-ácido clavulánico, CIP: ciprofloxacino, CN: gentamicina, F: nitrofurantoina, FF: fosfomicina, STX: trimetropin/sulfametoxal.
DISCUSIÓN
La creciente frecuencia de E. coli y Klebsiella spp. productoras de betalactamasas representa un desafío significativo para la salud. Estas bacterias, al producir enzimas capaces de desactivar estos antibióticos, desarrollan resistencia a los mismos incluyendo penicilinas, cefalosporinas y carbapenémicos, que son esenciales para el tratamiento de infecciones bacterianas (10,11).
Datos epidemiológicos muestran un aumento constante en la prevalencia de infecciones causadas por estas especies bacterianas resistentes a los antibióticos en todo el mundo y estas cepas resistentes, son a menudo aisladas en muestras clínicas como orina en casos de infecciones del tracto urinario, sangre en bacteriemias, así como respiratorias en neumonías nosocomiales y comunitarias, datos que concuerdan con este estudio en el cual, se presentó una mayor presencia de estas bacterias en el tracto urinario, sangre y en muestras respiratorias (12,13). Además, estudios han revelado que la prevalencia de Enterobacterias productoras de BLEE ha aumentado en un 5-10% anual en algunos países, lo que destaca su impacto en una amplia variedad de infecciones clínicas (14).
Esta investigación encontró resistencia a varios tipos de antibióticos en E. coli y Klebsiella spp, siendo similar a datos presentados por La Organización Mundial de la Salud (OMS), que ha identificado la resistencia a las cefalosporinas de tercera generación, carbapenémicos y fluoroquinolonas como preocupaciones especialmente graves debido a su papel crucial en el tratamiento de infecciones bacterianas graves (15). Otro estudio realizado en Uganda, encontraron que, en 209 muestras de heces, 189 presentaron crecimiento de E. Coli y Klebsiella spp con patrones de resistencia en contra de ampicilina en un 67%, amoxicilina/ácido clavulánico 37% y ciprofloxacina 31% (16); a nivel local, una investigación realizada en Azogues, determinó que los microorganismos más frecuentes aislados de urocultivos fueron E. Coli seguido de Klebsiella spp con elevada resistencia para ciprofloxacino y trimetropin/sulfametoxazol (17), que coinciden con el patrón identificado en esta investigación; además, es a través de estos estudios que se corrobora que nuestro medio enfrenta la misma problemática que se observa a nivel global.
Finalmente, la adopción de estrategias para prevenir y controlar la propagación de cepas resistentes es esencial para preservar la efectividad de los antibióticos existentes. Esto incluye la implementación de programas de uso racional de antibióticos, el fortalecimiento de las medidas de control de infecciones en entornos de atención médica y la promoción de nuevas investigaciones para el desarrollo de terapias antimicrobianas innovadoras (18,19).
CONCLUSIONES
Se identificó una alta frecuencia de bacterias productoras de BLEE en E. coli y Klebsiella spp, lo que subraya la importancia de monitorear y comprender la prevalencia de resistencia bacteriana. Este hallazgo representa un desafío significativo en el tratamiento de infecciones, especialmente considerando la resistencia observada a ciertos antibióticos comúnmente utilizados. Los resultados resaltan la necesidad de implementar estrategias de control de infecciones y fomentar un uso responsable de los antibióticos para prevenir el desarrollo y la propagación de la resistencia. Además, este estudio establece una alerta sobre la presencia significativa de cepas resistentes a los antibióticos en muestras de orina, sangre y tejido blando.
CONFLICTO DE INTERESES.
Los autores declaran que no existe conflicto de intereses para la publicación del presente artículo científico.
FINANCIAMIENTO
Autofinanciado
REFERENCIAS BIBLIOGRÁFICAS
1. World Health Organization. Global antimicrobial resistance and use surveillance system (GLASS) report 2022. 2022. Geneva. https://www.who.int/publications/i/item/9789240062702.
[ Links ]
2. Antimicrobial Resistance Collaborators. Global burden of bacterial antimicrobial resistance in 2019: a systematic analysis. Lancet. 2022;399 (10325):629-655. DOI: 10.1016/S0140-6736(21)02724-0.
[ Links ]
3. Miranda Mª C. Escherichia coli portador de betalactamasas de espectro extendido: resistencia. Sanid. Mil. 2013;69(4):244-248. DOI:10.4321/S1887-85712013000400003.
[ Links ]
4. Echeverri L, Cataño J. Klebsiella pneumoniae como patógeno intrahospitalario: epidemiología y resistencia. Iatreia. 2010; 23(3):240-249. https://acortar.link/oalm1H
[ Links ]
5. Khalifa S, Abd El-Aziz A, Hassan R, Abdelmegeed E. β-lactam resistance associated with β-lactamase production and porin alteration in clinical isolates of E. coli and K. pneumoniae. PLoS One. 2021;16(5): e0251594. DOI: 10.1371/0251594.
[ Links ]
6. Kiratisin P, Apisarnthanarak A, Laesripa C, Saifon P. Molecular characterization and epidemiology of extended-spectrum-beta-lactamase-producing Escherichia coli and Klebsiella pneumoniae isolates causing health care-associated infection in Thailand, where the CTX-M family is endemic. Antimicrob Agents Chemother. 2008; 52(8):2818-2824. DOI: 10.1128/AAC.00171-08
[ Links ]
7. Ho PL, Cheung YY, Wang Y, et al. Characterization of carbapenem-resistant Escherichia coli and Klebsiella pneumoniae from a healthcare region in Hong Kong. Eur J Clin Microbiol Infect Dis. 2016;35(3):379-385. DOI: 10.1007/s10096-015-2550-3.
[ Links ]
8. Cantón R, Loza E, Aznar J, et al. Antimicrobial susceptibility trends and evolution of isolates with extended spectrum β-lactamases among Gram-negative organisms recovered during the SMART study in Spain (2011-2015). Rev Esp Quimioter. 2018; 31(2):136-145.https://seq.es/wp-content/uploads/2018/04/canton12mar2018.pdf
[ Links ]
9. Nasiri M, Mirsaeidi M, Mousavi S, et al. Prevalence and Mechanisms of Carbapenem Resistance in Klebsiella pneumoniae and Escherichia coli: A Systematic Review and Meta-Analysis of Cross-Sectional Studies from Iran. Microb Drug Resist. 2020;26(12):1491-1502. DOI: 10.1089/mdr.2019.0440.
[ Links ]
10. Cheng M, Stenstrom R, Paquette K, Stabler S, Akhter M, Davidson A, et al. Blood culture results before and after antimicrobial administration in patients with severe manifestations of sepsis: a diagnostic study. Ann Intern Med. 2019;171(8):547-54. DOI: 10.7326/M19-1696.
[ Links ]
11. Pitout JDD, Nordmann P, Poirel L. Carbapenemase-Producing Klebsiella pneumoniae, a Key Pathogen Set for Global Nosocomial Dominance. Antimicrob Agents Chemother. 2015;59(10):5873-84. DOI: 10.1128/AAC.01019-15.
[ Links ]
12. Chotiprasitsakul D, Han J, Cosgrove S, et al. Comparing the Outcomes of Adults with Enterobacteriaceae Bacteremia Receiving Short-Course Versus Prolonged-Course Antibiotic Therapy in a Multicenter, Propensity Score-Matched Cohort. Clin Infect Dis. 2018;66(2):172-177. DOI: 10.1093/cid/cix767.
[ Links ]
13. Shao C, Wang W, Liu S, Zhang Z, Jiang M, Zhang F. Molecular Epidemiology and Drug Resistant Mechanism of Carbapenem-Resistant Klebsiella pneumoniae in Elderly Patients with Lower Respiratory Tract Infection. Front Public Health. 2021; 9 (1):669173. DOI: 10.3389/fpubh.2021.669173.
[ Links ]
14. Zowawi H, Forde B, Alfaresi M, Alzarouni A, Farahat Y, Chong T, et al. Stepwise evolution of pandrug-resistance in Klebsiella pneumoniae. Sci Rep. 2015;5 (1):15082. DOI: 10.1038/srep15082.
[ Links ]
15. World Health Organization (WHO). Global priority list of antibiotic-resistant bacteria to guide research, discovery, and development of new antibiotics. 2017. https://acortar.link/lE48Ky
[ Links ]
16. Stanley I, Kajumbula H, Bazira J, Kansiime C, Rwego I, Asiimwe B. Multidrug resistance among Escherichia coli and Klebsiella pneumoniae carried in the gut of out-patients from pastoralist communities of Kasese district, Uganda. PLoS One. 2018;13(7): e0200093. DOI: 10.1371/journal.pone.0200093.
[ Links ]
17. Guaraca L, Carchipulla C, Ortiz J. Infección del tracto urinario por enterobacterias en pacientes del laboratorio “San José”- Azogues. Rev.Vive. 2023;5(14):507-1. https://acortar.link/8tsIww
[ Links ]
18. Klein E, Tseng K, Pant S, Laxminarayan R. Tracking global trends in the effectiveness of antibiotic therapy using the Drug Resistance Index. BMJ Glob Health. 2019; 4(2): e001315. DOI: 10.1136/bmjgh-2018-001315.
[ Links ]
19. Dyar O, Huttner B, Schouten J, Pulcini C; ESGAP (ESCMID Study Group for Antimicrobial stewardshiP). What is antimicrobial stewardship? Clin Microbiol Infect. 2017;23(11):793-798. DOI: 10.1016/j.cmi.2017.08.026.
[ Links ]