INTRODUCCIÓN
La Organización Mundial de la Salud OMS define a las zoonosis como toda enfermedad o infección que se trasmite naturalmente de los animales al ser humano 1. Dentro de este tipo de enfermedades se incluye a la influenza, la toxoplasmosis y la salmonelosis, por mencionar tan solo unas pocas 2. En el mundo actual, la cría, exportación y venta de animales de consumo o compañía se produce con gran rapidez y alcanza volúmenes de intercambio que superan cualquier control sanitario; se trata, de hecho, de una de las principales fuentes de trasmisión de agentes microbianos causantes de zoonosis 3.
En la actual coyuntura pandémica, las zoonosis han adquirido una especial relevancia. En efecto, la COVID-19 -causada por el coronavirus SARS CoV-2- es considerada por muchos como una nueva zoonosis, altamente contagiosa 4. Se ha propuesto que este nuevo coronavirus evolucionó originalmente en murciélagos, y se trasmitió a los humanos a través de hospedadores intermediarios que, aunque aún no ha sido confirmado, podrían ser mamíferos como el pangolín malayo u otros animales exóticos 3. La propuesta no es novedosa, puesto que desde hace varias décadas se sabe que animales como los bovinos, cerdos, perros, gatos, y aves pueden infectarse con coronavirus que pueden transmitirse posteriormente a los seres humanos; de hecho, es lo que ocurrió hace algunos meses en granjas de visones, en Noruega 4.
Entre los agentes infecciosos causantes de zoonosis figura Staphylococcus aureus. Se trata de una bacteria patógena que forma parte de la flora normal de la piel, las glándulas cutáneas y las membranas mucosas, incluida las fosas nasales de individuos sanos 5. S. aureus es el principal causante de enfermedades intrahospitalarias graves pues, ocasionalmente, coloniza el torrente sanguíneo y los tejidos blandos de las vías respiratorias inferiores, ocasionando infecciones profundas graves, como la endocarditis y la osteomielitis 5,6. Este problema de salud pública se ve agravado por la frecuente colonización de las fosas nasales de animales, tanto de compañía como de granja 7. La exposición frecuente y muy cercana de los humanos con dichos animales favorece la propagación de este patógeno. Aún más preocupante es el surgimiento y diseminación de las cepas de S. aureus resistentes a antibióticos, particularmente aquellos pertenecientes a la clase de los beta-lactámicos, como la meticilina 7,8.
Este antibiótico es un derivado semi-sintético de la penicilina, que fue introducido en Europa en 1959; tan solo un año después se detectó la primera cepa de Staphylococcus aureus resistente a la meticilina -denominadas SARM (por S. aureus Resistente a Meticilina) cuya resistencia se debe a la producción de enzimas β-lactamasas, codificadas por el gen mecA 9. Lo preocupante de esta resistencia es que se transfiere en forma horizontal de una célula a otra; además, la resistencia a meticilina está asociada con la resistencia a otros antibióticos, como la vancomicina, un glucopéptido de elección en el tratamiento de infecciones graves causadas por S. aureus 9,10. Por tal razón, las opciones terapéuticas frente a infecciones causadas por cepas SARM se reducen de manera drástica.
Desde su aparición en la década de los cincuenta, las cepas SARM han sido consideradas como los patógenos intrahospitalarios por excelencia; sin embargo, en los años ’90 del siglo pasado aparecieron nuevas variantes de estas cepas, de transmisión comunitaria 11. Esto llevó a descubrir, algunos años más tarde, que las cepas SARM pueden colonizar animales de granja, de compañía y salvajes, los cuales actúan como reservorios de las mismas. En general, los linajes de S. aureus en animales se derivan de cepas humanas, cuyas modificaciones genéticas determinan el cambio de especificidad del hospedador. El ganado vacuno es uno de los reservorios animales más importantes, pero la transmisión de estas cepas puede ocurrir entre animales de distintas especies que coexisten en una misma granja. Muchas de estas cepas no portan el gen mecA en su genoma, sino su ortólogo denominado mecC 12,13.
Pese a su importancia epidemiológica y a la relevancia que tiene la cría de animales para las economías de pequeños y medianos productores en los países del sur del continente, no se han publicado hasta la fecha revisiones sistemáticas de la literatura especializada relacionada con infecciones de origen zoonóticas causadas por cepas SARM en América del Sur. De allí que el objetivo principal de este trabajo sea sistematizar los conocimientos disponibles en relación con la ocurrencia de SARM en animales de granja, con énfasis en los países suramericanos. La divulgación de esta información contribuirá a la actualización de profesionales y estudiantes, tanto de medicina humana como veterinaria, en torno a este tema, permitiendo conocer las particularidades epidemiológicas, fenotípicas y moleculares de cepas SARM que circulan entre animales de granja.
MÉTODO
La investigación realizada fue documental, de tipo revisión sistemática, y estuvo basada en la metodología recomendada en declaración PRISMA (Preferred Reporting Items for Systematic reviews and Meta-analysis) 14. Para llevarla a cabo se definió una búsqueda de artículos científicos, sin restricción de idioma, disponibles en las siguientes bases de datos: SCOPUS, MEDLINE y SCIELO. Muy ocasionalmente se identificaron artículos en forma manual, a partir de citas presentes en otros estudios. Se emplearon los siguientes criterios de elegibilidad: artículos publicados en los últimos diez años, que contengan información acerca de la ocurrencia de cepas SARM en animales de granja en países de América del Sur, que incluyan información relevante acerca de la patogenicidad de dichas cepas SARM (en humanos y animales), detalles genéticos (presencia de genes de resistencia), multirresistencia a antibióticos, datos de concentración inhibitoria mínima, entre otros.
Para la búsqueda en las diferentes bases de datos se usaron las siguientes palabras clave (dependiendo del idioma de las publicaciones): Staphylococcus aureus, methicillin resistance, SARM, MRSA, animals, farm animals, animales de granja, pet animals, livestock, Latin America, South America sus posibles variantes y combinaciones. Se revisaron los conceptos de acuerdo a los descriptores disponibles (Medical Subject Headings, MESH) y se conectaron los mismos con los operadores booleanos “AND”, “OR” y “NOT”. Se estructuró entonces la siguiente estrategia de búsqueda: (MRSA OR Methicillin resistant Staphylococcus aureus OR vancomycin resistant Staphylococcus aureus) AND (livestock OR poultry OR pig* OR goats OR sheep OR rabbits OR cattle OR chicken*) AND (Latin America OR South America OR Chile OR Colombia OR Venezuela OR Ecuador OR Peru OR Brazil OR Argentina OR Bolivia OR Uruguay OR Paraguay OR Guyana OR Surinam). Como se mencionó, la búsqueda igualmente se llevó a cabo en idioma español.
La información obtenida se analizó y sistematizó en base a las recomendaciones de la declaración PRISMA, según se presenta en el diagrama incluido en la Figura 1. El cribaje de los documentos se realizó en función de los siguientes criterios de exclusión: artículos que no se ajustan a los requerimientos de elegibilidad, artículos que no hayan sido publicados en revistas de alto impacto, artículos duplicados en dos o más bases de datos, otros artículos de revisión sistemática o metaanálisis.
RESULTADOS Y DISCUSIÓN
La búsqueda realizada permitió identificar 65 artículos relacionados con el tema de investigación planteado; sin embargo, luego del respectivo proceso de cribaje basado en los criterios anteriormente establecidos y que se detallan en la Figura 1, se seleccionaron finalmente 19 trabajos de investigación para su posterior análisis.
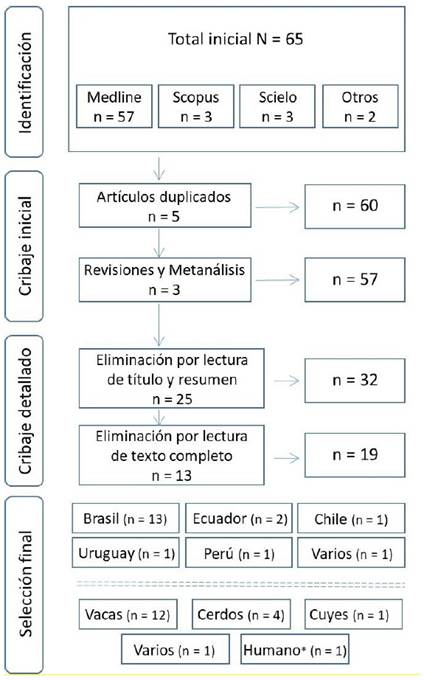
Figura 1 Flujograma del proceso de selección de artículos en base a recomendaciones de la declaración PRISMA (14).
De los 19 estudios seleccionados, 13 (68,42%) se realizaron en Brasil 12,13,15,25, y 2 (10,52%) en Ecuador 26,27, mientras tan solo un estudio de este tipo se ha llevado a cabo en Chile, Uruguay y Perú, respectivamente 28,30. Por último, un estudio se realizó simultáneamente en Brasil, Colombia y Argentina 31 (Figura 2A). No se encontraron artículos que refieran estudios similares y que hayan sido realizados en los demás países de Suramérica.
La mayoría de los estudios se centran exclusivamente en ganado vacuno (vacas lecheras) (N= 12, 63,15%), mientras que cuatro de ellos (21,05%) evaluaron la presencia de SARM en cerdos, uno (5,26%) lo hizo en cuyes (Cavia porcellus), y otro (5,26%) evaluó la presencia de este tipo de bacterias en diferentes animales (incluyendo vacas, ovejas, caballos, cerdos, cabras e, incluso, seres humanos) (Figura 2B). Pese a que uno de los trabajos publicados refiere al aislamiento de una cepa SARM perteneciente al clon ST398, a partir de una muestra de sangre de un paciente humano, se trata de una cepa que es exclusiva de animales 27. Ninguno de los artículos consultados refiere resultados obtenidos a partir de aves de corral, conejos u otros animales de granja.
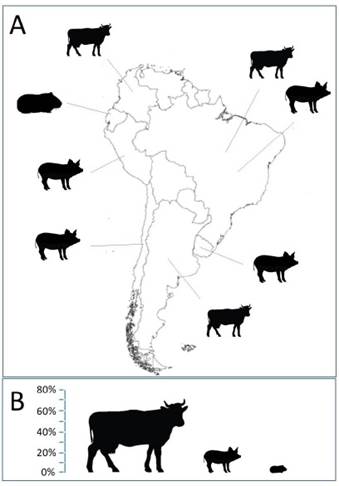
Figura 2 A) Origen geográfico de los artículos incluidos en la presente revisión; B) Representación porcentual de los artículos incluidos en función del tipo de animal estudiado.
Como era de esperarse, vistos los resultados anteriores, cuatro estudios (21.05%) emplearon muestras de leche para el aislamiento primario de las cepas de S. aureus, mientras que otros diez trabajos (52,63%) partieron directamente de cepas aisladas con anterioridad (Tabla 1). En seis casos, que equivalen al 31.57% de los artículos consultados, las cepas se aislaron también a partir de hisopados nasales de animales. De igual forma, el período de muestreo se especifica en seis (31,57%) de los estudios seleccionados. Por otra parte, trece trabajos (68,42%) mencionan que se estudiaron animales enfermos, mientras que en seis artículos (31,57%) se hace referencia a la identificación de cepas de Staphylococcus no aureus.
Tabla 1 Datos relevantes presentados en los artículos incluidos en la presente revisión sistemática
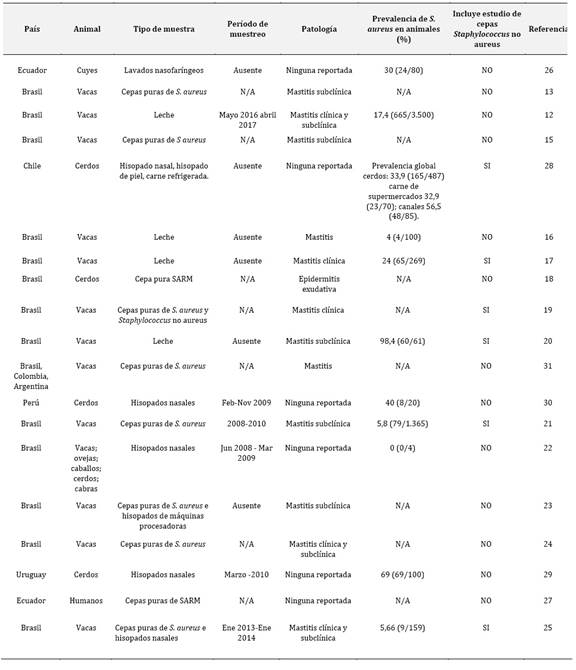
N/A: no aplica
Tabla 2 Características genotípicas y fenotípicas de las cepas de S. aureus presentadas en los estudios incluidos en la presente revisión sistemática.
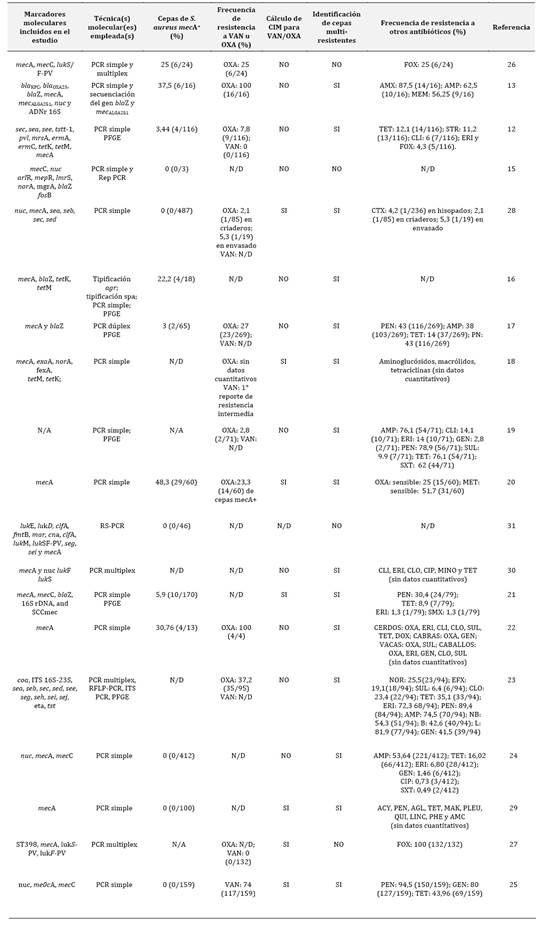
N/A: no aplica; N/D: no determinado OXA: oxacilina; TET: tetraciclina; STR: estreptomicina; CLI: clindamicina; ERI: eritromicina; FOX cefoxitina; AMX: amoxicilina; AMP: ampicilina; MEM: meropenema; PEN: penicilina; PN: penicilina+novoviocina. GEN: gentamicina; SUL: sulfonamida; SXT: trimetoprima+sulfametoxazol; MET: meticilina; SMX: sulfametoxazol; SUL: sulfamidas; DOX: doxiciclina; CLO: cloranfenicol; NOR: norfloxacina; EFX: enrofloxacina; NB: novobiocina; B: bacitracina; L: lincomicina: CIP: ciprofloxacina; ACY: aminociclitoles; AGL: aminoglicósidos; MAK: macrólidos; PLEU: pleuromutilina; QUI: quinolonas; PHE: Fenicoles; MINO: minoxiclina. Genes relacionados con la resistencia a los β-lactámicos: blaKPC, blaOXA23, blaZ, mecA, fosB, mecALGA251. Genes que codifican para sistemas de bombas de flujo: arlR, mepR, lmrS, norA, mgrA. Genes que codifican para enterotoxinas: sea, seb, sec, sed, seh, sej
Tal y como puede observarse en la Tabla 1, todos los artículos incluidos en esta revisión sistemática utilizaron técnicas de biología molecular para detectar genes relacionados con la resistencia a los diferentes antibióticos, o con la virulencia de las cepas SARM estudiadas. La identificación de las cepas aisladas se realizó en la mayoría de los casos por PCR simple y posterior secuenciación (N=14, 73,68%); sin embargo, cuatro estudios (21,05%) refieren el empleo de la técnica PCR multiplex, para la detección de más de un marcador molecular o gen de resistencia, y uno (5,26%) reporta el uso de la técnica de PCR en formato dúplex. Siete de estos artículos (42,10%) reportan, además, la secuencia de los primeros empleados para la detección de dichos marcadores 13,15,17,20,23,31.
La técnica de análisis de restricción de fragmentos amplificados por PCR (Restriction Site-PCR) fue empleada solo por un grupo de investigación para genotipificar las diferentes cepas aisladas en su estudio 31. Por su parte, el uso de la técnica de electroforesis en campo pulsado (PFGE) se menciona en seis (31,57%) de los estudios publicados, y fue igualmente empleada para la tipificación de las cepas aisladas 16,17,19,23,25,30. Finalmente, solo un artículo (5,26%) refiere la secuenciación nucleotídica de los marcadores moleculares amplificados 13, mientras que otro reporta la secuenciación del genoma completo de una cepa SARM aislada a partir de cerdos en Brasil 16.
Para la detección de la resistencia a la meticilina, en todos los estudios se optó por la amplificación del gen mecA mediante PCR (Tabla 2). La proporción de cepas mecA+ reportadas en los diferentes estudios osciló entre el 3 y el 100%.
En diez de los artículos consultados (52,63%) se reporta resistencia a oxacilina y/o vancomicina, datos que fueron obtenidos mediante la realización de antibiogramas. Destaca la cantidad de artículos que refieren resistencia a múltiples antibióticos (N=15, 78,94%). En diez de ellos se reporta la presencia de más de un marcador de resistencia en las cepas evaluadas 12,13,17,18,21,23,25,29,30, mientras que cuatro publicaciones reportan multiresistencia en base a ensayos fenotípicos 12,21,22,29. Finalmente, solo siete trabajos (36,84%) reportan la Concentración Inhibitoria Mínima (CIM) para los antibióticos evaluados 18,20,21,25,27,29.
Discusión
El presente estudio sistematiza los conocimientos disponibles en relación con la ocurrencia de cepas SARM en animales de granja en América del Sur, a partir de la información disponible en 19 artículos científicos publicados en los últimos 10 años. La mayoría de estos trabajos se refieren a estudios llevados a cabo en Brasil y estuvieron relacionados con ganado vacuno. Se encontró información de relevancia en cuanto a la ocurrencia de dichas cepas en animales de granja, algunas de las cuales estaban relacionadas, además, con seres humanos. Por otra parte, varios trabajos reportan la detección de cepas SARM multiresistentes, lo cual fue corroborado mediante el empleo de técnicas de biología molecular.
En un estudio publicado recientemente por Barberato-Filho y sus colaboradores (32), se presentaron los resultados de una revisión sistemática de la literatura relacionada con la prevalencia de cepas SARM en animales destinados a la producción de alimentos en granjas de la totalidad del continente americano. Dicho estudio solo incluyó cinco artículos cuyos resultados se refieren a trabajos realizados en América del Sur, de los cuales tres fueron ejecutados en Brasil. De estos cinco estudios, dos se refieren a vacas lecheras y tres a cerdos. En la presente revisión, se extiende la información a un total de 19 estudios, aunque sigue siendo muy evidente la mayor importancia que se le presta al ganado vacuno y porcino (89,47% de los trabajos aquí incluidos).
Llama poderosamente la atención la escasez de estudios relacionados con ganado ovino o caprino, muy frecuentes en distintos países del subcontinente, y la ausencia de reportes relacionados con otro tipo de animales como aves, conejos o caballos. De hecho, todos estos animales constituyen reservorios de cepas SARM, tal y como como se destaca en un meta-análisis publicado en el año 2018 33.
La presente recopilación bibliográfica también permitió identificar que existe un importante porcentaje de cepas de SARM multiresistentes circulando entre animales de granja en la región, lo cual puede atribuirse -en gran medida- al uso indiscriminado de antibióticos por parte de médicos veterinarios o productores agropecuarios. Se trata de una práctica muy común que privilegia la promoción del crecimiento de los animales, así como la prevención a ultranza de enfermedades infecciosas que pondrían en peligro la salud de las multitudinarias cohortes de animales que son criados en algunas granjas y que son fuente de importantes ingresos económicos 34,35. No obstante, es bien sabido que la presión selectiva impuesta por la utilización abusiva y sin control de este tipo de fármacos favorece la aparición de cepas resistentes y multiresistentes, debido a la facilidad con la que los determinantes genéticos de este tipo de resistencias (por ejemplo, plásmidos e integrones) se diseminan entre comunidades bacterianas 36. De forma que los resultados aquí presentados constituyen una señal de alarma adicional, que enfatiza la necesidad de controlar de manera estricta el uso de antibióticos con fines comerciales. Este tipo de restricciones son fundamentales para reducir la probabilidad de aparición de cepas SARM multiresistentes, y su posterior transmisión hacia los seres humanos.
Entre los resultados obtenidos en el presente estudio destaca también el uso de técnicas de biología molecular para la identificación de cepas SARM. Desde su publicación en el año 2003, por el grupo de Hallin y colaboradores 37, la amplificación del gen mecA mediante PCR convencional se ha convertido en el método de referencia para detectar aislados de SARM de manera rápida, barata y eficaz, y sin necesidad de llevar a cabo ensayos fenotípicos 38. Como hemos visto, la mayoría de los estudios aquí reportados hicieron uso de este método, confirmando su utilidad para fines diagnósticos.
Llama la atención que sólo un trabajo refiera el uso de técnicas de secuenciación masiva para la caracterización genómica de las cepas estudiadas 21. En efecto, el uso cada vez más frecuente de este tipo de técnicas ómicas y la disminución muy significativa de su costo, ha hecho posible conocer las particularidades genéticas de todo tipo de microorganismos, particularmente de aquellos que causan enfermedades en humanos 39; también ha permitido conocer la distribución geográfica de estas cepas, mediante aproximaciones filogenómicas 40. De hecho, este tipo de herramientas metodológicas son fundamentales para comprender mejor y controlar de manera más eficaz las enfermedades -incluso las epidemias- causadas por este tipo de microorganismos. Es evidente entonces la necesidad de ampliar este tipo de estudios en el futuro, con el fin de mejorar el conocimiento sobre las particularidades genómicas y epidemiológicas de las cepas SARM que circulan en nuestra región.
Lamentablemente, no es posible extrapolar los resultados obtenidos en los diferentes trabajos incluidos en la presente revisión a otras realidades debido, principalmente, a que la mayoría de ellos han sido realizados en Brasil y en ganado vacuno. Resulta por lo tanto imprudente intentar establecer la prevalencia real de este tipo de infecciones en otros animales de granja, pues los resultados disponibles actualmente son muy limitados. Por lo demás, en algunos de los estudios referidos no se incluye información sobre los períodos de recolección de muestras lo cual limita de manera importante cualquier análisis temporal.
En síntesis, la presente revisión sistemática aporta información relevante que confirma a las cepas SARM como patógenos que circulan fuera de los ambientes intrahospitalarios. Estas cepas, muchas de las cuales son multiresistentes, forman parte de la microbiota de los animales criados con fines de consumo en la región, con los cuales los seres humanos tienen contacto cercano y muy frecuente. De allí la importancia que tiene la producción de información de calidad, relacionada con este importante aspecto de salud pública. Es a partir de este tipo de información que se podrán mejorar las políticas de control y prevención de infecciones causadas por este tipo de bacterias patógenas.
CONCLUSIÓN
A partir del análisis de los 19 artículos incluidos en la actual revisión bibliográfica, se puede concluir que las cepas SARM son patógenos que circulan fuera de los límites de los centros hospitalarios en América del Sur, particularmente en comunidades extraurbanas y rurales, y que pueden encontrar nichos para establecerse y multiplicarse, más allá de los tejidos y órganos del ser humano. Se confirma que los animales de granja son importantes reservorios de este tipo de cepas, a partir de los cuales pueden transmitirse a las personas con las que están en contacto íntimo y frecuente. Por lo tanto, es necesario vigilar de cerca esta situación, para controlar de manera rápida y eficaz la propagación de las infecciones causadas por este tipo de bacterias patógenas. Se trata de un tema de gran importancia, que debe ser tomado en cuenta y abordado por la comunidad científica, para generar conocimiento de calidad que contribuya con tales objetivos















