INTRODUCCIÓN
La resistencia microbiana es un proceso natural de evolución de los microorganismos. Este proceso puede acelerarse por estimulaciones del medio externo que obliga a los patógenos a adaptarse para su supervivencia. Uno de los procesos de adaptación es el desarrollo de mecanismos de resistencia, que incluye la producción de enzimas, cambios en permeabilidad bacteriana, utilización de Bombas de expulsión y modificación de la proteína diana que altera el lugar del punto de unión del antibiótico por otra enzima, cambiando su función o una variación en su información genética 1.
Estos mecanismos están presentes en diferentes genes bacterianos, llamados resistoma, y pueden ser transferidos de una bacteria a otra por conjugación, a través de plásmidos, así como también por trasformación y transducción 2.
Las bacterias son capaces de adquirir varios de estos mecanismos a la vez, que le confieren multirresistencia a los antimicrobianos. En el mundo surgen nuevos mecanismos de resistencia y se extienden rápidamente en la población con la amenaza de convertir a las enfermedades infecciosas comunes en intratables, prolongando la infección, que puede llevar a la muerte 3.
La Organización mundial de la Salud (OMS) prevé que hasta el 2050 ya no se cuente con antibióticos para el tratamiento de enfermedades, por lo que las infecciones bacterianas causadas por patógenos multirresistentes será la primera causa de muerte a nivel mundial 4.
La resistencia bacteriana, por lo tanto, se ha convertido en un gran problema a escala mundial, un número considerable de bacterias de importancia medica han adquirido resistencia a los antibióticos y entre este grupo destaca, Escherichia coli, declarada por la OMS como un patógeno prioritario dentro del campo de la investigación y desarrollo de nuevas terapias farmacológicas, debido, a que es causante de infecciones intrahospitalarias y comunitarias letales por su alta multirresistencia antibiótica (5.
En referencia, Escherichia coli es un patógeno que forma parte de la microbiota normal del ser humano. Sin embargo, existen patotipos de E. coli responsables de causar enfermedades intestinales y extra-intestinales 6. En América Latina se han reportado cepas de esta bacteria productoras de betalactamasas de espectro extendido 7,8, cepas resistentes a cefalosporinas de tercera y cuarta generación 9. Así como también, resistencia a carbapenémicos 10,11 y presencia de gen mcr-1 que le confiere resistencia a colistina 12. Resultando ineficaz el uso de tratamientos en mucho de los casos, debido al alto nivel de resistencia a los antibióticos que se asocia una mayor morbilidad y mortalidad del ser humano.
El objetivo de este estudio es conocer los principales mecanismos de resistencia desarrollados o adquiridos por Escherichia coli dentro de América Latina. Para lo cual se realizó una revisión de fuentes primarias de la literatura presente hasta el momento. En Ecuador no existe una recopilación de los principales mecanismos de resistencia de E. coli en los últimos años a nivel de América Latina. Por tanto, surge la necesidad de realizar la síntesis de evidencia disponible con el propósito de coadyuvar la vigilancia activa de este patógeno, que impida, posteriormente, se convierta en una bacteria intratable. Esta información, además, puede servir de fundamento para mejorar las políticas de control de resistencia microbiana y de guiar al personal de salud al momento de aplicar un tratamiento.
MÉTODO
Se realizó una revisión sistemática de la evidencia científica con los artículos publicados desde enero del 2000 al 2020. La actual revisión se reporta de acuerdo a los elementos de informes preferidos para revisiones sistemáticas y pautas de meta-análisis (PRISMA). Este responde a la siguiente pregunta clínica: ¿Cuáles son los mecanismos principales de resistencia bacteriana en Escherichia coli en América Latina?
La presente investigación, se realizó de acuerdo a los siguientes pasos:
1. Determinar la pregunta de investigación
2. Búsqueda de los documentos relevantes
3. Selección de los estudios
4. Extracción de datos
5. Síntesis y reporte de resultados.
Estrategia de búsqueda
La búsqueda de información se recolectó de las siguientes bases de datos PubMed, Scopus, Scielo y Science Direct. Se utilizaron operadores booleanos y palabras clave de acuerdo con cada sistema de datos. Además, se revisaron las referencias citadas de los estudios incluidos, para no perder artículos que no se habían detectado previamente.
Pregunta PEO
P: Escherichia coli
E: Antimicrobianos
O: Mecanismos de resistencia
Se utilizó la siguiente sintaxis de búsqueda: “Escherichia coli” OR “E. coli AND “antibiotic resistance” OR “drug resistance” OR “drug resistance, microbial” OR “antibacterial resistance” OR “antibiotic resistance” OR “antimicrobial resistance” AND “mechanisms of bacterial resistance”.
Los artículos inicialmente identificados se almacenaron en el gestor de referencias bibliográficas Zotero, desde el cual se excluyeron los archivos que se encontraban por duplicado.
Criterios de inclusión
1. Publicaciones cuyo diseño metodológico corresponda a: ensayos clínicos aleatorizados (ECA), ensayos de intervención no aleatorizados (EINA), estudios de cohorte y transversales.
2. Estudios que evalúen o discutan el papel de la resistencia bacteriana por E. Coli.
3. Investigaciones realizadas desde el mes enero del 2000 al 2020
4. Manuscritos en inglés, español y portugués.
5. Estudios que mencione el método utilizado, microbiológico y/o molecular, para la detección de resistencia bacteriana.
Criterios de exclusión
1. Investigaciones incompletas y fuera del campo de estudio.
2. Estudios de fuentes secundarias
3. Documentos de investigación como tesis, informes, artículos de revisión, cartas al editor.
Selección de estudios y extracción de datos
1. Aplicación de la web gratuita Rayyan,
2. Revisión independiente, por parte de los autores, de títulos, resúmenes y el texto completo para su elegibilidad.
3. Ejecución de prueba piloto con 10 artículos para evaluar la claridad de los criterios de elegibilidad.
4. Ejecución de una segunda prueba piloto con 5 artículos antes de comenzar la selección de texto completo en tutoría con un experto.
5. Extracción de datos en Microsoft Excel, con las respectivas variables (tipo de estudio, autores, fecha de publicación, país origen, población estudio, método utilizado, hallazgos principales)
De las bases de datos utilizadas para la recopilación de la información se identificaron 430 elementos relevantes para su análisis. Posteriormente se realizó una lectura de títulos y resúmenes, excluyendo 385 artículos no relevantes. De los 45 artículos sobrantes, para su lectura a texto completo, se eliminaron 27 de acuerdo a los criterios de inclusión y exclusión.
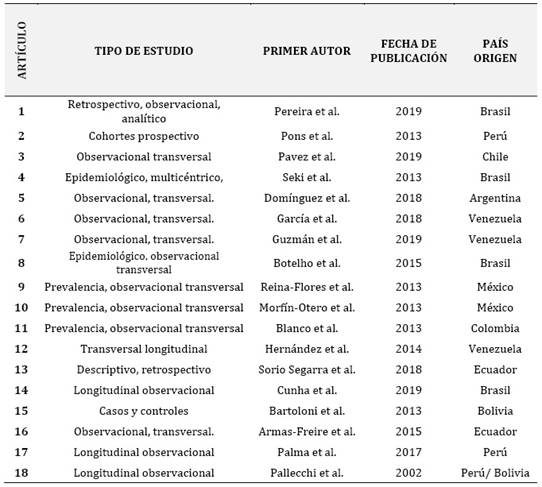
Finalmente se incluyó un total de 18 artículos relevantes para el análisis de la revisión sistemática. Tabla 1.
DESARROLLO Y DISCUSIÓN
Características y distribución de los estudios
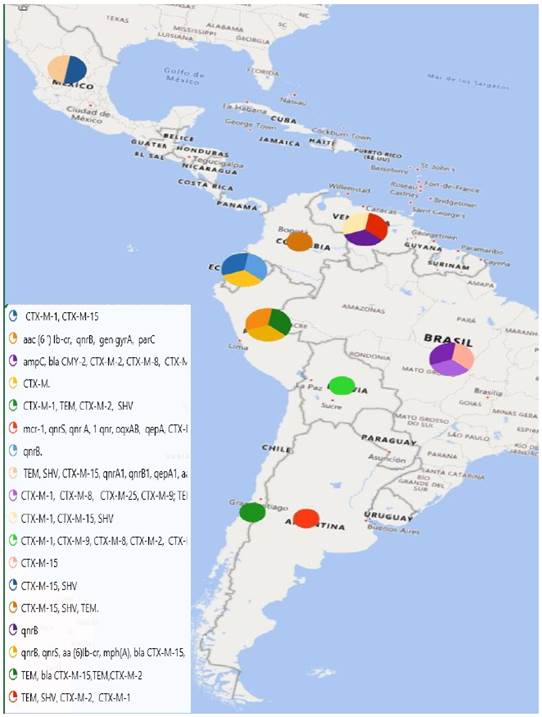
La revisión sistemática incluyo 18 estudios relevantes, que corresponden a 7 países. Dos pertenecientes a Ecuador, cuatro estudios de Brasil, uno de Colombia, 3 de Venezuela, dos de Perú, dos de Bolivia, uno de Chile, dos de México y uno de Argentina. La Figura 1 detalla de manera geográfica la distribución de los diferentes genes que confieren resistencia a E. coli, reportados dentro de Latinoamérica. La presencia de genes de resistencia a los betalactámicos fue el mecanismo común y el más reportado de resistencia en E. coli seguido de reportes de genes de resistentes a quinolonas.
Tabla 2.Características generales de los artículos incluidos.
Distribución de los mecanismos de resistencia de Escherichia coli
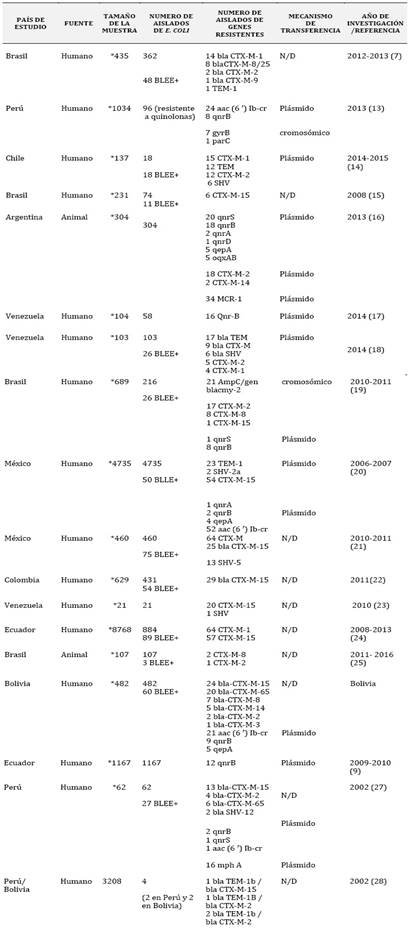
La Escherichia coli es un patógeno que ha desarrollado diferentes mecanismos para evadir la acción de los distintos antimicrobianos. De los estudios incluidos 15 de estos reportaron resistencia de E. coli mediante la producción de betalactamasas de espectro extendido (BLEE). Dentro del grupo de betalactamasas, las de tipo cefotaximasas (CTX-M) fueron las enzimas, que en mayor cantidad se reportaron como mecanismo de resistencia a los betalactámicos en diferentes países de Latinoamérica. Tabla 3.
Dentro del grupo de las cefotaximasas el gen CTX-M-15 fue reportado en la mayoría de las investigaciones, siendo el más representativo, seguido del gen CTX-M-2 que es reportado tanto en humanos como animales. Perú y Bolivia reporta el hallazgo del gen CTX-M-65 descrito por primera vez en estos países. (Tabla 3).
Se destaca un nuevo mecanismo de resistencia de Escherichia coli, y es el de la resistencia a las quinolonas. Perú, México, Brasil, Bolivia, Ecuador y Venezuela entre sus reportes destaca el gen aac (6′) Ib-cr como mecanismo de resistencia a quinolonas. Tabla 3.
En los estudios incluidos, no se reporta genes de resistencia a otro tipo de antibióticos, siendo estos los más característicos y reportados dentro de la investigación. Tabla 3.
Discusión
La producción de enzimas betalactamasas es el mecanismo de resistencia desarrollado por las enterobacterias hacia la acción de los betalactámicos. La producción de betalactamasas de espectro extendido (BLEE) tiene acción inhibitoria a penicilinas, cefalosporinas (con excepción de las cefamicinas, monobactámicos y carbapenémicos 29. Desde la aparición en 1980 de las penicilinasas de tipo bla SHV y bla TEM han surgido varios cambios de evolución en la resistencia bacteriana, como es la aparición a finales de 1990 de las betalactamasas de tipo CTX-M 30.
Entre las betalactamasas, los grupos más relevantes son TEM-1, SHV-1 y CTX-M (31).. El grupo CTX-M son las enzimas con mayor distribución en todo el mundo (32), así lo detallan varios reportes de estudios en animales y humanos.(16,33; aunque su prevalencia todavía no está clara, pero puede deberse a la trasferencia de cromosomas entre especies a través de plásmidos conjugativos 34. Es el caso de los grupos CTX-M encontrados en especies Kluyvera 35 y que actualmente lo encontramos en especies como E. coli 7,20,26,27.
La bibliografía encontrada concuerda con los resultados obtenidos, en donde, el grupo predominante de BLEE es CTX-M específicamente CTX-M-15, como el mecanismo de resistencia a los Betalactamicos adquiridos por E. coli dentro de América Latina.
Es importante recalcar que la resistencia por E. coli continua en evolución con el pasar de los años, y se ve reflejada en la investigación de Pallecchi et al. en el año 2002, en donde se reportó la presencia de genes de betalactamasas en niveles muy bajos y no hubo la presencia de genes de resistencia a quinolonas28, sin embargo luego de una década se ha presenciado un aumento de los mecanismos de resistencia de este patógeno, con mayor cantidad de aislados de genes de resistencia bacteriana a betalactamasas y quinolonas 7,13,26,33. Este suceso se lo podría relacionar con la transferencia de genes por plásmidos u otros elementos genéticos móviles, que confieren resistencia a otras bacterias a través del proceso de conjugación26,28,34) así como también la adquisición de genes resistentes, de manera zoonótico o alimentaria 36,37.
Los resultados obtenidos detallan también, importantes genes que confieren resistencia a las quinolonas, grupo de antibióticos importantes en el tratamiento de infecciones nosocomiales y comunitarias. El uso indiscriminado en la práctica médica, agrícola y alimentaria ha conllevado al desarrollo de resistencia bacteriana por diferentes patógenos como E.coli 38. El gen aislado en mayor medida en América Latina fue aaa 6 Ib-cr seguido del gen qnrB, ambos pertenecientes al grupo de genes mediados por plásmidos. Se ha reportado también en otros países del mundo 39.
CONCLUSIÓN
De acuerdo a los resultados obtenidos, se puede concluir que E. coli en América Latina expresa diferentes genes como mecanismo de resistencia a betalactámicos y quinolonas. Siendo la producción de BLEE, específicamente como grupo circulante predominante los genes CTX.M.15 y CTX-M-2, como mecanismo de resistencia a batalactamicos. En cuanto al mecanismo de resistencia a las quinolonas, se presenta por mutaciones cromosómicas y resistencia mediado por plásmidos, siendo este último el mecanismo de resistencia predominante con la expresión de los genes aac 6 Ib-cr y qnrB.
Por lo tanto, es importante atender esta situación, mediante la mejora de políticas de control de uso de los antimicrobianos. La resistencia a betalactámicos y quinolonas ha evolucionado rápidamente por lo que es indispensable establecer un buen diagnóstico en las diferentes patologías para establecer un tratamiento adecuado