INTRODUCCIÓN
A través del siglo XXI se han descrito diferentes brotes de Coronavirus. El primero en noviembre de 2002 en Guangdong- China, llamado Síndrome Respiratorio Agudo, causado por el Coronavirus (SARS-CoV), con un rango de mortalidad del 10%. En Jeddah, Arabia Saudita y Medio Oriente emerge un nuevo tipo de Coronavirus atribuyendo precisamente el nombre de Síndrome Respiratorio Agudo del Medio Oriente causado por (MERS-CoV) en enero de 2012 el cual mostró una tasa de mortalidad de 37% 1. Finalmente el actual coronavirus 2019-nCoV con características clínicas del mismo orden que los anteriores denominado Síndrome Respiratorio Agudo causado por el coronavirus tipo 2 (SARS-CoV-2) cuya primera detección fue en Wuhan, China en diciembre de 2019, causante de neumonía y oficialmente conocida como la COVID-19 según la Organización Mundial de la Salud (OMS) 2.
El nuevo coronavirus 2019-nCoV es el séptimo miembro de la familia de coronavirus que infectan a los humanos, su secuencia se obtuvo a partir de muestras de los primeros grupos de pacientes con neumonía de origen desconocido del Hospital Jinyintan de Wuhan. En este contexto se emplearon células epiteliales de vías respiratorias humanas; logrando clasificarlo en el subgénero sarbecovirus y subfamilia Orthocoronavirinae3.
La PCR (Reacción en cadena de la Polimerasa) es una técnica que hace posible la amplificación del Ácido Desoxirribonucleico (ADN) además de la detección simultánea del producto de amplificación y la cuantificación del material genético en una muestra. La técnica fue creada por Kary Mullis, es un proceso cíclico que amplifica exponencialmente pequeñas cantidades de ADN, tras la separación a altas temperaturas, dos cadenas de ADN podrían copiarse utilizando una enzima de unión, a partir de oligonucleótidos cortos o cebadores 4)(5)6). Dado que la PCR sólo amplifica cadenas de ADN, y los coronavirus son virus de Ácido Ribonucleico (ARN) de una sola cadena en sentido positivo recubiertos por una estructura de glicoproteínas y lípidos, se realiza una variante de la PCR estándar: la reacción en cadena de la polimerasa con transcriptasa inversa (RT-PCR). Para lo cual es necesario realizar la conversión del ARN monocatenario viral a ADN. En la RT-PCR, la muestra se añade a sondas que se unen únicamente a secuencias específicas del ADN retrotranscrito del virus y emiten fluorescencia y presenta en pantalla los resultados en tiempo real 7.
Los inmunoensayos incluyen algunas variantes, los de flujo lateral rápido, inmunoensayo de quimioluminiscencia automatizado y ELISA (Enzimoinmunoanálisis) que utilizan proteínas virales en dominios recombinantes de proteínas virales a las que se unen los anticuerpos humanos, en el caso que estén presentes en la sangre 8. Así para la determinación del virus en cuestión, se emplean proteínas de la nucleocàpside (NP) de murciélago, pues éste comparte más del 90% de identidad de los aminoácidos con todos los SARS-CoV 2, así como anticuerpos monoclonales conjugado anti-IgG y para IgM se emplea IgM antihumana 9.
Las características del virus causante de SARS-CoV-2 desconocidas en sus inicios, hizo que el método diagnóstico se fundamentara en características clínicas, estudios radiológicos e incluso identificación de bacterias causantes de neumonía lo que retardó el tiempo entre la identificación del patógeno, su tratamiento e implementación de medidas de control.
Tras la implementación de plataformas diagnósticas aparecieron incongruencias entre el resultado de las pruebas con la verdadera positividad o negatividad por lo que se requiere valorar el uso de los métodos de diagnóstico de laboratorio tanto moleculares como serológicas, caracterizar estas pruebas analizando la literatura científica actual que aborde la precisión diagnóstica y con ello determinar ventajas, usos y limitaciones y así aportar un sustento y apoyo informativo a la comunidad científica ecuatoriana.
MÉTODO
Se eligieron artículos originales contentivos de datos para construir la pregunta PICO (Paciente: Pacientes con sintomatología de SARS-CoV2; Intervención: Diagnostico a través del método molecular (RT-PCR); Comparación: Diagnóstico por pruebas serológicas mediante la cuantificación de IgG e IgM; Objeto variable: Valoración la especificidad y sensibilidad de las pruebas) publicados entre 31 de diciembre de 2019 al 05 de marzo de 2021 tanto en el idioma español e inglés.
La búsqueda de la información se realizó a través de las bases científicas Scopus y Web of Science, eligiendo artículos originales indexados, empleando palabras clave y conectores booleanos para cada base de datos. (Ver Tabla 1).
Los criterios de elegibilidad comprendieron investigaciones en las que realizaron el diagnóstico del virus a pacientes hospitalizados con sintomatología de SARS-CoV2 a través de hisopado nasofaríngeo y otros T-PCR en tiempo real dirigido a genes específicos del virus; ensayos de diagnóstico por métodos serológicos tales como Enzimoinmunoanàlisis (ELISA) o quimiolumninscencia dirigido a las inmunoglobulinas G y M u otras; todos los ensayos deben evaluar tanto sensibilidad y especificidad, entre otros parámetros de importancia. Entre los criterios de exclusión están ensayos a pacientes ambulatorios en laboratorios de nivel básico, valoración a partir de valores obtenidos a través de base de datos estudios y que no evaluaban la sensibilidad o especificidad. La selección de estudios se realizó por un solo revisor considerando una fase inicial de lectura de títulos y resúmenes, seguida de una fase de lectura a texto completo de las referencias potencialmente relevantes identificadas para su análisis y posterior correlación.
Tabla 1 Estrategias de búsqueda utilizada en las diferentes bases de datos.
| Combinación de términos | Base de datos | Criterios de inclusión y fecha de búsqueda | ||
|---|---|---|---|---|
| Scopus | Web Of Science | |||
| TITLE-ABS-KEY (severe AND acute AND respiratory AND syndrome OR coronavirus 2019 AND molecular AND diagnosis AND serological AND diagnosis) | 59 | Año: 2019-2021 Tipo de documento: Artículos originales Palabras clave : Human Idioma: Inglés Fecha de búsqueda : 20 de Febrero de 2020 y 05 de Marzo de 2021 | ||
| TITLE-ABS-KEY (severe AND acute AND respiratory AND syndrome OR coronavirus 2019 AND molecular AND diagnosis AND serological AND diagnosis) AND (LIMIT TO ( DOCTYPE, "ar")) AND (LIMIT-TO ( EXCACTKEYWORD, "HUMAN")) | 31 | |||
| (TS=(SARS COV 2 AND MOLECULAR DIAGNOSIS AND SENSITIVE) ) AND LANGUAGE: (English) AND DOCUMENT TYPES: (Article) Indexes=SCI-EXPANDED, SSCI, A&HCI, ESCI Timespan=2019-2021 | 30 | |||
| (TS=(SARS COV 2 AND SEROLOGICAL DIAGNOSIS AND SENSITIVE) ) AND LANGUAGE: (English) AND DOCUMENT TYPES: (Article) Indexes=SCI-EXPANDED, SSCI, A&HCI, ESCI Timespan=2019-2021 | 16 | |||
| TOTAL: | 90 | 46 | ||
RESULTADOS
mpleando combinaciones de términos y operadores booleanos en la búsqueda general de la base de datos Scopus, se obtuvo 59 artículos, tras una búsqueda adicional se sumaron 31 artículos más, dando un total de 90 artículos; para facilitar la búsqueda en la plataforma Web of Science se realizó una búsqueda avanzada de la cual se obtuvo un total de 46 artículos. Siguiendo la metodología prisma a partir de 136 artículos se obtuvo 25 estudios aptos para la revisión completa. (Ver Figura 1).
El mayor número de publicaciones halladas provienen de China (5) y Estados Unidos (4), países de Europa (8) y Asia (6) en diferentes países como Italia, Reino Unido, Holanda, España, Korea, aportan también importantes estudios mientras que, en América Latina se encontraron publicaciones procedentes de Brasil (1) y Ecuador (1).
Todos los estudios indican desarrollo y validación clínica de diferentes técnicas diagnósticas, estandarización de métodos, propuestas de nuevas metodologías diagnósticas entre ellas LAMP (Amplificación Isotérmica mediada por bucle de transcripción inversa), RT-PCR, variantes de la PCR como PCR anidada y RFLP (Técnica de Polimorfismos de longitud de fragmento de restricción) usando enzimas de restricción, así como validación de Kits comerciales o combinaciones de dos o más técnicas, presentando 25 estudios los valores de sensibilidad y especificidad, tipo de muestra empleada y genes blanco de detección; se describieron 17 elementos correspondientes al diagnóstico de SARS-CoV-2 por técnicas moleculares y 8 por técnicas serológicas. (Ver Tabla 2).
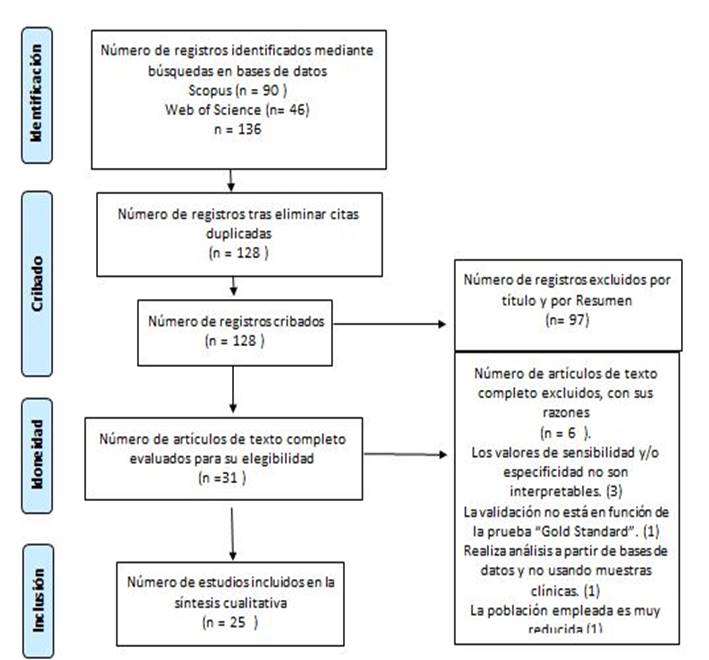
Figura 1 Diagrama de flujo de estrategia de búsqueda utilizada para la revisión de los métodos diagnósticos de SARS - CoV-2.
Tabla 2 Revisión de estudios sobre los métodos diagnósticos de SARS-CoV -2
| No. | BASE DE DATOS | AÑO | REVISTA | TÍTULO | AUTOR | PAÍS |
| 1 | WEB OF SCIENCE | 2020 | Heliyon | A one-step, one-tube real-time RT-PCR based assay with an automated analysis for detection of SARS-CoV-2 | Dharavath et ál. | INDIA |
| 2 | SCOPUS | 2021 | American Society for Clinical Pathology AJCP | A comparison of five SARS-CoV-2 molecular assays with clinical correlations | Procop, et ál. | USA |
| 3 | SCOPUS | 2020 | Nano BioMed ENG | A Rapid and high-sensitive real-time reverse transcription-polymerase chain reaction assay used for the detection of severe acute respiratory syndrome coronavirus 2 | Chen et ál | CHINA |
| 4 | SCOPUS | 2020 | J Med Virology | A simple method for detection of a novel coronavirus (SARS-CoV-2) using one-step RT-PCR followed by restriction fragment length polymorphism | Son et ál | VIETMAN |
| 5 | WEB OF SCIENCE | 2020 | Clinica Chimica Acta | Clinical Performance of A Semi-Quantitative Assay for SARS-CoV2 IgG and SARS-CoV2 IgM Antibodies | Jung et ál | USA |
| 6 | WEB OF SCIENCE | 2020 | PLoS Pathog | Comparative assessment of multiple COVID-19 serological technologies supports continued evaluation of point-of-care lateral flow assays in hospital and community healthcare settings | Pickering et ál | London, UK |
| 7 | WEB OF SCIENCE | 2020 | Journal of Clinical Virology | Comparison of commercial lateral flow immunoassays and ELISA for SARSCoV-2 antibody detection | Serrano et ál | ESPAÑA |
| 8 | SCOPUS | 2020 | Clinical Microbiology and Infection | Comparison of diagnostic accuracies of rapid serological tests and ELISA to molecular diagnostics in patients with suspected coronavirus disease 2019 presenting to the hospital | Ong et ál | HOLANDA |
| 9 | SCOPUS | 2020 | Journal of Clinical Virology | Contribution of VitaPCR SARS-CoV-2 to the emergency diagnosis of COVID-19 | Fournier et ál | FRANCIA |
| 10 | SCOPUS | 2020 | BMC Infect Dis | Detecting SARS-CoV-2 at point of care: preliminary data comparing loop-mediated isothermal amplification (LAMP) to polymerase chain reaction (PCR) | Österdahl et ál | London, UK |
| 11 | SCOPUS | 2020 | American Society for Microbiology mSphere | Development and clinical application of a rapid and sensitive loop-mediated isothermal amplification test for SARS-CoV-2 infection | Hu et ál | CHINA |
| 12 | WEB OF SCIENCE | 2021 | J Clin Lab Anal | Development and clinical application of a rapid SARS-CoV-2 antibody test strip: A multi-center assessment across China | Liao et ál | CHINA |
| 13 | SCOPUS | 2020 | IJMS | Development and evaluation of novel and highly sensitive single-tube nested real-time rt-pcr assays for sars-cov-2 detection | Yip et ál | HONG KONG |
| 14 | WEB OF SCIENCE | 2020 | Emerging Microbes and Infections | Development of a reverse transcription-loop-mediated isothermal amplification as a rapid early-detection method for novel SARS-CoV-2 | Baek et ál | KOREA |
| 15 | SCOPUS | 2020 | Indian J Med Res | Development of indigenous IgG ELISA for the detection of anti-SARS-CoV-2 IgG | Sapkal et ál | INDIA |
| 16 | SCOPUS | 2020 | Journal of Clinical Virology | Evaluation of rapid diagnosis of novel coronavirus disease (COVID-19) using loop-mediated isothermal amplification | Kitagawa et ál | JAPON |
| 17 | WEB OF SCIENCE | 2021 | Journal of Clinical Microbiology | Highly Sensitive and Specific Multiplex Antibody Assays To Quantify Immunoglobulins M, A, and G against SARS-CoV-2 Antigens | Dobaño et ál | ESPAÑA |
| 18 | WEB OF SCIENCE | 2020 | Sociedad Brasilera de Microbiologìa | Lower cost alternatives for molecular diagnosis of COVID-19: conventional RT-PCR and SYBR Green-based RT-qPCR | Dorlass et al. | BRAZIL |
| 19 | SCOPUS | 2021 | Journal of Molecular Diagnostics | Performance of Severe Acute Respiratory Syndrome Coronavirus 2 Real-Time RT-PCR Tests on Oral Rinses and Saliva Samples | Babady et al. | USA |
| 20 | SCOPUS | 2020 | Virology Journal | Poor sensitivity of "AccuPower SARS-CoV-2 real time RT-PCR kit (Bioneer, South Korea)" | Freire-Paspuel, B., Garcia-Bereguiain, M.A. | ECUADOR |
| 21 | SCOPUS | 2020 | Journal of Clinical Virology | Rapid and sensitive detection of SARS-CoV-2 RNA using the Simplexa™ COVID-19 direct assay | Bordi et ál. | ITALIA |
| 22 | SCOPUS | 2020 | Clinical Microbiology and Infection | Rapid and visual detection of 2019 novel coronavirus (SARS-CoV-2) by a reverse transcription loop-mediated isothermal amplification assay | Yan et ál. | CHINA |
| 23 | SCOPUS | 2020 | Nat Biomed Eng | Rapid lateral flow immunoassay for the fluorescence detection of SARS-CoV-2 RNA | Wang et ál. | CHINA |
| 24 | SCOPUS | 2020 | Clinica Chimica Acta | SARS-CoV-2 infection serology validation of different methods: usefulness of IgA in the early phase of infection. | Pieri et ál. | ITALIA |
| 25 | SCOPUS | 2020 | Journal of Clinical Virology | Validation and verification of the Abbott RealTime SARS-CoV-2 assay analytical and clinical performance | Degli-Angeli et ál | USA |
Diagnóstico molecular de SARS-CoV-2
La RT-PCR (Reacción en cadena de la polimerasa con retro transcripción). Es el método considerado como estándar de oro “Gold standard” para la detección del SARS-CoV-2, permite detectar el virus tras la aparición de la enfermedad y entre pacientes con síntomas desde leves hasta graves 10. Es eficiente, aunque costosa y poco asequible, de ahí la variabilidad y adecuación de la prueba diagnóstica en busca de disminución de costos, mejora del rendimiento y obtención de resultados en menor tiempo.
Emplear RT-PCR convencional con uso de reactivos como el Syber Green puede abaratar costos así lo concluye Dorlass et al., 11 reflejando adecuados valores de sensibilidad y especificidad aunque ofreciendo resultados en mayor tiempo. Una alternativa confiable es la NESTED RT-PCR (PCR anidada), ensayada por Yip et al., 12 pues muestra un aumento en la sensibilidad y especificidad al compararla con la RT-PCR no anidada y además se puede emplear diferentes especímenes como saliva en lugar del hisopado nasofaríngeo. Por otra parte, Son et al., 13 hizo uso de la RFLP-PCR, usando enzimas de restricción lo que permite diferenciar entre SARS-CoV y SARS-CoV-2, de gran importancia por la alta similitud del genoma entre los miembros de la familia Coronaviridae.
La PCR multiplex es una técnica que mejora la especificidad de detección, puede ser empleada en el ambiente intrahospitalario pues como indica el autor para detectar confecciones lo cual puede ser útil en épocas de incidencia de afecciones respiratorias 14. La técnica LAMP es una técnica de amplificación de ácidos nucleicos que emplea una combinación de dos o más conjuntos de cebadores. El ensayo de Dharavath et ál., 15 emplea un cebador dirigido al gen N y otros dos cebadores, uno dirigido a una región no superpuesta del gen N y otro dirigido al gen ORF-1ab. Los autores que aplicaron y validaron el ensayo la defienden como una técnica rápida que da resultados de 30 a 60 minutos 16 sensible y sencilla, recomendada para manejar en pacientes asintomáticos 17 así como en un abundante número de muestras, dirigido al diagnóstico en el punto de atención (POCT) 18,19, así como en el seguimiento a pacientes sospechosos, contactos cercanos y grupos de alto riesgo 20.
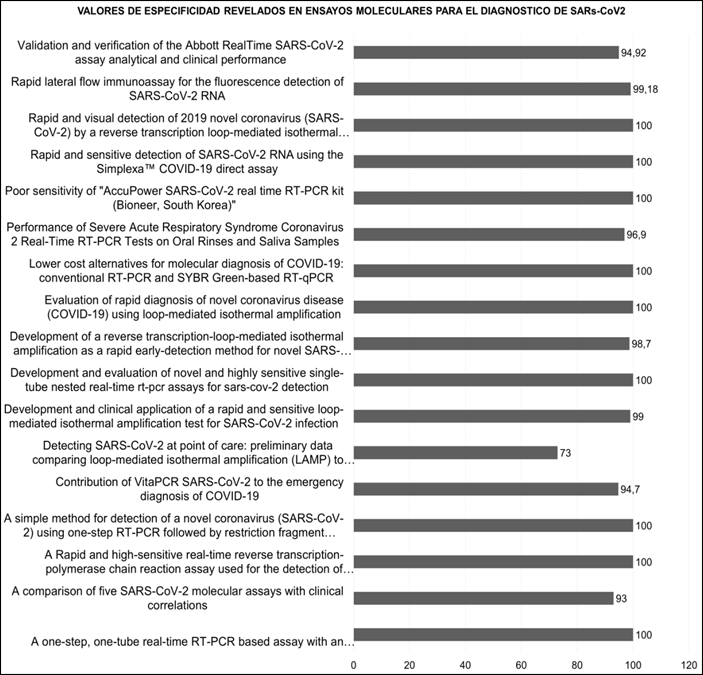
Finalmente, cinco ensayos validaron el rendimiento de kits que usan metodología RT-PCR 21)(22)(23. La sensibilidad y especificidad de los métodos diagnósticos moleculares se indican en la Figura 2 y Figura 3.

Figura 2 Sensibilidad en los ensayos moleculares. Se indica el ensayo molecular junto a su valor de Sensibilidad en porcentaje (%); Dos estudios RT-PCR muestran valores entre el 60 y 70%.
Diagnóstico serológico
Los estudios para la determinación y cuantificación de anticuerpos a través de técnicas serológicas se encontraron en menor proporción, en 8 de los 25 artículos revisados; los autores emplearon esta metodología dada la necesidad de procesos que revelen resultados en menor tiempo y empleando muestras menos invasivas y menos dolorosas, sin embargo al no existir el “Gold estándar” se debió comparar con ensayos internos, así lo hizo Pickering et al., 24. Por su parte, Sapkal et al., considerando la microneutralización del virus como un método de referencia para los ensayos serológicos desarrolló un ELISA (Enzimoinmunoanàlisis de adsorción) para la detección de IgG en suero o plasma empleando el antígeno del virus, revelando un importante rendimiento de sensibilidad y especificidad, además de ser recomendado para una valoración rápida en individuos expuestos como trabajadores de la salud o de la industria 25. Fue además el único de los ensayos en indicar valores pronósticos positivos y valores pronósticos negativos.
Entre las técnicas más empleadas y validadas por los autores se encuentra el ELISA, que tras su estandarización 26 se recomendó su uso tanto en la forma manual como semiautomática; 6 estudios validaron clínicamente la técnica sola o comparada con otras metodologías entre ellas electroquimioluminiscencia (ECLIA) quimioluminiscencia indirecta (CLIA) donde resultaron siempre con un mejor rendimiento 27. Los valores de sensibilidad y especificidad se indican en la Tabla 3.
Tabla 3 Sensibilidad y especificidad para ensayos serológicos. S= Sensibilidad; E= Especificidad; Cada estudio muestra valores de S y E heterogéneos e independientes del blanco de detección y de la técnica, solo uno de los ensayos valoró la detección de IgE, recomendada únicamente en fases iniciales de la infección.
| Título del artículo | S | E | S | E | S | E | S | E |
|---|---|---|---|---|---|---|---|---|
| Ig G | Ig M | Ig A | Anticuerpos totales | |||||
| Clinical Performance of A Semi-Quantitative Assay for SARS-CoV2 IgG and SARS-CoV2 IgM Antibodies | 94,7 | 100 | 97,4 | 100 | ||||
| Comparative assessment of multiple COVID-19 serological technologies supports continued evaluation of point-of-care lateral flow assays in hospital and community healthcare settings | 95 | 100 | 95 | 100 | ||||
| Comparison of commercial lateral flow immunoassays and ELISA for SARSCoV-2 antibody detection | 81,5 | 100 | 93,1 | 81,2 | ||||
| Comparison of commercial lateral flow immunoassays and ELISA for SARSCoV-2 antibody detection | 60 | 64 | ||||||
| Development and clinical application of a rapid SARS-CoV-2 antibody test strip: A multi-center assessment across China | 92,9 | 98,7 | ||||||
| Development of indigenous IgG ELISA for the detection of anti-SARS-CoV-2 IgG | 92,37 | 97,9 | ||||||
| Highly Sensitive and Specific Multiplex Antibody Assays To Quantify Immunoglobulins M, A, and G against SARS-CoV-2 Antigens | 95,78 | 100 | ||||||
| SARS-CoV-2 infection serology validation of different methods: usefulness of IgA in the early phase of infection. | 99 | 100 | ||||||
Liao et al., desarrolló un ensayo de inmunocromatografìa con base en oro coloidal que resalta por su rapidez en obtención de resultados, aunque no diferencia entre inmunoglobulinas, pero podrá emplearse en el monitoreo constante a personas con exposición perenne al virus como es el caso del personal médico 28. Ensayos cuantitativos basados en matriz de suspensión cuantitativa (qSAT) fue también una alternativa empleada por Dobaño et al., mostrando incluso mejor rendimiento y en menor tiempo que los ensayos ELISA 29.
Espécimen para diagnóstico molecular y serológico
Todos los estudios de diagnóstico molecular emplearon hisopado nasofaríngeo como muestra de análisis, 6 hicieron dos combinaciones de hisopado nasofaríngeo con esputo, hisopado nasal y lavado bronco alveolar respectivamente, mientras que 3 estudios apuntaron a mayores combinaciones como hisopado nasofaríngeo con frotis de garganta, saliva y enjuagues orales. Ver figura 3.
La práctica invasiva de toma de muestra a través del hisopado nasofaríngeo se podrá reemplazar por el uso de saliva con el kit empleado por Babady et al. Pues el índice de detección del virus fue igual al comparar estos dos tipos de muestras; no así usando enjuagues orales, ya que el mismo estudio revelo un bajo rendimiento del mismo 30. El kit SimplexaTM COVID-19 Direct, permite usar lavado bronco alveolar recomendado a pacientes con afección del tracto respiratorio inferior 22.
Genes y proteínas virales blanco de detección
El diagnóstico molecular se basa en la detección de zonas altamente conservadas del genoma viral, que tras su secuenciación permitió diseñar sondas y cebadores específicos que apuntan a la complementariedad del virus y su detección. Ver Figura 4. El ensayo de Chen et al. demuestra que la especificidad mejora al usar tres blancos de detección en comparación de uno solo 31. Por otra parte, entre los ensayos serológicos, de los ocho estudios uno no especifica el blanco de detección, mientras que otro usa antígeno viral, 1 estudio empleó solo un blanco de detección, la glicoproteína N, los demás emplearon combinaciones de los componentes proteicos del virus, Ver Figura 5.

Figura 4 Tipo de muestra y blanco de detección en ensayos moleculares. La coloración azul y verde intensa representa el uso de una sola muestra o un solo gen respectivamente; las coloraciones tenues indican combinaciones. Los 17 estudios emplearon ya sea la detección de un solo gen o combinaciones, entre éstos: gen ORF 1ab (open reading frame, marco de lectura abierta), gen RdRp (RNA polimerasa dependiente de ARN) gen N (nucleocápside), y gen E (envoltura). 11 de los estudios revisados hizo combinación de dos y hasta tres genes; 6 estudios emplearon un solo gen.
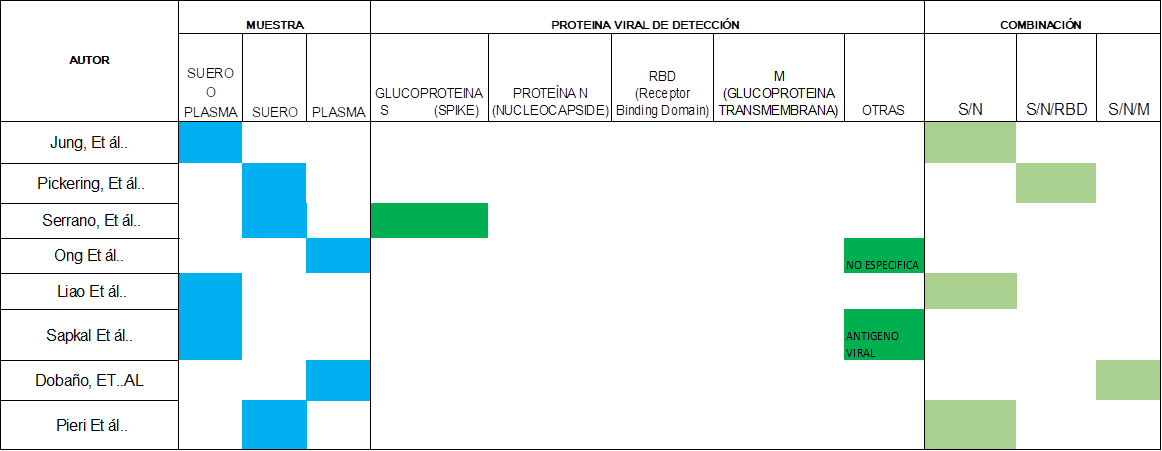
Figura 5 Tipo de muestra y blanco de detección en ensayos serológicos. Los 8 estudios serológicos emplearon muestras de sangre, 3 de ellos usaron suero y plasma sin mostrar variación en los resultados, por lo que no se consideraría un factor decisivo al momento de escoger una prueba de detección, no así en las pruebas moleculares. Dos estudios apuntaron a la detección de la glicoproteína spike (S) y de la envoltura (E), otros adicionaron la detección del receptor de unión RBD más la glicoproteína transmembrana (M). Todos los estudios apuntaron a la detección de IgM, IgG, o Ig A según el protocolo o etapa de la enfermedad.
Pruebas de reacción cruzada
En el proceso de desarrollar un diagnóstico a través de técnicas moleculares o serológicas se requiere estricta atención para evitar contaminantes de otros virus de las vías respiratorias, así como de virus de la misma familia Coronaviridae, pues serían los principales causantes de una falsa reactividad, solo dos de los 25 ensayos no realizó pruebas de reacción cruzada frente a otros virus lo que disminuiría la validez de los valores de rendimiento que notificaron, así se indica en la validación de la sensibilidad del kit “AccuPower SARS-CoV-2 real time RT-PCR (Bionner, South Korea) que no usa genes de controles negativos 32; también lo hace la prueba serológica desarrollada por Yunbao et al., 33 que no especifica en ningún punto de su estudio valores de especificidad o uso de controles negativos.
DISCUSIÓN
La identificación temprana de casos COVID-19 ayudó en primera instancia a establecer medidas de control, aislamiento, y tratamiento oportuno, la detección de anticuerpos de pacientes afectados por el virus y su cuantificación permite emplear el suero como medida de tratamiento de emergencia mientras se llevaba el proceso de desarrollo de vacuna y tratamiento farmacológico, además, la aparición abrupta y desconocida del virus y la variabilidad de síntomas no permitió hacer un diagnóstico clínico, siendo el diagnóstico de laboratorio la herramienta pilar para el manejo de la pandemia. En este punto de la enfermedad su valor no pierde significado, continuará siendo una herramienta importante pues tras el proceso de vacunación puede monitorearse la inmunidad alcanzada.
La identificación del virus empleando cebadores dirigidos a varias regiones del virus es la clave en las pruebas moleculares, Chen et al., 31 en donde se usó una combinación de hasta tres genes dianas que a pesar de no indicar el porqué de estas combinaciones podría relacionarse con el perfeccionamiento del diseño de cebadores pudiendo emplearse cualquiera de ellos por la confiabilidad que mostraron sus resultados de sensibilidad y especificidad; de la misma forma el bajo rendimiento de las pruebas puede deberse a fallas en el diseño del primer empleado.
Los protocolos moleculares han sido desarrollados en laboratorios independientes y aunque se encuentran bajo la aprobación de entidades reguladoras por la emergencia, estos no han demostrado un rendimiento adecuado, el estudio llevado por Procop et al., 23 en el que compara cinco kits, todos avalados por la FDA (Food and Drug Administration), el Kit ID Now COVID-19 (Abbott) mostro el más bajo rendimiento en términos de sensibilidad y especificidad por lo que tendrá un menor desempeño en muestras con carga viral baja. Aunque Yan et al., 22 realizó la validación del mismo kit mostrando un buen rendimiento la población de estudio fue solo de 30 individuos. Bordi et al., sugiere que debería haber una mayor ordenación antes de liberar al mercado kits con bajo sensibilidad por debajo del 70% como es el caso del kit AccuPower SARS-CoV-2 real time RT-PCR kit (Bioneer, South Korea) 32.
Además de la convencional RT-PCR la técnica más nombrada y con mayor cantidad de ensayos es la amplificación de ácidos nucleicos por LAMP, recomendada para su uso en un futuro en lugares de alta afluencia de público como farmacias, aeropuertos, centros de atención alejados, es decir en el punto de atención; sin embargo, debido a las mutaciones que pueden generarse en los genes diana de detección, la precisión del ensayo podría verse afectado.
El diagnostico a través de técnicas moleculares puede ser realizado en cualquier etapa de la enfermedad 23, no así en las pruebas serológicas de las cuales se probaron en grupos de pacientes y su rendimiento fue según los días de la aparición de los síntomas, así como de acuerdo a los días de afección 34.
Las validaciones de técnicas serológicas por su parte mostraron resultados bastante heterogéneos, de ahí la importancia de emplearlas sólo de acuerdo a la etapa de la enfermedad. Así para la determinación de cada inmunoglobulina (M, G, A) es necesario conocer el estadio de la enfermedad del individuo, así lo demostraron los estudios revisados pues todos emplearon grupos de pacientes clasificados por día de afección.
CONCLUSIÓN
n el análisis expuesto, se encontró una mayor cantidad de ensayos con un importante contenido sobre la validación y especificación de ensayos moleculares, mostrando adecuados valores de sensibilidad y especificidad. Las pruebas moleculares pueden ser usadas en varios especímenes biológicos como el hisopado nasofaríngeo, pero además en esputo, hisopado nasal y oro faríngeo, frotis de saliva, lavado bronco alveolar y enjugues bucales lo que reducirían las molestias en el paciente al momento de la toma de muestra. Los valores elevados especificidad son producto del uso de genes diana dirigidos a zonas altamente específicas del virus.
Las pruebas moleculares pueden ser aplicada en cualquier etapa de la enfermedad, y aun que requiere equipamiento y/o reactivos especiales, pueden usarse tanto en el ambiente intrahospitalario como en el punto de atención.
Por su parte, existe una minoría en ensayos de validación de técnicas serológicas las cuales revelaron valores de sensibilidad menores al 90%. La falta de una prueba Gold estándar, impide que sean utilizadas como único método de diagnóstico. Los valores de especificidad mostraron un rendimiento apropiado. Así también, el tipo de muestra necesaria para su aplicación es fácil de obtenerla y segura para el laboratorista encargado.
Por su parte, las entidades diagnósticas deberán reconocer el uso combinado tanto de pruebas moleculares y complementarlas con las serológicas de acuerdo a la información proporcionada por el paciente en relación al tiempo de infección o contacto cercano con un caso positivo.
Finalmente al momento de escoger una u otra técnica será necesario verificar además de su regulación, la validación clínica pues esto garantizará el uso de la misma.