INTRODUCCIÓN
La biósfera es una porción del planeta donde viven los seres vivos (Vernadsky, 1997). Mientras que los componentes físicos, químicos y biológicos, relacionados recíprocamente, ha sido denominado en conjunto como ecosistema, según los biólogos. Asimismo, el ecosistema se rige por sus leyes como la biodiversidad, fundamental para la supervivencia de las especies, equilibrio y vida. La interrelación de suelo, planta, atmósfera en la biósfera promueven el desarrollo de las plantas, generando además éstas un 20 % más (oxígeno, nitrógeno (N₂), argón (Ar), dióxido de carbono (CO₂) y vapor de agua (H₂O) desde hace unos 400 millones de años (Reichardt y Timm, 2020; Chang y Goldsby, 2013).
Las especies vegetales que fijan N₂ pertenecen a la familia Fabaceae (Leguminosae) compuesta por casi 20.000 especies (Llamas y Acedo, 2016), que incluyen algunos cultivos importantes, como también (hierbas, arbustos y árboles) (Gómez et al., 2002). El origen de las leguminosas se remonta a unos 59 millones de años (Sprent y James, 2007), siendo reconocidos actualmente 750 géneros y tres subfamilias (Caesalpinioideae, Mimosoideae y Papilionoideae) (Llamas y Acedo, 2016; Gómez et al., 2002), relevante por ser un grupo grande que varía, desde plantas perennes hasta agrícolas (Burns y Hardy, 1975). También, según Burns y Hardy (1975) la fijación de N₂ ocurre en 90 % de las Mimosoideae y Papilionatae y en una tercera parte de la Caesalpinioideae; además están distribuidas en los trópicos y valles, asimismo las 100-200 leguminosas más importantes están dentro de las Papalionatae como la soya (Glycine max (L.) Merr.), maní (Arachis hypogaea L.), lenteja (Lens culinaris Medik.), guisante (Pisum sativum L.), frijol (Phaseolus vulgaris L.), alfalfa (Medicago sativa L.) y trébol (Trifolium repens L.). Por otra parte, las leguminosas a través del proceso de fijación simbiótica conjuntamente las bacterias del género Rhizobium aportan N₂ que beneficia la producción de pastos y forrajes para los ganados (Tang, 1986).
El N₂ y el agua (H₂O) son considerados factores limitantes del rendimiento en la agricultura. El N₂ gaseoso de la biósfera no es asimilado por las plantas, pero sí por las procariotas (Eubacteria y Archaeas) que poseen el complejo enzimático nitrogenasa que transforma el N₂ a NH₄ (Moat et al., 2002; Dixon y Kahn, 2004; Madigan et al., 2015). La fijación biológica de nitrógeno (FBN) por Rhizobium-leguminosa, es el sistema con mayor captación de N₂ por la cantidad de N₂ que fija y por la diversidad de entornos donde ocurre según García et al. (2021). Los nutrientes disponibles del suelo condicionan el crecimiento de las plantas como el caso del N₂, que incide en la productividad de los cultivos (Calvo, 2011), pero, para García y Picone (2021) el fósforo (P) tiene también un efecto en el desarrollo de las plantas. Por otra parte, se conocen simbiosis entre 14 géneros de angiospermas en 8 familias y 7 órdenes; 1/3 de 300 especies están noduladas y son fijadoras de N₂, pero el reporte real podría ser mayor ya que varias especies no se estudiaron. La taxonomía de géneros de los angiospermas indica una relación filogenética (Burns y Hardy (1975). También, indica que puede ser temporal como resultado de la incidencia por simbiosis. Los simbiontes que fijan N₂ en nódulos de las raíces de las angiospermas (no leguminosas) están distribuidas a nivel mundial y en lugares donde la vegetación no se estableció aún. Algunos géneros (Alnus, Coriaria, Elaeagnus e Hippophae), se encuentran en todo el mundo, mientras que otros (Ceanothus y Shepherdia) tienen una repartición restringida (Burns y Hardy, 1975).
El nitrógeno es un nutriente primario crítico para la supervivencia de todos los organismos vivos; también forma parte de biomoléculas, proteínas, ADN y clorofila. Cuando el N₂ se convierte en amoníaco (NH₃), está disponible para la producción primaria mediante la simbiosis como en el caso de las plantas.
Además, existe el N₂ inorgánico (amoniaco (NH₃) y nitrato (NO₃⁻)) y orgánico (Amino (-NH2) y ácidos nucléicos) (Bernhard, 2010). Presente en la naturaleza con 4/5 partes de aire y combinado (ácidos nucleicos, aminoglúcidos, urea (CH₄N₂O), poliaminas, nitratos, nitritos (NO₂⁻), vitaminas, proteínas (animal y vegetal); responsables de la disponibilidad de energía de adenosíntrifosfato (ATP) y trifosfato de guanosina (GTP) y del potencial reductor nicotinamida adenina dinucleótido fosfato (NAD(P⁺) y dinucleótido de flavina y adenina (FAD) (Calvo, 2011; Macías et al., 2018; NIH, 2021). Por lo tanto, el N₂ experimenta diferentes transformaciones como la fijación de N₂, la nitrificación, la desnitrificación, la oxidación anaeróbica del ion amonio (anammox) y la amonificación en el ecosistema, donde los organismos lo utilizan para el crecimiento y en algunos casos como energía. Por otra parte, el N₂ en sus estados de oxidación es vital para la productividad en la biósfera y depende de las actividades de un conjunto diverso de microorganismos (bacterias, arqueas y hongos) (Bernhard, 2010; Calvo, 2011). Tecnologías recientes en metagenómica a través de la secuenciación de nueva generación (NGS), proporcionan un enfoque para cuantificar taxones en el
microbioma del suelo (Simón y Daniel, 2011), que permite detectar y cuantificar directamente las secuencias de ADN evitando además aislar los organismos que pueden ser raros en proporción o ser exigentes e incapaces de cultivar. La secuenciación metagenómica de escopeta (Shotgun) evita el sesgo de amplificación por reacción en cadena de la polimerasa (PCR) y proporciona un registro funcional a través del análisis genético rico y el análisis de ruta (Sharpton, 2014). Este artículo de revisión estudia la fijación biológica del nitrógeno a través de la interacción y simbiosis de bacterias-plantas.
METODOLOGÍA
Se realizó una revisión de libros, artículos, conferencias publicadas y tesis accesibles en internet y base de datos de la Web of Science, Scopus y Google Academic aplicando palabras clave como bacteria, fijación del N₂, leguminosas, metagenómica y simbiosis.
DESARROLLO Y ANÁLISIS DE BACTERIAS Y PLANTAS EN LA FIJACIÓN DEL NITRÓGENO
Suelo y organismos
En el siglo XX la humanidad ha experimentado avances científicos y tecnológicos. Las plantas, los animales y los microorganismos que viven en un área determinada, constituyen una comunidad biológica interconectada por una red compleja de relaciones funcionales y su entorno. El suelo, es un sistema de interacción entre las fases sólida, (constituida por materia mineral y orgánica), líquida y gaseosa o atmosférica del suelo. La materia orgánica procede de la actividad de los organismos del suelo, variable en composición y cantidad, principalmente en función del tipo de cubierta vegetal. Los microorganismos desarrollan procesos de edafogénesis. Asimismo, los ciclos biogeoquímicos en base al carbono (C), nitrógeno (N₂), oxígeno (O), azufre (S), fósforo (P), hierro (Fe) y otros metales son relevantes; por otra parte, es trascendental la fertilidad de las plantas y protección frente a patógenos y la degradación de compuestos xenobióticos. Los organismos del suelo siguen patrones espaciales de agregación a escalas (nm a km) que se superponen, debido a factores de control (dinámico y equilibrado). Sin embargo, con técnicas de observación de secciones ultrafinas de suelo mediante microscopía electrónica, tomografía, análisis geoestadístico y la elaboración de modelos, especialmente basados en fractales, se demostró que la distribución de las bacterias edáficas está altamente estructurada, vital para el funcionamiento del suelo. Las bacterias se organizan en microcolonias compuestas de pocas células que pueden pertenecer a diferentes morfotipos. Factores como la presencia de raíces, pequeños agregados, nutrientes y poros condicionan la distribución de bacterias en los microhábitats. La complejidad del suelo como ecosistema a nivel microscópico y características de los microorganismos (tamaño, morfología) proporcionan una visión de la naturaleza microbiana edáfica con función desconocida. Asimismo, estudiaron la biomasa microbiana para determinar enzimas, constituyendo un acercamiento a la actividad y abundancia microbiana, y a los flujos de intercambio (atmósfera y el subsuelo) de compuestos que permitieron el análisis de procesos (respiración) (Nogales, 2005; Reichardt y Timm, 2020).
El suelo es un ambiente complejo en el cual los seres vivos desempeñan roles importantes para el mantenimiento de la fertilidad; es el hábitat en el cual existen eucariotas y procariotas, los cuales interactúan y contribuyen en las características del suelo. Últimamente, las técnicas de biología molecular han tenido un avance en el conocimiento de la diversidad de microorganismos de los diferentes ecosistemas en el campo de la microbiología del suelo (Nogales, 2005). Sin embargo, en este contexto, cualquier suelo puede ser considerado como extremo para los microbios colonizadores que constantemente enfrentan inanición, desecación, depredación y otros ataques (Dion y Chandra, 2008). El suelo es un depósito de diversos microorganismos, que con frecuencia se ha utilizado para aislar y explotar microbios para uso industrial, ambiental y aplicaciones agrícolas; el conocimiento sobre la estructura y la dinámica de las comunidades bacterianas en el suelo es condicionado porque solo una pequeña fracción de la diversidad bacteriana accede a los métodos de cultivo (Dubey et al., 2006; Suárez, 2010).
Importancia del ciclo del nitrógeno
Las contribuciones humanas al ciclo global del N₂ a través de la movilización de N₂ fijado de la atmósfera y su deposición en ecosistemas naturales son relevantes (Galloway et al., 1995; Vitousek et al., 1997). Los organismos (bacterias) simbióticas como parte del ciclo del N₂ permiten fijar el N₂, y componer NO₃⁻ y NH₄; asimismo los animales eliminan (amoniaco, urea o ácido úrico) que son intervenidas por bacterias denitrificantes que retornan el N₂ inorgánico de los suelos a la atmósfera (Calvo, 2011). Por otra parte, es difícil controlar todos los factores externos y los agentes involucrados en el ciclo del N₂, ya que suele ser eventual como cualquier proceso biogeoquímico, por lo que la acción de los microrganismos fijadores de N₂ puede presentarse solo cuando tienen suficiente humedad y elementos minerales (Fonseca-López et al., 2020). Asimismo, depende de la actividad de una amplia variedad de organismos, donde cada uno de los cuales posee conocimiento adquirido evolutivamente incrustado en su material genético (Gorman, 2013). Los compuestos e iones inorgánicos más significativos en la naturaleza son: gas N₂, ácido nítrico (HNO₃), ion nitrato (NO₃⁻), ion nitrito (NO₂⁻), óxido nitroso (N₂O), óxido nítrico (NO), dióxido de nitrógeno (NO₂), ion amonio (NH₄⁺), gas amoniaco (NH₃) y las formas inestables dimida (N₂H₂) e hidrazina (N₂H₄). Dentro de los compuestos orgánicos el N₂ está asociado con C formando biomoléculas (amidas, bases púricas, pirimídicas, aminoácidos, proteínas, ácidos nucleicos y otros metabolitos) (Luengas, 2014). Sin embargo, el ciclo del N₂ en ecosistemas agrícolas podría ser afectado con aplicaciones excesivas de fertilizantes causando contaminación del suelo, aire y agua; como con aplicaciones deficientes contribuyendo a la degradación del suelo por disminución de las reservas naturales de N₂, fundamentalmente de N₂ orgánico (García y Daverede, 2007). Por lo tanto, es necesario el diagnóstico de las necesidades de N₂ de los cultivos para alcanzar el máximo rendimiento biológico y económico, a nivel especifico de zona y lote (García y Daverede, 2007).
La simbiosis en el crecimiento de plantas
Es complicado estudiar la vida e historia evolutiva sin la simbiosis que se refiere a la asociación permanente de dos o más organismos (especies) diferentes donde pueden o no beneficiarse ambos, esto ocurre en la naturaleza en bacterias y hongos que viven junto a raíces de plantas terrestres; donde las bacterias son hospederos de virus por una parte y por otra los virus satélites dependen de otros virus para su expresión genética (Paracer y Ahmadjian, 2000). Existen varios tipos de simbiosis, ya sean beneficiosas o perjudiciales, se describen con los términos de comensalismo, mutualismo y parasitismo. Una asociación en la que un simbionte se beneficia y el otro no se ve afectado ni se beneficia es una simbiosis comensalista. Una asociación en la que se benefician ambos simbiontes es una simbiosis mutualista. Una relación en la que un simbionte recibe nutrientes a expensas de un organismo huésped es una simbiosis parasitaria (Paracer y Ahmadjian, 2000). Los diazótrofos varían en complejidad de la bacteria primitiva, Clostridium, a través de simbiosis fisiológica y genéticamente enredadas de nódulos de raíces de leguminosas. Esto es mínimo en las asociaciones sueltas como entre microorganismos de vida libre, pero se intensifica a medida que participan formas de plantas superiores y culmina en el sistema de nódulos de raíces altamente complejas (Burns y Hardy, 1975).
Por tanto, en este contexto se hace necesario comprender como funciona (directo e indirecto) la promoción del crecimiento de las plantas a través de las rizobacterias (PGPR) (Figura 1). En el método directo usa nutrientes y reguladores del crecimiento para promover el crecimiento de plantas (PGP) y en el método indirecto dependen de diferentes factores (estrés biótico y abiótico) (Meena et al., 2017). El análisis del genoma y genes (identificados) que contribuyen a la actividad beneficiosa de PGPR facilitan comprender los mecanismos moleculares de las bacterias y también ayudan a la creación de nuevas tecnologías de fitorremediación por PGPR (Meena et al., 2017).
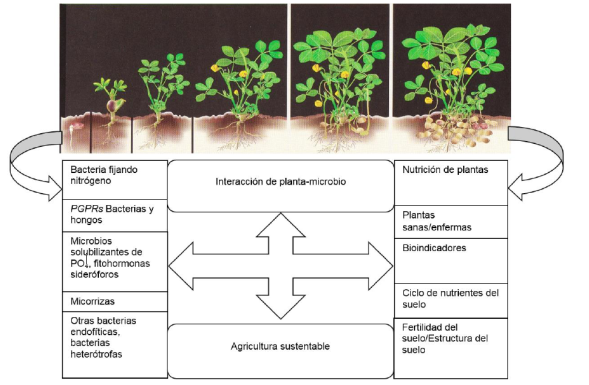
Figura 1 Diagrama esquemático de las interacciones microbianas de las plantas en la rizosfera modificado de Meena et al. (2017). Imagen, planta de maní (Arachis hypogaea L.) (Laserna, 2008).
Las plantas pueden interactuar con una diversidad de microorganismos. Se considera que la capacidad de las primeras plantas terrestres para formar relaciones simbióticas con los hongos terrestres ha sido uno de los factores críticos para la colonización de la tierra, un evento crucial para el surgimiento de ecosistemas terrestres (Delaux et al., 2015). Las bacterias endofíticas a menudo promueven el crecimiento de las plantas y protegen a su planta (huésped) contra patógenos, herbívoros y factores ambientales que producen estreses abióticos, tales como la sequía, el aumento de la salinidad o la contaminación (Papik et al., 2020). Por otra parte, según Coleman-Derr et al. (2015) son desconocidas las potencialidades de las comunidades bacterianas que soportan estrés en el desierto y que viven en simbiosis con sus hospederos (plantas) sobrevivientes.
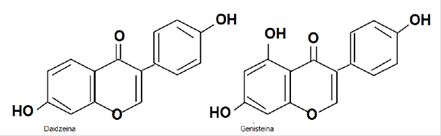
En la primera fase, el establecimiento de la simbiosis es la liberación de productos fotosintéticos por parte de las plantas conjuntamente con un proceso de reconocimiento celular por parte de la bacteria. Las plantas liberan materia orgánica (carbohidratos, ácidos orgánicos, vitaminas, aminoácidos o compuestos fenólicos). Los flavonoides (quimioatractantes) están dentro los compuestos fenólicos. Las plantas exudan flavonoides detectados por los productos de los genes nodD de las bacterias. Los flavonoides (bioflavonoides, vitamina P y citrina), son metabolitos secundarios de las plantas. Sintetizados de una molécula (fenilalanina) y tres moléculas (malonil-CoA). La estructura (C6-C3-C6) se cicla debido al enzima isomerasa. El eriodictiol y la apigenina-7-O-glucósido inducen la nodulación de la agrobacteria Rhizobium leguminosarum, como también la daidzeina y genisteína (Figura 2) estimulan los genes de la nodulación de varias cepas de Bradyrhizobium japonicum. Por ejemplo la daidzeina es exudada por la soya, como también exudan las plantas (aminoácidos y ácidos dicarboxílicos) y atraen nutrientes (Calvo, 2011).La bacteria actúa como receptor de estas señales y busca sitios de invasión en la raíz de la planta emisora (Calvo, 2011).
En la segunda fase, la bacteria de adhiere a su hospedero, por su parte la planta libera (glicoproteínas) específicas (ricadesina y lectinas), para atraer bacterias. Éste cerca de la raíz libera lipopolisacarídico como factor NOD que produce deformaciones en los pelos radiculares. Posteriormente la planta emite proteínas que facilitan la expresión de genes de fijación como genes FIX de las bacterias. Éstos provocan que las bacterias reconozcan los lugares (pelo radicular) donde pueda ingresar a la planta. La invaginación en la membrana de las bacterias es fundamental para su colonización. En la tercera fase, se estable el simbiosoma (simbiosis diatrofa) (Calvo, 2011).
Descripción histórica de la interacción de plantas y bacterias en la fijación del nitrógeno
A partir de los avances logrados por Hellriegel y Willfarth en 1888 en primera instancia, año en que Beijerinck aisló la bacteria del nódulo y la denomino Rhizobium radicicola que luego la cambio a R. leguminosarum. Por su parte, Nobbe en 1892 recomendó fijar N₂ con una no-leguminosa nodulada Elaeagnus angustifolia y también por microorganismos de vida libre que fue establecida por la demostración de Winogradsky en 1893 de que Clostridium pasteurianum podía fijar N₂ lo cual posteriormente no pudo ser sostenida por la investigación de esa época. Los escasos ensayos y cultivos puros dificultaban la fijación de N₂ y así poder establecer con certeza los agentes fijadores de N₂. Además, Willson en 1952 señaló que “la naturaleza no pudo proporcionar una señal brillante comparable a la clorofila para marcar los fijadores de N₂” (Burns y Hardy, 1975). Existen dos tipos de fijación del N₂: abiótica (forma óxidos) o biológica (a través de bacterias). Definida también como la oxidación o reducción del N₂ para dar iones amonio (NH₄⁺), nitrito (NO₂⁻), nitrato (NO₃⁻) y óxidos (NO), (NO₂). El (NO₂) reacciona fácilmente para originar algunas de las anteriores. Una vez convertido el N₂ a formas metabolizables estas pueden ser incorporadas por los seres vivos (Calvo, 2011).
La FBN se realiza en los nódulos radiculares, cuyo resultado es la relación de simbiosis entre las plantas y las bacterias. Durante la simbiosis se forma un bacteroide que expresa su actividad nitrogenasa que con el complejo nitrogenasa fija el N₂. La formación de nódulos radiculares se realiza por etapas entre las plantas y las bacterias (Calvo, 2011). En la Figura 3 podemos ver los microhábitats en el interfaz suelo-planta (Reinhold-Hurek et al., 2015).
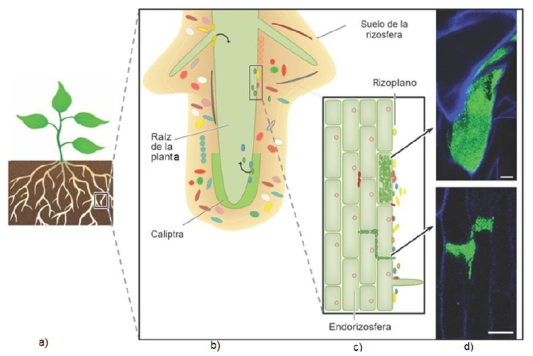
Figura 3 a) Microhábitats en la interfaz planta-suelo. b) y c) primeros planos de la raíz con el suelo de la rizosfera circundante y de los compartimentos del rizoplano y la endorizosfera; d) micrografías de escaneo láser confocal que muestran una densa colonización intracelular (arriba) e intercelular (abajo) de las raíces por bacterias endofíticas. Micrografías tomadas de un cultivo hidropónico gnotobiótico de Oryza sativa inoculado con una cepa informadora etiquetada con gfp de Azoarcus sp., cepa BH72. Las barras de escala representan 15 μm. Modificado de Dommergues y Krupa (1978); Reinhold-Hurek y Hurek (1998); Reinhold-Hurek et al. (2015).
La rizosfera es un interfaz crítico que apoya el intercambio de recursos entre las plantas y su entorno asociado al suelo. La diversidad microbiana de la rizosfera es influida por las propiedades físicas y químicas de ésta zona, algunas de las cuales están determinadas por la genética de la planta huésped (Peiffer et al., 2013).
Del suelo a los hábitats radiculares: modelo de enriquecimiento en tres pasos (Figura 4). Una síntesis de estudios basados en comunidades microbianas y evidencia genética, sugiere un mecanismo de enriquecimiento de tres pasos para comunidades microbianas en asociación con raíces. Este mecanismo conduce gradualmente a una menor diversidad y a un mayor grado de especialización hacia el interior de la raíz. Además de un modelo de enriquecimiento de dos pasos propuesto por Coutinho et al. (2015), en la que considera hábitats de diferentes raíces y funciones bacterianas (Figura 4) (Reinhold-Hurek et al., 2015). Por otra parte, el suelo proporciona un conjunto de microbios muy diverso que se basa en factores edáficos y ambientales, y probablemente se ve afectado por la historia de la vegetación. A medida que se reclutan colonizadores de raíces de este reservorio, su efecto sobre el microbioma asociado a las raíces es alto, aunque algunos endófitos pueden transmitirse verticalmente desde las semillas (Reinhold-Hurek et al., 2015).
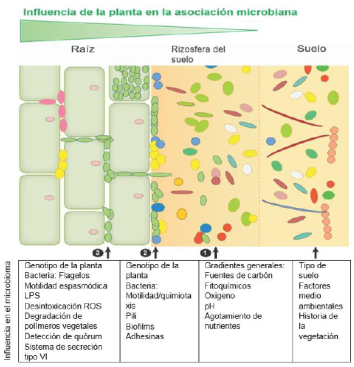
Figura 4 Un modelo de enriquecimiento de tres pasos para microorganismos que colonizan diferentes microhábitats de raíces. En el suelo a granel, las comunidades microbianas dependen del tipo de suelo, los factores ambientales y la historia de la vegetación. (1) Hacia la raíz, los gradientes de suelo de la rizosfera impuestos por la raíz (como las fuentes de carbono liberadas y los fitoquímicos, los gradientes de oxígeno y pH y el agotamiento de los nutrientes) afectan la composición de la comunidad, y el genotipo de la planta tiene una influencia relativamente pequeña. (2) Una comunidad más especializada se enriquece en el rizoplano bajo la influencia de factores específicos del genotipo de la planta. (3) La endorizosfera está habitada por una comunidad de menor complejidad. Se requieren rasgos bacterianos específicos para la entrada y el establecimiento dentro de este compartimento, y es probable que el genotipo del huésped tenga la mayor influencia en la estructura de la comunidad aquí en comparación con otros compartimentos. Abreviaturas: LPS, lipopolisacárido; ROS, especies reactivas de oxígeno (Reinhold-Hurek et al., 2015).
Reacción química de la fijación del nitrógeno
La fijación del N₂ por las asociaciones Rhizobium-leguminosas es una fuente económica, ecológicamente útil en el suministro de este elemento. El estudio de la FBN busca aumentar la tasa y la eficiencia de la fijación para incrementar la proteína/área, disminuir el consumo de fertilizantes nitrogenados y evitar la disminución del contenido de N₂ de suelos arables (García et al., 2021). En la fijación abiótica Fritz Haber obtuvo dos moléculas de amoniaco combinando una molécula de N₂ y tres de H₂. Esta reacción se realiza a 500 ºC y a 200 atm. Asimismo, se tiene H₂ utilizando metano (CH₄). Según la reacción química: N₂ + 3H₂ → 2NH₃ que requiere un catalizador (níquel (Ni) (Calvo, 2011).
La segunda etapa del método de Haber-Bosch fue oxidar el NH₃ hasta ácido nítrico (HNO₃), que al combinarse de nuevo con NH₃ produce nitrato amónico (NH₄NO₃), este es el producto final y se utiliza como explosivo o fertilizante. Otro proceso abiótico de fijación de N₂ es la producción de cianamida (CH₂N₂), empleada para elaborar cianuro (CN−) y como fertilizante. Este proceso hace pasar el N₂ sobre cianuro de calcio (CaC₂N₂) caliente. Al igual que el método de Bosch, la elaboración de cianamida también necesita la presencia de un catalizador. Asimismo, parte del N₂ puede fijarse abióticamente mediante rayos o ciertos procesos industriales, incluida la combustión de combustibles fósiles (Bernhard, 2010; Calvo, 2011).
Por su parte, la FBN que considera a la comunidad de la rizosfera estructurada por la microbiota (bacterias, hongos y algas) y microfauna (protozoos, nematodos, insectos y ácaros), mediante la simbiosis fijan el N₂ y las plantas generan compuestos solubles como (NH₃) (Calvo, 2011). Luego el NH₃ (aminoácidos y proteínas) son parte de la cadena alimenticia. La FBN demanda de bastante energía y de enzimas nitrogenasas, según la siguiente ecuación química: N₂ + 8H⁺ + 8e⁻ + 16 ATP → 2NH₃ + H₂ + 16ADP + 16 Pi, (Bernhard, 2010; Calvo, 2011).
Como resultado, solo un grupo selecto de procariotas puede llevar a cabo este proceso energéticamente exigente. Aunque la mayor parte de la fijación de N₂ la llevan a cabo los procariotas (Bernhard, 2010; Calvo, 2011). Algunas bacterias utilizan sus enzimas para fijar el N₂ en el entorno microaerófilo (SB, 2021). La fijación simbiótica del N₂, equivalente a 120 Tg relativamente baja al asimilado de los minerales del suelo, pero representa el 8,5% del consumo total por la planta debido a la alta eficacia de la transferencia (Calvo, 2011).
Estudios de bacterias que fijan nitrógeno en diferentes ecosistemas
Las últimas contribuciones potenciales de la metagenómica al estudio de las rizobacterias promotoras del crecimiento de las plantas (PGPR) tienen una función importante en la agricultura sostenible. Actualmente, varios estudios se centran en dilucidar el mecanismo molecular implicado en las interacciones planta-microbio para comprender la funcionalidad de los PGPR (Menna et al., 2017). Sin embargo, partiendo del concepto de ecosistema, aquí por ejemplo los resultados logrados por Carbonetto et al. (2014) en las Pampas de Argentina, describen el impacto en la estructura y composición del microbioma (Tabla 1) donde los perfiles metagenómicos metabólicos fueron afectados.
Tabla 1 Impacto por prácticas agrícolas en diferentes Países.
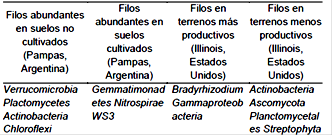
Fuente: Carbonetto et al. (2014) y Chang et al. (2017).
Complementar los datos metagenómicos con metatranscriptómica y metaproteómica mientras se emplea a la a la vez la metabolómica y se tiene en cuenta la información compleja sobre los factores bióticos y abióticos posiblemente ayudará a resolver varias incógnitas del endofitismo a futuro. Estas tecnologías avanzadas de “ómica” ayudan a comprender los mecanismos de selección de bacterias endofíticas inducidos por las plantas y descubrir la magnitud de las interacciones entre los factores que dan forma a las comunidades mencionadas y los ambientes, así como profundizar el conocimiento sobre la dinámica de la diversidad endofítica inexplorado. Además, las informaciones técnicas dependientes e independientes de cultivos revelará todo el potencial biotecnológico de la diversidad bacteriana endofítica (Papik et al., 2020). Con un conocimiento de la base genética del endofitismo y las interacciones multipartitas, se podrá manipular los microbiomas de las plantas para obtener los resultados buscados (mejor aptitud, mayor rendimiento, resistencia al estrés, respuesta a fitorremediación, inoculación) a través de endófitos beneficiosos (Papik et al., 2020).
En este contexto, por ejemplo, según Trinick y Hadobas (1989) Parasponia andersonii (Planch.) es la única no leguminosa que nodula con (Rhizobium/Bradyrhizobium). Con cualidades de fijar importantes cantidades de N₂; aspecto relevante para estudiar a nivel molecular desde una visión agronómica y a futuro replicar éstos atributos en otras no leguminosas. También encontró hemoglobina en el tejido del nódulo de Parasponia y otras no leguminosas noduladas; el gen que las origina y expresa está en plantas como Trema tomentosa y Celtis australis. Las cepas de Bradyrhizobium aisladas de especies de Parasponia que crecen en Papua Nueva Guinea no se nodulan eficazmente (excepto CP283). La eficacia de la simbiosis está definida por el hospedero, Bradyrhizobium y el medio ambiente. La simbiosis con P. andersonii es eficaz en relación a las otras estudiadas.
Coleman-Derr et al. (2015) afirmó que el perfil filogenético de las procariotas está determinado por el vegetal (hospedero) y en el caso de hongos (comunidades) incide la biogeografía. En el caso de Agave tequila cultivada, ésta muestra bajos niveles de procariotas en relación a especies nativas de Agave. Bacterias y hongos promueven el crecimiento de plantas y brindan tolerancia al estrés abiótico a partir de la interacción (Agave-microbio).
A su vez Chang et al. (2017) estudió el microbioma en Illinois en campos agronómicos de los Estados Unidos, donde la soya sobresale y su rotación de cultivos es el maíz. Los estudios (metagenoma) se detallan en la Tabla 1. Asimismo, se identificó cuatro grupos de
bacterias y dos grupos de eucariotas que se asociaron significativamente con la productividad de los cultivos. El uso de un modelo de bosque aleatorio (RF) predijo con éxito la productividad del cultivo con una precisión de 0.79. Con el progreso activo en la anotación de metagenomas, la comparación estadística y el poder computacional para manejar datos de alta dimensión, se espera que MWAS (asociación de todo el metagenoma) y el aprendizaje automático brinde una nueva comprensión sobre cómo las comunidades microbianas interactúan con los cultivos y brindan beneficios directos a la agricultura (Chang et al., 2017). Por tanto, se utilizan los genomas comunitarios aleatorios (Metagenomas) para estudiar microbios en diferentes entornos a través de análisis de secuencias.
La secuenciación de nueva generación de alto rendimiento y a bajo costo ha proporcionado acceso a la metagenómica a una amplia gama de investigadores (Meyer et al., 2008; Carbonetto et al., 2014).
Distribución de bacterias en leguminosas
Según Wang et al. (2019) los microorganismos del suelo durante las actividades agrícolas son importantes debido a un estudio realizado en el Sur de los Estados Unidos en cultivares de maní (Arachis glabrata Benth.) se identificó filos según se detalla en la Figura 5, a partir de muestras de secuencias (equivalente de 1.800 amplicones del gen ARNr 16S seleccionados al azar.
En otro caso Chen et al. (2014) sostiene que la salud de las plantas y la fertilidad son afectadas por la interacción planta-microbioma. En el mencionado estudio que se realiza en cultivos de maní a través de clones de genes de ARNr 16S determinando así los cambios de las comunidades bacterianas del suelo en diferentes campañas agrícolas (Tabla 2).
En Veracruz-México, Valles y Cadisch (2010) examinaron cultivos realizados en invernadero donde observaron que la fijación de N2 depende de la cepa Rhizobium, siendo la más eficaz Bradyrhizobium CIAT 3101. Sin embargo los ecotipos Arachis pintoi CIAT 17434 y 18744 reportaron valores significativos en la fijación del N2 en relación a Arachis hypogaea (referente) e igual en crecimiento a Arachis pintoi.
Distribución de bacterias en gramíneas
Los exudados de las raíces del maíz (Zea mays L.), como azúcares, ácidos orgánicos, aromáticos y enzimas, interactúan con los parámetros del suelo, (pH, potencial hídrico, textura, nutrientes), y las poblaciones microbianas para PGP. Estudios realizados sobre la asociación entre el genotipo del maíz y la diversidad microbiana utilizando chips de ADN (microarrays) de genes de ARNr 16S o huellas genómicas en (invernadero y campo) apoyan la ponencia de que la diversidad microbiana está relacionada con el genotipo de la planta (Aira et al., 2010; Bouffaud et al., 2012).
En este contexto bajo el criterio de usos diversos de suelos y distribución variable de filo, ratifico como un sistema de modelo para investigar la fijación del N₂ en el maíz Sierra Mixe, validando la asociación con bacterias diazotróficas que poseen operones genéticos canónicos para la fijación de N₂ (Bennett et al., 2020). Según Higdon et al. (2020) es fundamental el mucílago de la raíz aérea para el fenotipo de fijación de N₂ del sistema; confirmaron la presencia de bacterias fijadoras de N₂ clásicas en la microbiota del mucílago (potencial genómico) que permiten derivar energía para la FBN a partir del polisacárido (mucílago). También, demostró que los diazótrofos incorporaron N₂ en su metabolismo a través de vías (metabólicas desconocidas), delimitadas por el actual conocimiento sobre la FBN como la conversión bacteriana de dinitrógeno (N₂) a (NH₃) a través de la expresión de productos del gen nif canónico dentro de los modelos de Bond y Klebsiella. Asimismo, lograron recuperar y caracterizar diazótrofos encontrando (diazótrofos) que no contenían ningún gen canónico nif, recomendando así el uso de genes novedosos para la conversión de N₂ en formas orgánicas que sean asimiladas en moléculas pequeñas exportadas por los organismos. Algunos miembros (específicos) del microbioma (diazotrofia) del maíz Sierra Mixe poseen variadas funciones moleculares.
Higdon et al. (2020) confirma que las bacterias diazotróficas aisladas de mucílagos de raíces aéreas poseen la capacidad de llevar a cabo múltiples métodos de PGP directa, además de la FBN que resultan en la promoción del crecimiento de cultivos no relacionados con el maíz Sierra Mixe. También identificó que la mayoría de los aislamientos diazotróficos poseen genes marcadores para la desaminasa del ácido 1-aminociclopropano-1-carboxílico (ACC), la producción del ácido indolacético (IAA) y la solubilización de PO₄ además de los genes nif, presentan evidencia de sus capacidades y funcionalidades in vitro a capacidades variables que se encontraron significativamente afectadas por el contenido genético nif. El uso de estos diazótrofos de mucílago como inoculantes para plantas de papa (Solanum tuberosum L.) y una variedad híbrida convencional de maíz también confirmó la capacidad de varios aislamientos para conferir fenotipos de PGP para cultivos fuera de su entorno nativo. Además dilucida la posibilidad de que los diazótrofos del mucílago funcionan como PGPB en un amplio rango de hospederos, como lo demuestra el descubrimiento informado de múltiples funcionalidades de PGP conferidas por Raoultella BCW-201900 (Brown e Irber, 2016; Higdon et al., 2020).
Según Martínez-Peña et al. (2016) la siembra de maíz en los Altos de Jalisco es para producción de forraje y autoconsumo, de razas hibridas o de zamorano amarillo. En esta región consideraron las características de las plantas y del suelo, porque no se conoce la composición de los microorganismos en este ambiente. Indico que el uso de secuenciación de nueva generación (NGS) para la descripción de un cultivo de maíz mixto para autoconsumo y otro para forraje en los Altos de Jalisco logro una descripción inicial de la composición microbiana de la rizosfera microbiota (Figura 6). Asimismo, detectó una alta diversidad de unidades taxonómicas operativas (OTUs) en tres variedades de maíces (autoconsumo y forraje), el cual se sostiene en (tipo de manejo agronómico y el tipo de maíces sembrados).
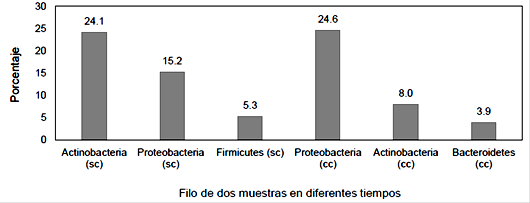
Figura 6 Filo de dos muestras en diferentes tiempos (sc = sin cultivo; cc = con cultivo) (Martínez-Peña et al., 2016).
En Bolivia, no se han realizado estudios metagenómicos similares, sin embargo, según Barrientos et al., (2017) la fijación simbiótica de N₂ suele ser una de las alternativas del manejo de los nutrimentos del suelo, especialmente cuando se utilizan leguminosas como el tarwi (Lupinus sp mutabilis Sweet) como abono verde según valido (como positivo) la Fundación PROINPA y como alternativa de rotación de cultivos recomiendan usar K'ela (Lupinus sp.) en la producción de la quinua (Chenopodium quinoa Willd.).
CONCLUSIONES
Los estudios confirman las interacciones de plantas y microorganismos, a través de la simbiosis, en diversos tipos de suelos, donde estos, coadyuvan al incremento de la producción de cultivos agrícolas a través de la FBN, en leguminosas y gramíneas como en el caso del maní y el maíz. La determinación de la distribución de filos, en diversos ecosistemas de producción, mediante el uso de herramientas (metagenómica, bioinformática), dilucida e identifica las diferencias genéticas de la diversidad microbiana, asimismo permite predecir sus funciones, como la fijación del N₂ y la promoción del crecimiento de plantas por bacterias de la rizosfera, información relevante para la agricultura sustentable. También, la distribución de las bacterias de los suelos, en los diferentes ecosistemas, son variables, en función de la presencia de cultivos, en relación a los que no tienen cultivos, dependiendo además, de las especies agrícolas cultivadas y su biogeografía.