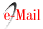Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO  uBio
uBio
Share
Revista CON-CIENCIA
Print version ISSN 2310-0265
Rev.Cs.Farm. y Bioq vol.6 no.1 La Paz June 2018
ARTÍCULOS DE ESTUDIANTES
Diseño y evaluación de primers in silico del gen E1 del virus de chikungunya
para Real-Time PCR (qPCR)
Design and Evaluation of primers in silico of the E1 gene of chukungunya virus
for Real-Time PCR (q PCR)
DE LA FUENTE, ARIEL1 ROMERO CALLE, DANITZA XIOMARA1 CÁRDENAS ALEGRÍA, OSCAR1
ÁLVAREZ ALIAGA, MARÍA TERESA1
1Instituto de Investigaciones Fármaco Bioquímicas (IIFB), Facultad de Ciencias Farmacéuticas y Bioquímicas, UMSA.
FECHA DE RECEPCIÓN: 13 DE MARZO DE 2018 FECHA DE ACEPTACIÓN: 9 DE MAYO DE 2018
Resumen
El diseño de primers es una parte sustancial dentro de los ensayos de PCR, el mismo es aplicado en la caracterización de microorganismos, secuenciación, cuantificación, etc. En los últimos años se ha incrementado herramientas para este fin, se realizaron nuevas variantes, crearon nuevos enfoques, muchas de estas herramientas son de licencia comercial y otros de acceso libre y código abierto permiten al usuario mejorar o modificar según requerimiento el diseño de primers. En este trabajo comparamos las características básicas dentro del diseño de primers, empleando 2 tipos de herramientas web de acceso libre y código abierto: Primer-BLAST y Primer3Plus, se realizó el diseño in silico, análisis de estructuras secundarias y verificación de la especificidad de los primers obtenidos. Los resultados mostraron que el uso de variables adicionales o uso de penaltis mediante el software Primer-3Plus afectan en la especificidad, formación de estructuras secundarias de los primers y de los amplicones. Además, se determinó que los parámetros que tienen muchos software por defecto, no aseveran el diseño de primers convenientes y/o específicos, en este sentido es importante realizar el uso de penaltis. Asimismo, se identificó que el uso de Primer3Plus ha permitido disminuir la formación de estructuras secundarias, sin afectar la especificidad de amplificación del marcador seleccionado.
PALABRAS CLAVE Diseño de primers, estructuras secundarias, qPCR y software.
Abstract
Primer design is a substantial part of PCR assays, which is used in characteriza-tion of microorganisms, sequencing, quanti-fication, etc. In recent years, new tools have been added for this purpose, new variants and approaches have been created, many of these tools are commercial and others are free including open source, giving the pos-sibility to improve or modify as required. In this work, we compared the basic charac-teristics of the primers design, using 2 types of free and open source web tools, Primer-BLAST and Primer3Plus, in this sense the in silico design, analysis of secondary struc-tures and verification of primers designed. The results showed that the use of addi-tional variables or use of penalties using the software Primer3 Plus affect in specificity, secondary structures formation in primers and in the products of amplification. In ad-dition, default parameters, that many software have, do not imply that suitable and / or specific primers can be obtained, mean-ing the importance of the use of penalties. Therefore, the use of Primer3Plus has al-lowed to decrease the formation of second-ary structures without affecting the ampli-fication specificity of the selected marker.
KEYWORDS Design of primers, secondary structures, qPCR and software.
INTRODUCCIÓN
La tecnología genómica moderna permite estudiar millones de regiones de cada muestra de ADN. La mayoría de estas tecnologías requiere la amplificación de la reacción en cadena de la polimerasa (PCR), técnica que requiere el uso de primers específicos los mismos no deben formar estructuras secundarias que afecten el rendimiento de la PCR.
El amplio uso de primers universales o publicados en artículos de investigación de referencia son generalmente el resultado del diseño primers, el cual requiere experiencia y destreza por estar sujeto a posibles errores (Camilo et al., 2016), en ausencia de primers de referencia es imprescindible el diseño de los mismos. En este sentido, existe una demanda reciente de métodos o algoritmos para su diseño, debido también a la proliferación de nuevas tecnologías dentro la genómica y otros campos relacionados (Kim, Kang, An, Koo, & Kim, 2016), además se ha reportado que estos métodos presentan resultados favorables (Chuang, Cheng, & Yang, 2013) en los diferentes ensayos e investigaciones incluyendo aplicaciones clínicas (Thierry, y otros, 2014) para la identificación de diferentes microorganismos emergentes. No obstante, varios de estos primers han sido diseñados para marcadores específicos o particulares, por lo tanto, la amplificación de estos marcadores no podría ser las mismas o semejantes, afectando de esta forma el éxito del PCR.
Aunque no todos los aspectos del proceso de la qPCR están completamente descritos y modelados, se conocen ciertos factores que afectan la tasa de éxito de la qPCR, en sistemas que usan el SYBER1 como intercalante Green, la detección es más simple y económica de qPCR pero la unión del primer a regiones repetidas de la secuencia de DNA puede ser errónea y generar productos inespecíficos, otro factor determinante es la temperatura de fusión (Tm) (Pachón, 2017). Los falsos positivos resultan en la formación de primers dimers y amplificación no especifica. El éxito de la PCR depende de la calidad del templado, Taq polimerasa, solución buffer y el diseño de primers en los cuales debe haber un balance entre especificidad y eficiencia.
El desarrollo informático ha permitido la creación de diversos softwares que facilitan el diseño de primers incrementando la tasa de éxito del qPCR. Los cuales permiten tanto diseño automático como diseño particular o especifico, de tal forma que nuevos usuarios puedan realizar el diseño manual, y un usuario con experiencia puede realizar un diseño más avanzado (Wang eí al., 2016; Lee et al., 2015; Yang et al., 2015), en el que se emplea el uso de parámetros adicionales que son un conjunto de variables que intervienen en su características (Presti et al., 2014).
Existen una variedad de software para el diseño de primers muchos de acceso libre como PrimerBIast y Primer3Plus empleado. Los autores del diseño y la creación digital de Primer3Plus, han examinado las versiones más recientes de navegadores tales como Firefox, Internet Explorer, Safari and Konque-ror, permitiendo su uso en cualquier navegador. Por otro lado, Primer3Plus funciona con JavaScript, Microsoft Windows y en cualquier sistema operativo basado en Unix/Linux.
Cuando se diseñan y analizan primers se deben tomar en cuenta principalmente 3 aspectos descritos por Thornton & Basu: 1) Los dímeros y horquillas de los primers que se produzcan no deberán tener un AG muy negativo (< -3.5 kcal/mol), 2) No debe existir horquillas en el extremo 3', y 3) Las estructuras secundarias que resulten, no deben tener un Tm mayor a la temperatura de alineamiento.
El diseño de primers está siendo extensamente implementado en muchos laboratorios, sean clínicos o de investigación, permitiendo reducir costos, también permite tener un control personalizado y más completo sobre el experimento de la PCR.
El presente estudio tiene por finalidad diseñar primers para el gen E1 del virus Chikungunya mediante 2 tipos de software, ambos de acceso libre, Pri-merBIast y Primer3Plus para qPCR. La identificación de este gen (E1 codifica una proteína de membrana conocida por incrementar su habilidad de infección posterior a su mutación) podría ser útil para emplearlo como marcador genético si inicialmente se realiza una retrotranscripción puesto que el virus de tipo RNA de cadena sencilla.
MATERIALES Y MÉTODOS
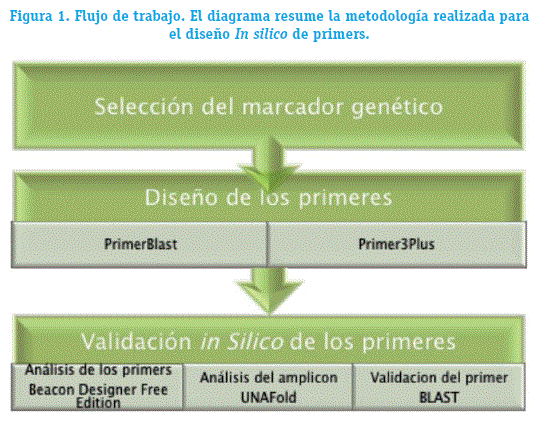
El diseño de primers inicialmente se seleccionó el marcador o región genética del blanco, en gen E1 del virus de Chikunguya (Rodríguez eí al., 2015), se utilizó dos programas diferentes para diseño de oligonucleótidos denominados PrimerBIast y Primer3Plus y posteriormente se realizó el análisis de los oligos diseñados con el programa Beacon Designer Free Edition y del ampli-cón resultante mediante UNAFold, figura1.
Búsqueda del marcador genético
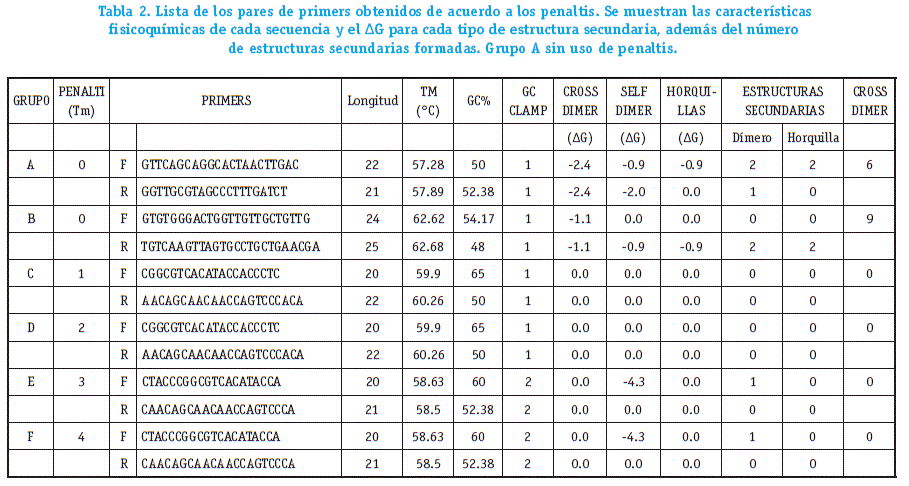
La secuencia nucleotídica (563 bp) del gen E1 del virus Chikungunya (CHKV) (Evia, 2015) fue obtenida mediante una búsqueda en la base de datos GenBank, número de acceso JX142137.1. (Benson et al., 2000) para el diseño de los primers orientados al ensayo de PCR cuantitativo (qPCR) mediante parámetros estándar descritos por Rodríguez et al. (2015).
Diseño de primers para el gen El del virus CHKV
Se ha diseñado primers que amplifiquen la región del gen E1 del virus CHKV mediante dos sitios web que proporcionan software oniine: 1) Primer-BLAST (Ye et al., 2012) y 2) Primer3Plus (Untergasser, et al., 2007).
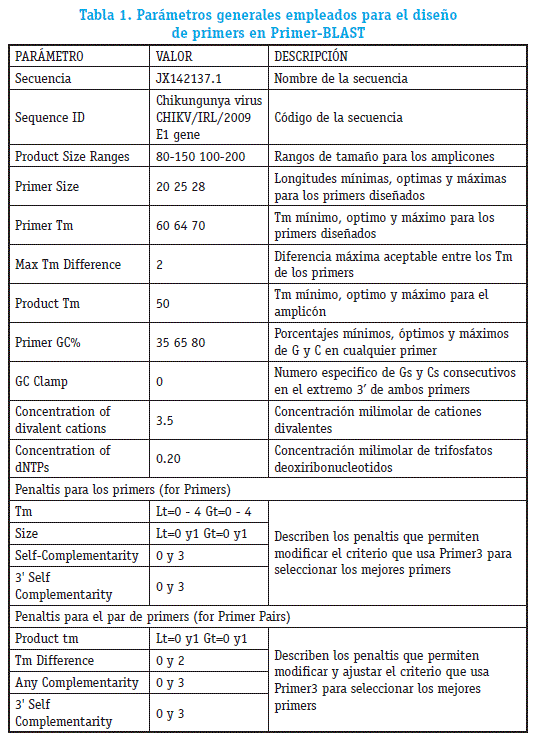
1. El diseño de primers en el software Primer-BLAST del NCBI (https://www.ncbi.nlm.nih.gov/tools/primer-blast/index.cgi?LINK_LOC=BlastHome) fue realizado de acuerdo a Rodríguez et al. (2015) y Untergasser et al. (2015), para el resto de los parámetros se utilizó los valores predeterminados por el software, figura2 y tabla 1 (Rodríguez eía/., 2015; Untergasser, et al., 2007).
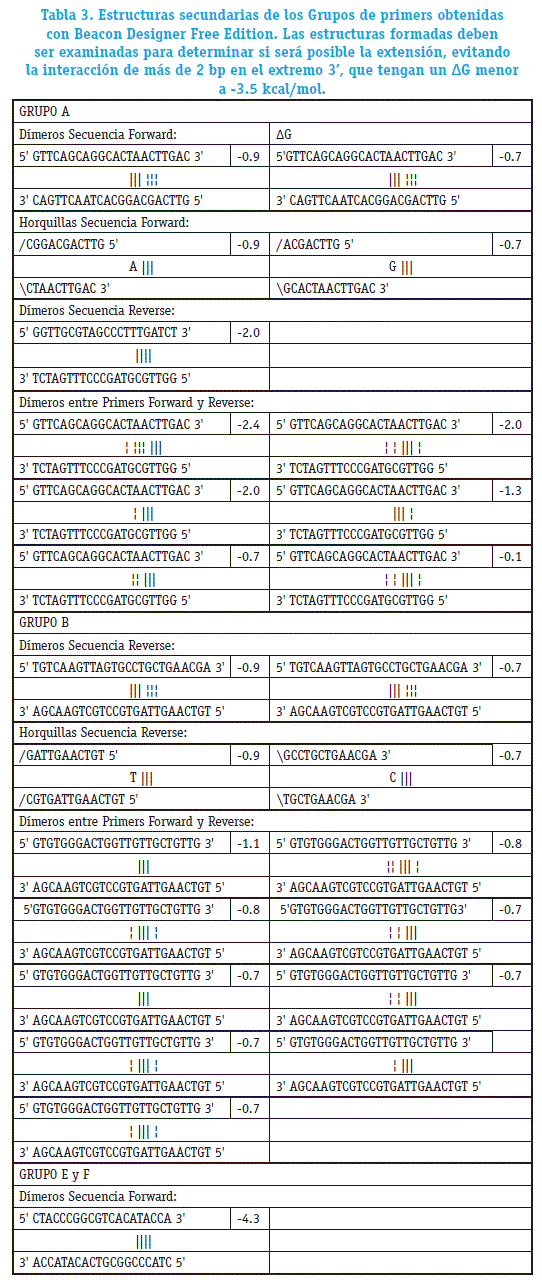
2. Primer3Plus (Bioinformatics, http://www.bioinformatics.nl/cgi-bin/primer3plus/primer3plus.cgi) fue empleado para el diseño de primers(figura 3), inicialmente la secuencia del gen E1 en formato FASTA fue ingresada en Principal (Main), posteriormente se asignaron valores fisicoquímicos óptimos para el diseño de primers (tabla 1) en la sección de configuraciones avanzadas (Advanced Settings), consecutivamente los penaltis (Penalty weights) fueron ajustados en un rango de 0 a 4 en mediante la variación de la temperatura de fusión (Tm) uno de los parámetros más influyentes para la obtención de primers específicos (Untergasser eí a/., 2007; Rodríguez eí a/., 2015), de acuerdo a estas modificaciones se obtuvieron 6 grupos de primers (A, B, C, D, E, y F), en el grupo A se asignó el valor de 0 a todos los parámetros en la sección de Penaltis y para el resto de los grupos se asignó los valores de penaltis predeterminados por el software, el resto de los parámetros fueron estándar (Thornton & Basu, 2011).
Análisis In silico de los primers y productos de amplificación
La calidad y especificidad de los primers obtenidos fueron analizados empleando Beacon Designer Free Edition (PRIMER Biosoft, http://www.pre-mierbiosoft.com/qOligo/Oligo.jsp?PID=1), se ingresó el par de primers dentro su interface, seleccionando la opción SYBR®Green y dejando el resto de los campos de forma predeterminada (Figura 4). Asimismo, se identificó la formación de posibles estructuras secundarias (Apte, 2007).
Por otro lado, se analizaron los amplicones resultantes mediante el software UNAFold (IDT, Integrated DNA Technologies, Inc., https://www.idtdna.com/UNAFold), se ingresó la secuencia del amplicon resultante y asigno el valor de 60 °C a la temperatura y realizó click en Submit, consecutivamente las variables termodinámicas de los amplicones y la formación de dímeros fueron evaluados.
Finalmente, se determinó la especificidad de los primers de acuerdo a los valores de identidad (Ident) y E-Value presentados en Nucleotide-BLAST (National Center for Biotechnology Information, U.S. National Library of Medicine 8600 Rockville Pike, Bethesda MD, 20894 USA https://blast.ncbi.nlm.nih. gov/Blast.cgi?PAGE_TYPE=BlastSearch).
2 RESULTADOS Y DISCUSIÓN
Diseño de primers mediante el Software Primer-BLAST
Inicialmente, se utilizó la secuencia obtenida con número de acceso JX142137.1 (Figura 5), se ingresó esta secuencia en la interfaz de Primer-BLAST (Figura 2).
El tiempo de procesamiento del programa para la búsqueda de los pri-mers fue aproximadamente 72 segundos, al terminar el programa reporto una lista de los primers encontrados (Figura 6), en esta lista el primer par de pri-mers obtenidos se muestran en la tabla 2.

Posteriormente, el par de primers obtenidos fueron analizados de forma In silico con los programas correspondientes.
Análisis de los primers diseñados en PrimerBLAST
Inicialmente, se analizó el par de primers con Beacon Designer Free Edi-tion, diseñado para la evaluación de oligos por sus propiedades termodinámicas y estructuras secundarias. Posteriormente, se analizó la secuencia amplificada con UNAFold, que permite predecir los plegamientos o hibridación de ácidos nucleicos (Markham & Zuker, 2008).
Los valores fisicoquímicos, los dímeros y horquillas que podrían formar los primers en el experimento de PCR fueron determinados mediante el software Beacon Designer (Figura 7).
De acuerdo a las condiciones de reacción estándar, los resultados obtenidos presentan un vínculo de color rojo que al activarlos se observa la sección donde se encuentran las estructuras secundarias identificadas, agrupadas según el tipo de estructura secundaria, Cross Dimer son las formaciones entre Forward y Reverse, Self Dimer son las formaciones entre dos primers del mismo tipo. Para este experimento en la Figura 7 se observan los resultados con AG negativos, lo que implica la formación de varios dimeros y horquillas.
1) Para el primer forward (Sense primer) el valor de AG fue de -5,2 kcal/ mol en categorizado un valor bajo de acuerdo a los valores de AG reportados por Thornton &Basu, 2011, los mismos no deben ser menores a -3,5 kcal/mol. Por lo tanto, existe la probabilidad que se formen estructuras secundarias de tipo 'Self Dimer', la energía necesaria para romper los enlaces de estos dimeros tendrá que ser mayor. Sin embargo, se debe observar si los dimeros tienen interacciones en los extremos 3'.
2) Para el primer reverse (Anti-sense primer) la posible formación del 'Self Dimer' presento un AG de -1,5 kcal/mol.'
Asimismo, se observó la presencia de una horquilla (AG bajo -1 kcal/mol) y un dímero en el primer Forward con interacción en el extremo 3' (Lorenz, 2012). Asimismo, en el parámetro 'Cross Dimer' se observó valores inferiores a -1 kcal/mol lo cual indica la formación dimeros con interacciones en el extremo 3' con (Thornton & Basu, 2011).
Análisis del producto de amplificación del gen El obtenido mediante Primer-BLAST
El amplicon obtenido de 80 bp fue analizado mediante el software UNA-Fold, se identificó una estructura secundaria (Figura 4). Asimismo, se determinó que la temperatura fusión era superior a la temperatura de alineamiento, por tanto, la formación de estructuras secundarias podría interferir en el desarrollo de la qPCR.
Por lo tanto, el Grupo de primers diseñados mediante Primer-BLAST para amplificar el gen E1 del virus CHKV deben ser descartados, y se deben evaluar otros primers.

Diseño de los primers con Primer3Plus
Se obtuvo 6 grupos de primers: A, B, C, D, E y F de acuerdo a la variación de penaltis, los grupos C y D no formaron estructuras secundarias (AG negativas) de acuerdo a el software Beacon Designer, el primer reverse del Grupo B (Tabla 3), el AG se encuentra dentro de los rangos de referencia (Thornton & Basu, 2011). Adicionalmente, se observa que los otros grupos si bien presentan una temperatura dentro del rango establecido, forman estructuras secundarias. La temperatura de fusión no debe superar los 60 °C (Tm) (Thornton & Basu, 2011), ya que podría existir la formación de estructuras secundarias.
Se observaron estructuras secundarias en los grupos de primers A, B, E y F, no obstante, los grupos A y B están dentro de los rangos aceptables (> -3.5 kcal/mol y las interacciones entre las bases están distantes al extremo 3'). Sin embargo, los grupos E y F deben descartarse porque presentan AG muy negativas y muestran interacciones en más de 2 pares de bases, estas interacciones pondrían afectar el rendimiento de la PCR.
Se determinó que al asignar valores 1 y 2 como penaltis, los primers diseñados son de mayor estabilidad, y al analizarlos en Beacon Designer no formaron estructuras secundarias, lo que implica que la PCR que se realice con estos primers tendría mejores resultados. Por otro lado, al asignar valores de penaltis mayores a 2, se observa la formación de estructuras secundarias.
Análisis de los amplicones obtenidos en Primer3Plus.
Los amplicones por cada Grupo de primers diseñados en Primer3Plus fueron analizados en UNAFold. En la Figura 5, los primers del Grupo C y D presentaban un AG positivo o nulo, no obstante los valores de Tm están dentro de los valores de referencia, por tanto se determinó que los mismos son adecuados para realizar un ensayo de qPCR en el cual la reacción no excede la temperatura de alineamiento de 60°C, por tanto, esta reacción es efectiva.
Con respecto a los resultados obtenidos, se observó que el uso de penaltis en el software Primers3plus permitió una búsqueda más adecuada de pri-mers, que poseen un equilibrio entre especificidad y eficiencia para un mejor rendimiento en el ensayo de PCR en tiempo real. A diferencia del diseño de primers mediante el uso del software Primers3 en el cual se observó la formación de estructuras secundarias.
Para diseñar primers se debe tener en cuenta no solo los tiempos de análisis, sino el tiempo de búsqueda que dependerá principalmente de la longitud de la secuencia. En este sentido, diferentes software tienen en sus parámetros o variables, valores predeterminados automatizados y avanzados, dirigidos específicamente al diseño de primers de acuerdo a los tipos de PCR.
Especificidad de los primers diseñados
La especificad de los primers fueron evaluados mediante el sitio web de Nucleotide-BLAST, los resultados obtenidos se observan en la Tabla 5, todos los primers presentaron un porcentaje de cobertura de 100%, lo cual indica alta similitud de la secuencia con el gen E1 de virus de la Chikungunya.
Se determinó que el E-valor más cercano a la nulidad corresponde al grupo B con 0.002, seguido del grupo A con 0.024, pero este grupo no coincide con el target que se busca; y por último los grupos C, D, E y F coinciden con el target que se busca, esta medida es más significativa cuanto más cerca de 0.0 esté, (Sánchez, 2012), la cual es la medida más significativa para evaluar la especificidad de primers en el portal Blast del NCBI.
Por lo tanto, todos los grupos, excepto el grupo A, han tenido resultados significativos para el target de búsqueda, óptimos para su uso en la identificación del virus de la Chikungunya mediante PCR en tiempo real.
CONCLUSIONES
Existen varias guías para el diseño de primers, en este trabajo se han diseñado primers In silico empleando dos tipos de software libres, Primer-BLAST y Primer3Plus, para amplificar la región del gen E1 del virus de la Chikungunya empleando los mismos parámetros de entrada para ambos softwares y se determinó la factibilidad, eficacia que presenta el uso de penaltis y la eficiencia de Primer3Plus que permitió ajustar los parámetros establecidos y el ajuste de penaltis a un solo parámetro (Tm), para obtener primers más específicos sin alterar los parámetros requeridos para llevar a cabo el ensayo de PCR. Los primers obtenidos mediante Primer3Plus son más estables y específicos. Por lo tanto, emplear el uso de penaltis a los diferentes parámetros permitiría mejorar la calidad de los primers y por ende efectivizar un ensayo de qPCR.
AGRADECIMIENTOS
Este trabajo ha sido realizado gracias al apoyo académico del Dr. Oscar Cárdenas.
REFERENCIAS
Alicia Rodríguez, M. R. (2015). Design of Primers and Probes for Quantitative. En J. M.Walker, PCR Primer Design (págs. 31-56). New York: Chhandak Basu. [ Links ]
Apte, A. (8 de Agosto de 2007). PREMIER Biosoft International announced today the reléase of a free edition of Beacon Designer, their popular oligo design tool for real time PCR. Obtenido de PREMIER Biosoft: http://www.premierbio-soft.co/news/news/Beacon_Designer_ FreeEdition.html [ Links ]
Camilo, C, Lima, G., Maluf, V, Guido, R., & Polikarpov, I. (2016). HTP-OligoDesig-ner: An Online Primer Design Tool for High-Throughput Gene Cloning and Site-Directed Mutagenesis. Journal of Compational Biology, 23(1), 27-29. [ Links ]
Chuang, L.-Y, Cheng, Y.-H., & Yang, C.-H. (2013). Specific primer design for the polymerase chain reaction. Biotechnol Lett, 1-9. [ Links ]
Benson, D., Karsch, I., Lipman, D., Ostell, J., Rapp, B. & Wheeler, P. (2000). GenBank. Obtenido de National Center for Bio-technology Information. Nucleic Acids Res. 28(1): 15-18. [ Links ]
Evia, J. R. (2015). Fiebre chikungunya. ¿Es acaso la próxima amenaza? Revista Latinoamericana de Patología Clínica y Medicina de Laboratorio. 62 (1): 20-32. [ Links ]
Ye, J., Coulouris, G., Zaretskaya, I., Cutcuta-che, I. & Rozen, S. (2012). Primer-BLAST: A tool to design target-specific primers for polymerase chain reaction. BMC Bioinformatics. 13:134. [ Links ]
Kim, H., Kang, INI., An, K., Koo, J., & Kim, M.-S. (6 de Mayo de 2016). MRPrimerW: a tool for rapid design of valid high-quali-ty primers for múltiple target qPCR ex-periments. Nucleic Acids Research Ad-vance Access, 1-8. [ Links ]
Lee, E., Kim, E., Shin, Y, & Song, J. (2015). Design and testing of multiplex RT-PCR primers for the rapiddetection of influenza A virus genomic segments: Application toequine influenza virus. Journal of Vi-rological Methods. 228 (1): 114-122. [ Links ]
Lorenz, T. C. (Mayo de 2012). Polymerase Chain Reaction: Basic Protocol Plus Troubleshooting and Optimization Stra-tegies. Journal of Visualized Experi-ments. (63): 1-15. [ Links ]
Markham, N. & Zuker, M. (2008). UNAFold: software for nucleic acid folding and hy-bridization. Methods Mol Biol. 453:3-31 [ Links ]
Miya, M., Sato, Y, Fukunaga, T., Sado, T., Poulsen, J. Y, Sato, K., . . . Iwasaki, W. (2015). MiFish, a set of universal PCR primers for metabarcoding environ-mental DNA from fishes: detection of more than 230 subtropical marine spe-cies. Royal Society Open Science, 33. [ Links ]
NA. (si.). TECHVAULT. Obtenido de Inte-grated DNA Technologies, Inc.: http://www.idtdna.com/pages/support/te-chnical-vault/faq-ofa/apphcation-support/scitools/faqs/2011/06/24/ what-is-the-recommended-delta-g-va-lue-for-self--and-hetero-dimers-
Pachón, D. M. (8 de Febrero de 2017). DISEÑO DE OLIGONUCLEOTIDOS INICIADORES (Primers). Obtenido de Centro de Bioinformática del Instituto de Biotecnología Universidad Nacional de Colombia: http://bioinf.ibun.unal.edu.co/ documentos/primers/primer.php [ Links ]
Prediger, E. (si.). Designing PCR primers and probes. Obtenido de Integrated DNA Technologies, Inc.:http://www.idtdna.com/pages/decoded/decoded-articles/pipet-tips/decoded/2013/10/21/desig-ning-pcr-primers-and-probes [ Links ]
Prestí, A., Lai, A., Celia, E., Zehender, G., & Ciccozzi, M. (2014). Chikungunya virus, epidemiology, clinics and phylogenesis. Asian Pacific Journal of Tropical Medicine. 7 (12): 925-932. [ Links ]
Rodríguez, A., Rodríguez, M., Córdoba, J., & Andrade, M. (2015). Design of Primers and Probes for Quantitative Real-Time PCR Methods. Methods Mol Biol. 1275 (1):31-56. [ Links ]
Sánchez, A. (20 de Junio de 2012). Aplicaciones guiadas: Blast. Genome Browsers. Obtenido de UNITAT D'ESTADISTICA I BIOINFORMATICA: http://ueb.vhir.org/dl584 [ Links ]
Thierry, A. R., Mouliere, F., Messaoudi, S. E., Mollevi, C, Lopez-Crapez, E., Rolet, F., . .. Nouaille, M. (2014). Clinical validation of the detection of KRAS and BRAF mu-tations from circulating tumor DNA. Na-ture Medicine, 430-435. [ Links ]
Thornton, B., & Basu, C. (2011). Real-Time PCR (qPCR) Primer Design Using Free Online Software. Biochemistry and Molecular Biology Education. 39 (2): 145-54. [ Links ]
Untergasser, A., Nijveen, H., Rao, X., Bis-seling, T., Geurts, R., & Leunissen, J. A. (2007). Primer3Plus, an enhanced web interface to Primer3. Nucleic Acids Res. 35 (Web Server issue): W71-W74. [ Links ]
Wang, Y, Tiwari, V. K., Rawat, INI., Gilí, B. S., Huo, INI., You, F., & Gu, Y. (2016). GSP: a web-ba sed platform for designing. Bioinformatics Advance Access. 32 (15): 1-2. [ Links ]
Yang, Y, Kung, T., Hu, C, & Lin, S. (2015). De-velopment of primer pairs from diverse chloroplast genomes for use in plant phylogenetic research. Genetics and Molecular Research. 14 (4): 14857-14870 [ Links ]