Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO  uBio
uBio
Share
Revista CON-CIENCIA
Print version ISSN 2310-0265
Rev.Cs.Farm. y Bioq vol.4 no.1 La Paz June 2016
ARTÍCULO DE INVESTIGACIÓN
Identificación genética del agente causal y el insecto vector de la enfermedad de chagas circulante en la amazonía boliviana: Provincia Iturralde
BUITRAGO ROMERO, ROSIO1 BRENIÉRE SIMONE, FRÉDÉRIQUE2 TABORGA MANRIQUE, XIMENA1 QUISPE ARUHUITO, RICHARD1 REVOLLO ZEPITA, SUSANA1
CORRESPONDENCIA: VIRZOTAU@YAHOO.COM
1 Laboratorio de Genética Molecular, Instituto SELADIS, Fac. Cs. Farmacéuticas y Bioquímicas, UMSA
2. Institut de Recherche pourle Développement (IDR), Francia
FECHA DE RECEPCIÓN: 29/02/2016 FECHA DE ACEPTACIÓN: 9/05/2016
Resumen
Trypanosoma cruzi es el agente causal de la enfermedad de Chagas, la misma que representa una grave amenaza para la salud en las Américas, con 8 a 9 millones de personas infectadas y 25 a 90 millones en riesgo (Schmunis y Yadon, 2010; WHO, 2008). Este parasito pertenece a la familia Trypanosomatidae, y es transmitida por insectos hematófagos pertenecientes a tres géneros (Triatoma, Rhodniusy Panstrongylus).
En Bolivia la distribución geográfica del Género Rhodnius y su rol en la transmisión de T. cruzi no está clara porque se realizaron identificaciones erróneas basadas solamente en métodos morfológicos. En el presente trabajo se ha introducido la tecnología molecular para la identificación tanto del insecto vector como del agente transmisor de la enfermedad de Chagas.
Un total de quince Triatominos fueron capturados en regiones silvestres de los Municipios de San Buenaventura e Ixiamas a partir de 82 palmeras utilizando 4 trampas por cada palmera. De los quince Triatominos recolectados solo diez se utilizaron para la identificación de la especie y cinco de ellos estaban infectados por Trypanosoma cruzi.
La identificación de la filogenia de los especímenes de Triatominos se realizó por análisis de genes de citocromo B "cyt b" del DNA mitocondrial y gen nuclear D2, correspondiente a la región 28S del RNA. Al mismo tiempo se realizó la identificación del DTU de los aislados de Trypanosoma cruzi por la técnica de Multiplex PCR del Minie-xon (MMPCR).
Los resultados nos permitieron revelar que todos los especímenes provenientes de San Buenaventura, pertenecen a la especie Rhodnius stali (SB3, SB4, SB5 y SB6), mientras que, de los ocho especímenes provenientes de Ixiamas, cuatro pertenecen a la especie Rhodnius stali (IX3, IX4, IX6, IX8), y dos a la especie Rhodnius robustus (IX2, IX5) basados en el análisis del DNA mitocondrial cyt b, confirmando estos hallazgos con el análisis del gen D2. Asimismo, se identificó al DTU TcI de Trypanosoma cruzi circulante en esta región.
Estos resultados confirman algunos estudios morfométricos realizados en la identificación de las especies del Género Rhodnius y dan a conocer el DTU "TcI" de Trypanosoma cruzi que está circulando en la amazonía boliviana.
Palabras clave
Enfermedad de Chagas, Rhodnius, Amazonia boliviana
Abstract
Trypanosoma cruzi, the agent of Chagas disease, is a serious threat to health in the Americas, accounting for the highest disease burden in Latin American, with eight to nine million people infected and 25-90 million at risk (Schmunis and Yadon, 2010; WHO, 2008). This parasite, which belongs to the Trypanosomatidae family, is mainly transmitted by blood-sucking bug vectors, belonging to three genera (Triatoma, Rhodnius, and Panstrongylus).
In Bolivia the geographical distribution of the genera Rhodnius and its roll in the T. cruzi transmission is not clear, because erroneous identifications were realised based only on morphological methods In the presentwork the molecular technology has been introduced in order to identify the insect vector as the transmitting agent of the disease of Chagas.
A total of fifteen Triatomines were captured in wild regions of the "Municipios San Buenaventura" and "Ixiamas" from 82 palms trees using 4 by each palm. Of the fifteen Triatomines collected only ten were used for the species identification and five of them were infected by Trypanosoma cruzi.
The identification of the philogeny of specimens of Triatomines was performed by analysis of genes of cytochrome B "cyt b" of the mitocondrial DNA and nuclear gene D2, corresponding to the region 28S of the RNA. At the same time DTU identification of isolates of Trypanosoma cruzi by Multiplex PCR technique miniexon (MMPCR) itwas performed.
The results allowed to reveal that all the specimens from San Buenaventura, belong to the species Rhodnius stali (SB3, SB4, SB5 and SB6), whereas, of eight specimens from Ixiamas, four belong to the species Rhodnius stali (IX3, IX4, IX6, IX8), and two to the species Rhodnius robustus (IX2, IX5), based on the analysis of the mitocondrial DNA cyt b, confirming these finding with the analysis of the gene D2. Also, the DTU Tcl Trypanosoma cruzi was identified circulating in this region.
These results confirm some morphometric studies identifying the species of Rhodnius and they show the DTU "Tcl" of Trypanosoma cruzi that is circulating in the Bolivian Amazon.
Key words
Chagas disease, Rhodnius, Bolivian Amazon
INTRODUCCIÓN
La Enfermedad de Chagas, es producida por Trypanosoma cruzi, un protozoario flagelado de la familia Trypanosomatidae, y transmitida por insectos hematófagos pertenecientes a los géneros Triatoma, Rhodnius y Panstrongylus, que viven en ecosistemas diversos (WHO, 2007; Hotez y col., 2008; Schmunis y Yadon, 2010). Es considerada un problema de salud pública dada su magnitud y trascendencia, siendo una de las enfermedades transmisibles de más amplia distribución en el continente americano (Silveira, 2007).
El parásito causal se presenta en una gran variedad de cepas e infecta 150 especies de 24 familias de animales domésticos y silvestres, siendo la infección en el hombre de carácter accidental (Guhl, 2009). Al igual que en muchas otras enfermedades metaxénicas, la principal vía de transmisión es la vectorial, en la que interactúan el parásito, el vector y el ser humano, y en la que además desempeña un papel importante, un conjunto de condiciones ambientales y sociales que permitan este encuentro Briceño-León, 2009). Sin embargo, se han descrito otras formas de transmisión, entre las que se encuentran la transfusional, la congénita, por trasplante de órganos, por accidentes laborales y, más recientemente, la controversial transmisión oral (Guhl, 2009).
Se ha indicado que la enfermedad de Chagas se encuentra presente en todo el territorio del continente Suramericano y gran parte de Centroamérica, representando una grave amenaza para la salud de los países de la región (Guhl, 2009). La Organización Mundial de la Salud (OMS, 2012) calcula que a nivel mundial alrededor de 10 millones de personas están infectadas, principalmente en América Latina, donde la enfermedad es endémica; además refiere que más de 25 millones de personas están en riesgo de adquirir la enfermedad, indicando que en 2008,10.000 personas murieron por esta causa y en ese sentido, la Organización Panamericana de la Salud refirió que a lo largo de 21 países, existían 7'694.500 infectados para 2005, representando una prevalencia de 1.448% (OMS-2, 2012), sin embargo, no se cuenta con información más reciente que refleje la verdadera magnitud del problema en zonas nuevas afectadas por estos insectos como la Amazonia.
El rol de los insectos transmisores de esta enfermedad en la Amazonia Boliviana es poco conocida debido a que los investigadores, epidemiólogos y las medidas de control han sido focalizadas hasta ahora sobre Triatoma infestans, el mayor vector de la región Andina de este país. Se ha reportado que insectos del género Rhodnius los principales involucrados en la transmisión de Trypanosoma cruzi en la Amazonia. La importancia epidemiológica de Rhodnius está relacionada a una fuerte tendencia a la domesticación pero también a la capacidad de entrar esporádicamente a las casas sin colonizarlas. Una caracterización es necesaria para establecer que especies podrían estar involucradas, más aún si ya se tiene un reporte de transmisión oral de la enfermedad de Chagas en la región Amazónica de Bolivia por estos insectos (Santalla y col., 2010).
Con el fin de describir atributos genéticos en especies del Género Rhodnius los mismos que pueden distinguir variaciones mínimas de especie, realizamos estudios genéticos basados en el análisis de genes de citocromo B "cyt b" del DNA mitocondrial y genes D2, región 28S del RNA. Nuestros resultados han permitido identificar las especies del Género Rhodnius que están circulando en la Amazonía Boliviana.
Por otra parte, entre los marcadores genéticos que pueden identificar los diferentes grupos de T. cruzi previamente descritos están los que discriminan TcI, TcII y TcIV de T. cruzi usando la técnica de Multiplex PCR del Miniexon (MMPCR) (Fernandes y col., 2001. Recientemente, el análisis de MMPCR fue aplicado a un gran muestreo de cepas (caracterizadas previamente por tipificación multilocus) perteneciendo a seis DTUs, mostrando 3 tamaños precisos de amplificación: a 200 pb los productos de PCR para TcI, a 250 pb para TcI I, a 200 pb para TcI, TcI I, TcV y TcVI y a 150 pb para TcI I I y TcI V (Bosseno y col., 1996). Este método fue exitosamente aplicado en la identificación de rápida de DTU en el aparato digestivo de triatomineos (Bosseno y col., 2009; Bosseno y col., 2006).
En este trabajo se ha caracterizado genéticamente a especies del género Rhodnius recolectados en una región amazónica de Bolivia y se ha establecido su rol en la trasmisión de T. cruzi, identificando además la identidad del parasito por la técnica MMPCR para la caracterización de los DTUs de T. cruzi directamente en el aparato digestivo de los insectos triatominos recolectados en el área de trabajo. Asimismo, se ha realizado la búsqueda de casos humanos autóctonos infectados por Trypanosoma cruzi usando métodos serológicos y moleculares, estableciendo la prevalencia de la enfermedad y contribuyendo al conocimiento del nuevo paradigma de la Enfermedad de Chagas en la Amazonia Boliviana.
MATERIAL Y MÉTODOS
Captura de Triatominos
Un total de quince Triatominos fueron capturados en regiones silvestres de los Municipios de San Buenaventura e Ixiamas de octubre a noviembre de 2014. Las recolecciones fueron realizadas usando trampas adhesivas con carnada de ratón (Noireau y col., 1999) en ecótopos propios de la Amazonia "copas de las palmeras". El colocado de trampas se hizo en 82 palmeras utilizando 4 por cada palmera. Antes de la disección, las heces de cada insecto fueron examinadas en busca de la presencia de Trypanosomatides por observación directa al microscopio a una ampliación de X400. Los insectos luego fueron disecados bajo una campana de bioseguridad y los aparatos digestivos fueron almacenados a -20°C.
Extracción del DNA y amplificación por PCR de Triatominos
El DNA genómico fue obtenido a partir de las patas de los insectos adultos y de estadios N3, N4 y N5 e insecto entero en los estadios N1 y N2. Estas estructuras del insecto fueron previamente lavadas con buffer PBS (Phosphated Buffered Saline) para evitar contaminación y posteriormente fueron criofracturadas según el método reportado por García y col., 1998. Con el sedimento obtenido se realizó la extracción del DNA por el mini kit QIAamp (Quiagen, Courtaboeuf, France) y el producto final fue resuspendido en 40 uL de solución de rehidratación (Millery col., 1988).
Para cada espécimen, fragmentos de 682-bp del gen "cytochrome B" (cyt b) del DNA mitocondrial fue amplificado por PCR, usando los primers: CYTB7432 (5'-GGACGWGGWATTTATTATGGATC-3') y CYTB7433 (5'-GCWC-CAATTCARGTTARTAA-3') (Lyman, 1999). Las reacciones de PCR para este gen mitocondrial fueron llevados a un volumen final de 35 ul usando 30-ng de DNA, 1X PCR buffer (0.1 M Tris-HCl, 0.5 M KCl, y 0.015 M MgCl2, pH 8.3), 250- [iM dNTP, 0.016-uM de cada primer, 35-mM MgCl2 y 2 U of Taq DNA po-limerasa. Los fragmentos fueron amplificados en el Termociclador Verity de Applied Biosystem con el siguiente programa termal: 94°C por 5 min; 35 ciclos de 94°C por 30 s, 55°C por 30 s, y 72°C por 30 s; 72°C por 10 min.
Para D2, región variable del gen 28S rDNA (D2-28S) del DNA nuclear, un fragmento de 434-bp fue amplificado por PCR usando los primers D2F (5'-GCGAGTCGTGTTGCTTGATAGTGCAG-3') y D2R, (5'-TTGGTCCGTGTTT-CAAGACGGG-3') (Porter y Collins, 1996). Las reacciones de PCR fueron llevados a un volumen final 35 ul usando 30-ng de DNA, 1X PCR buffer (0.1 M Tris-HCl, 0.5 M KCl, y 0.015 M MgCl2, pH 8.3), 250- uM dNTP, 0.025-uM de cada primer, 3-mM MgCl2 y 2 U de Taq DNA polimerasa. Después de una de-naturación inicial de 94°C por 5 min, las reacciones de PCR se llevaron a cabo en 35 ciclos a 94°C por 30 s, 60°C por 30 s, y 72°C por 30 s, seguido de una extensión final de 72°C por 5 min (Herrera-Aguilar, 2009). Los amplicones fueron enviados al Institute de Recherche pour le Dévelopment (IRD) de Montpellier, Francia, para ser purificados y secuenciados en ambas direcciones.
Mini-exon multiplex PCR (MMPCR) para T. cruzi
De los diez Triatominos vivos, cinco estaban infectados por Trypanosoma cruzi. El DNA de T. cruzi fue aislado a partir del tracto digestivo de los triatominos con el mini kit QIAamp (Quiagen, Courtaboeuf, France), de acuerdo a protocolos de muestras desangre. Tres "primers" derivados de regiones repetitivas hipervariables del mini-exon de T. cruzi y un "primer" común opuesto correspondiente a la secuencia presente en la región conservada del gen mini-exon, fueron utilizados en la reacción de Multiplex PCR. Las condiciones de PCR estuvieron basadas en las descritas por Fernandes y col., 2001), con pequeñas modificaciones. El DNA fue amplificado en 25 ul de volumen de reacción conteniendo 1X de buffer de la reacción, 1.5 mM MgCl2, 50 uM de cada nucleótido, 0.2 uM de cada "primer", 0.5 UI de Taq polimerasa. La amplificación fue realizada en un termociclador (Verity de Applied Biosystem) en condiciones previamente descritas (Fernandes y col., 2001). Los productos de PCR fueron separados en un gel de agarosa al 1.5% usando patrones de peso molecular (Eurogentec, Angers, France) y visualizado bajo luz ultravioleta con Ez-vision (Amresco, Solon, OH, USA). La discriminación entre DTUs fue realizada de acuerdo a Aliaga y col., 2011.
Análisis Genético
Todas las secuencias obtenidas fueron analizadas con el GenBank. Los alineamientos fueron hechos usando el software Bioedit 7.0.5 (Hall, 1999). Diversos datos de patrones de referencia del Género Rhodnius fueron incluidos en el análisis filogenético. Las distancias genéticas entre los especímenes estudiados fueron estimados para los genes D2 - 28S y cyt b basados en el modelo de sustitución de K 2p (Kimura,1980) como es reportado para especies emparentadas del Género Rhodnius, usando el software MEGA 6 (Tamura y col., 2011).
RESULTADOS
Filogenia de Triatominos
Nueve especímenes fueron capturados de Palmeras de zonas silvestres del Municipio de Ixiamas (IX1, IX2, IX3,IX4, IX5, IX6, IX7, IX8 e IX9) y seis especímenes de palmeras ubicadas en los alrededores del Municipio de San Buenaventura (SB1, SB2, SB3, SB4, SB5 y SB6). De los nueve especímenes de Ixiamas, seis fueron sometidos al análisis genético (IX2, IX3, IX4, IX5, IX6, IX8), mientras que, de los seis especímenes capturados en San Buenaventura solo cuatro fueron analizados (SB3, SB4, SB5 y SB6).
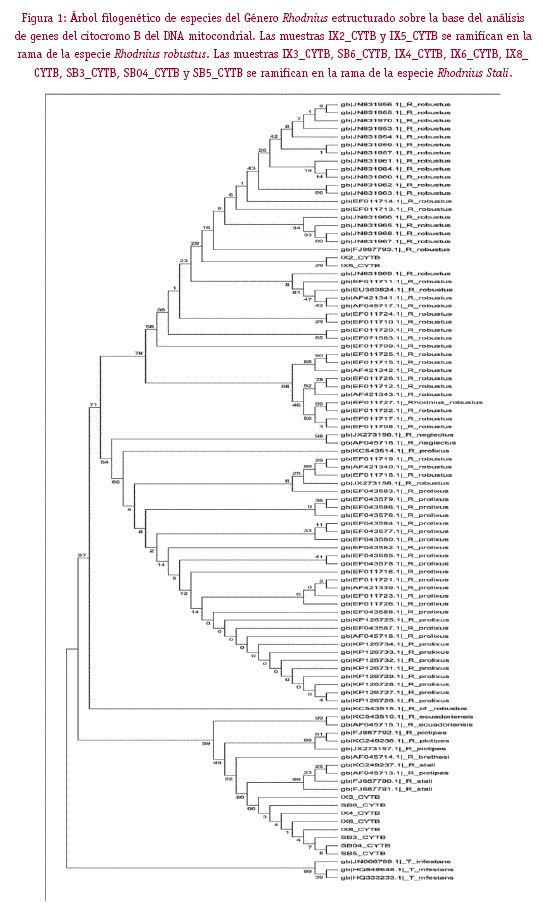
El análisis del DNA mitocondrial a través de las secuencias de citocromo B de las muestras analizadas con el GenBank, ha permitido revelar que todos los especímenes provenientes de San Buenaventura, pertenecen a la especie Rhodnius stali (SB3, SB4, SB5 y SB6), mientras que, de los ocho especímenes provenientes de Ixiamas, cuatro pertenecen a la especie Rhodnius stali (IX3, IX4, IX6, IX8), y dos a la especie Rhodnius robustus (IX2, IX5), como se puede ver en el árbol filogenético basado en este gen (Figura 1).
Realizado el análisis de los genes D2, correspondiente a la región 28S del RNA con el GenBank, se ha podido confirmar que los dos especímenes que estaban integrados en la rama de la especie Rhodnius robustus bajo el análisis del gen de citocromo B, lo están también bajo el análisis del gen D2 (IX2, IX5). Sin embargo, los especímenes provenientes tanto del Municipio de Ixiamas como de San Buenaventura bajo el análisis del gen D2, se integran más en la rama de la especie Rhodnius pictipes, siendo esta una especie muy cercana a Rhodnius stali, como se puede evidenciar en el árbol filogenético basado en este gen (Figura 2).
Análisis del mini-exon multiplex PCR (MMPCR) de T. cruzi
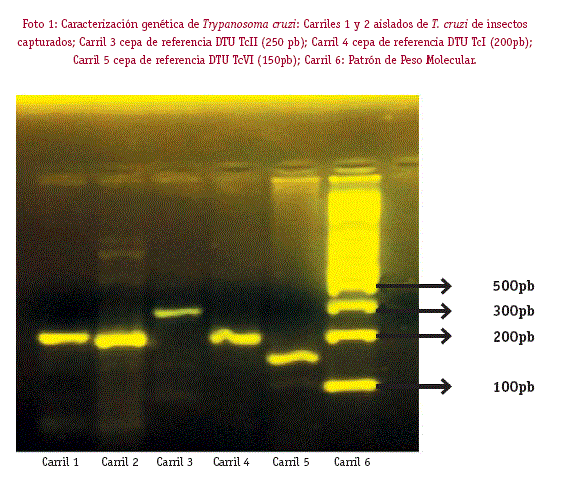
Un total de 5 muestras de DNA provenientes del tracto digestivo de Triatominos fueron procesadas por Multiplex PCR (MMPCR). La identificación de tres DTUs de Trypanosoma cruzi fueron evaluados, determinados por el peso molecular de los productos del PCR (a 200 pb los productos de PCR para TcI, a 250 pb para TcI I y a 150 pb para TcI I I y TcIV). Los resultados evidencian de los cinco especímenes de Triatominos infectados, todos portaban la cepa TcI. (producto de PCR de 200 bp) Foto 1.
DISCUSIÓN
El Género Rhodnius incluye 16 especies cuya distribución geográfica es desde el norte de la Argentina hasta América Central con mayor riqueza de especies en la Amazonía. La importancia epidemiológica de este Género no está relacionada a una vigorosa domesticación de especies como R. prolixus. Varias especies sinantrópicas esporádicamente colonizan el hábitat humano, mientras que otras entran a las casas donde pueden entrar en contacto con humanos y por lo tanto transmitir T. cruzi (Abad-Franch y col., 2010). En Colombia, Venezuela y América Central, los principales vectores de la enfermedad de Chagas son del Género Rhodnius (R. prolixus, R. pallescens) bien representados en la región de la Amazonía. En la Amazonía boliviana la distribución geográfica de Rhodnius y su rol en la transmisión de T. cruzi no está clara, especialmente porque hubo identificaciones erróneas realizadas en estudios anteriores donde especies de R. prolixus fueron registrados incorrectamente debido a la confusión con R. Robustus basados en métodos morfológicos. Monteiro y col., 2003, reportaron en un estudio de filogeografia molecular, la presencia de vectores del Género Rhodnius presentes en la Amazonia de países latinoamericanos agrupando a los mismos en cinco "clades", uno representando a Rhodnius prolixus y cuatro a Rhodnius robustus, manifiestan además que, mientras la diversidad de R. Prolixus es mas homogénea, la de R. Robustus es más dispersa y apoyado en el análisis del gen nuclear D2 (región del 28S de RNA) esta especie agrupa a una mayor variedad de especímenes del género Rhodnius. Estos datos apoyan que R. Robustus representa a un gran complejo de especies. Nuestros resultados, permitieron identificar dos especímenes dentro del complejo Rhodnius robustus (IX2, IX5) (Figura 1 y 2), apoyados en el análisis del ADN mitocondrial Cyt b y gen nuclear D2, resultados que confirman la distribución dispersa que reporta Monteiro y col., 2003.
Asimismo, la identificación genética de especies de Rhodnius stali confirma su distribución en la Amazonia boliviana como especie exclusiva de esta área. Nuestro estudio pudo detectar ocho especímenes (SB3, SB4, SB5, SB6, IX3, IX4, IX6, IX8) dentro del complejo R. stali apoyados en el análisis del ADN mitocondrial Cyt b, no obstante que, los resultados con el gen D2 en nuestro árbol filogenético no muestra la presencia de R. stali, debido a que pocos estudios genéticos han sido realizados a la fecha sobre especies circulantes en la Amazonia boliviana con estos marcadores y como consecuencia la base de datos del GenBank no incluye nuestra diversidad.
Estudios morfométricos en años pasados han demostrado la presencia de las especies R. stali y R. robustus en Bolivia (Justi, 2009, Matias y col., 2003), sin embargo, información epidemiológica acerca de R. robustus falta realizar como lo indica Matias y col., 2001, puesto que la dispersión es bastante amplia según reporte de Monteiro y col., 2003 quienes sugieren que esta especie pasó a través de un embotellamiento reciente y fue dispersada posteriormente por el hombre, conduciendo a que los niveles de divergencia secuencial entre clades dentro de cada región son compatibles con el origen Pleistocene.
Por otra parte, la diversidad genética de Trypanosoma cruzi en las zonas endémicas de territorio boliviano es bastante grande. De acuerdo a la ultima clasificación por DTUs, en Bolivia se reporta con mayor porcentaje (98,3%) el DTU TcI (producto PCR de 200 pb) mientras que los DTUs TcI I, TcV, o TcVI (producto PCR de 250 pb) y DTUs TcI I I yTcIV (producto PCR de 150 pb) solo se encontró una cepa y tres cepas, respectivamente (Breniere y col., 2012). Nuestro estudio permitió detectar la presencia del DTU TcI en los cinco aislados recolectado del Municipio de Ixiamas y el Municipio de San Buena ventura representando este encuentro en una parte del territorio de la Amazonia boliviana, confirmando esa gran dispersión de esta cepa en nuestro país. Sin embargo, deben realizarse mas estudios en otras regiones de la Amazonia que confirmen la presencia de los diversos DTUs que pudieran estar en estas regiones amazónicas.
REFERENCIAS
Aliaga C, Brenière SF, Barnabé C. (2011) Further interest of miniexon multiplex PCR for a rapid typing of Trypanosoma cruzi DTUgroups. InfectGenetEvol 11:1155-1158. [ Links ]
Bosseno MF, Telleria J, Vargas F, Yaksic N, Noireau F, y col. 1996. Trypanosoma cruzi: study of the distribution of two widespread clonal genotypes in Bolivian Triatoma infestans vectors shows a high frequency of mixed infections. Exp Parasitol ; 83:275-282. [PubMed]. Bosseno MF, Garcia LS, Baunaure F, Gastelum EM, Gutierrez MS, y col. (2006) Identification in triatomine vectors of feeding sources and Trypanosoma cruzi variants by heteroduplex assay and a multiplex miniexon polymerase chain reaction. Am J Trop Med Hyg 74: 303-305. [ Links ] [ Links ]
Bosseno MF, Barnabé C, Sierra MJ, Kengne P, Guerrero S, y col. (2009) Wild ecotopes and food habits of Triatoma longipennis infected by Trypanosoma cruziTriatomalongipennislineages I and II in Mexico. Am J Trop Med Hyg 80:988-991. [ Links ]
Briceño-León R. 2009. La enfermedad de Chagas en las Américas: una perspectiva de ecosalud. Cad Saúde Pública; 25(1):S71-S82. [ Links ]
Fernandes O, Santos SS, Cupolillo E, Mendonca B, Derre R, y col. (2001) A miniexon multiplex polymerase chain reaction to distinguish the major groups of Trypanosoma cruzi and T. rangeli in the Brazilian Amazon. Trans R Soc Trop Med Hyg 95: 97-99. [ Links ]
García A., Carrasco H., Schofield C., Stothard J., Frame I., Valente S., y col. (1998). Ran-dom Amplification of Polymorphic DNA as a Tool for Taxonomic Studies of Triatomine Bugs (Hemiptera: Reduviidae). [ Links ]
Guhl F. 2009. Enfermedad de Chagas: Realidad y perspectivas. Rev Biomed; 20:228-34. [ Links ]
Hall TA (1999) BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/ NT. NucleicAcids Symp Ser 41: 95-98. [ Links ]
Herrera Aguilar M, Be Barragán LA, Ramirez Sierra MJ, Tripet F, Dorn P, y col. (2009) Identification of a large hybrid zone between sympatric sibling species of Triatoma dimidiata in the Yucatan peninsula, Mexico, and its epidemiological importance. Infect Genet Evol 9: 1345-1351. doi: 10.1016/j. meegid.2009.09.009. [ Links ]
Hotez PJ, Bottazzi ME, Franco-Paredes C, Ault SK, Periago MR. 2008. The neglected tropical diseases of latin America and the Caribbean: a review of disease burden and distribution and a roadmap for control and elimination. PLoS Neg Trop Dis. 2: 300. [ Links ]
Kimura M (1980) A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences. J Mol Evol 16: 111-120. doi: 10.1007/bf01731581. [ Links ]
Lent H, Wygodzinsky P (1979) Revision of the Triatominae (Hemiptera, Reduviidae), and significance as vectores of Chagas' disease. Bull Am Museum Nat Hist 163:125-520. [ Links ]
Lyman DF, Monteiro FA, Escalante AA, Cordon-Rosales C, Wesson DM, y col. (1999) Mitochondrial DNA sequence variation among triatomine vectors of Chagas' disease. Am J Trop Med Hyg 60: 377-386. [ Links ]
Miller S., Dykes D. & Polesky H. (1988). A simple salting out procedure for extracting DNA from human nucleated cells. Nucl. Acids. Res. 16:1215 -1217. [ Links ]
Monteiro FA, Barrett TV, Fitzpatrick S, Cordon-Rosales C, Feliciangeli D, y col. (2003) Molecular phylogeography of the Amazonian Chagas disease vectors Rhodnius prolixus and R. robustus. Mol Ecol 12: 997-1006. doi: 10.1046/j.1365-294x.2003.01802.x. [ Links ]
Noireau F, Flores R, Vargas F (1999) Trapping sylvatic Triatominae (Reduviidae) in hollow trees. Trans R SocTrop Med Hyg 93: 13-14. [ Links ]
Organización Mundial de la Salud. La enfermedad de Chagas (tripanosomiasis americana). Centro de prensa. Nota descriptiva N°340. 2012 [citado sept 2012]. Disponible en: http://www.who.int/mediacentre/factsheets/fs340/es/index.html [ Links ]
Organización Mundial de la Salud - 2. 2007. Reporte sobre la Enfermedad de Chagas. Reporte del grupo de trabajo científico sobre la enfermedad de Chagas. Buenos Aires; [citado sept 2012]. p. 104. Disponible en: http://whqlibdoc.who.int/hq/2007/TDR_SWG_09_spa.pdf [ Links ]
Porter CH, Collins FH (1996) Phylogeny of nearctic members of the Anopheles maculipennis species group derived from the D2 variable region of 28S ribosomal RNA. Mol Phylogenet Evol 6: 178-188. doi: 10.1006/mpev.1996.0070. [ Links ]
Santalla J., Oporto P., Espinoza, E., 2011. Primer brote reportado de la enfermedad de Chagas en la Amazonia Boliviana: reporte de 14 casos agudos por transmisión oral de Trypanosoma cruzi en Guayaramerín, Beni-Bolivia. BIOFARBO. jun. 2011,vol.19, no.1, p.52-58. [ Links ]
Schmunis G.A., Yadon Z.E. Chagas disease; 2010: a Latin American health problem becoming a world health problem. Acta Trop. 2010; 115: 14-21. 4. Moncayo A, Silveira AC. Current epidemi
Silveira A. O manejo da doença de Chagas como problema de Saúde Pública. 2007. En: La enfermedad de Chagas, a la puerta de los 100 años del conocimiento de una endemia americana ancestral. OPS; [citado sept 2012]. p. 119-27. Disponible en: http://www.mundosano.org/index.php/contenidos-principales/publicaciones-y-materiales/publicaciones/publicaciones-unitarias/ [ Links ]
Simone Frédérique Brenière, Claudia Aliaga, Etienne Waleckx, Rosio Buitrago,Renata Salas,Christian Barnabé, Michel Tiba-yrenc, and François Noireau. (2012). Genetic Characterization of Trypanosoma cruzi DTUs in Wild Triatoma infestans from Bolivia: Predominance of TcI. PLoS Negl Trop Dis. 2012 May; 6(5): e1650. [ Links ]
Tamura K, Peterson D, Peterson N, Stecher G, Nei M, y col. (2011) MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Mol Biol Evol 28: 2731-2739. doi: 10.1093/molbev/msr121. [ Links ]
WHO, World Health Organization Global health atlas. Accessed 2 March 2008. Available: http://www.who.int/globala-tlas/. 2007. [ Links ]
J. Med. Entomol. 35: 38-45. [ Links ]