Servicios Personalizados
Revista
Articulo
Indicadores
Links relacionados
Compartir
Revista CON-CIENCIA
versión impresa ISSN 2310-0265
Rev.Cs.Farm. y Bioq vol.1 no.1 La Paz oct. 2013
ARTICULOS DE INVESTIGACIONES FACULTATIVOS DOCENTE
Evaluación de un sistema optimizado de tipificación MIRU-VNTRs para estudios de epidemiología
molecular de Tuberculosis en Bolivia.
VASQUEZ MICHEL, ANETH1 LUNA BARRÓN, RUDDY2 TORRES, EMMA3
CORRESPONDENCIA: MSC. ANETH VASQUEZ MICHEL
TELF. 70103933/ 2786007 ANETHVASQUEZ@YAHOO.ES
1 Investigador Instituto de Investigaciones Fármaco Bioquímicas, Docente Investigador Instituto SELADIS, Facultad de Ciencias Farmacéuticas y Bioquímicas, Universidad Mayor de San Andrés
2 Perito Investigador del Centro de Investigación Genética del Instituto de Investigaciones Técnico Científicas de la Universidad Policial
3 Investigadora Adjunta del Centro de Investigación Genética del Instituto de Investigaciones Técnico Científicas de la Universidad Policial
Resumen
El Programa Nacional de Tuberculosis en Bolivia basa sus parámetros de control en métodos de epidemiología clásica; actualmente la Genética Molecular logra datos precisos mediante la aplicación de la epidemiología molecular, estudiado la dinámica de transmisión, permitiendo conocer fuentes y áreas afectadas en la transmisión, mejorando la comprensión de factores involucrados en la epidemiología de la Tuberculosis (TB). La Mycobacteriología molecular actualmente facilita el análisis y comparaciones entre bases de datos por medio de secuenciación automática.
El presente estudio pretende evaluar un sistema optimizado de tipificación MIRU-VNTRs (Unidades repetitivas interespa-ciadas de Mycobacterias) para estudios de epidemiología molecular de Tuberculosis en Bolivia.
Se colectaron 85 cepas de Mycobacte-rium tuberculosis de 8 departamentos de Bolivia. Se realizó la caracterización de estas cepas, mediante PCR multiplex, utilizando seis marcadores MIRU-VNTR específicos, considerando los complejos A (loci 4,26,40) y complejos B (loci 10,16,31) y se llevó a cabo la genotipificación por elec-troforésis capilar. Posteriormente se evaluó la variabilidad de genotipos entre cepas de los 8 departamentos y se establecieron las bases para la construcción de árboles filo-genéticos con las poblaciones estudiadas.
La tipificación de cepas de M. tuberculosis obtenidas y la estructura genética de las mismas, establecida a través de frecuencias alélicas de los MIRU analizados indica que existe una evolución independiente de las cepas de M. tuberculosis en los departamentos de Oruro, La Paz, Santa Cruz, Chuquisacay Potosí, siendo el departamento de Oruro el más relacionado a la cepa control (H37Rv), Por otro lado, en los departamentos de Cochabamba y Beni, las cepas muestran una relación evolutiva cercana, lo que es indicio de un proceso de migración entre estos departamentos.
El análisis filogenético basado en el perfil de resistencia a antibióticos de las cepas de M. tuberculosis muestra un proceso de evolución diferencial en cada una de las cepas evaluadas, siendo las cepas del departamento de La Paz las más relacionadas a las características de la cepas control.
PALABRAS CLAVE
Mycobacterium tuberculosis, Epidemiología Molecular, Unidades Repetitivas interespaciadas de mycobacterium (MIRU), PCR multiplex,, electroforesis capilar, árbol filogenético, evolución.
Abstract
The National Tuberculosis Programme in Bolivia control parameters are based on conventional epidemiological methods; Molecular Genetics currently manages ac-curate data through the application of molecular epidemiology, transmission dynam-ics studied, allowing knowledge sources and transmission affected areas, improving understanding of factors involved in the epidemiology of tuberculosis (TB). The molecular Mycobacteriology currently facilitates analysis and comparisons between data-bases using automated sequencing.
This study aims to evaluate a optimized system MIRU-VNTR typing (Mycobacteri-um interspersed repetitive units) for molecular epidemiology studies of tuberculosis in Bolivia.
We collected 85 Mycobacterium tuberculosis strains from 8 departments of Bolivia. We fulfill the characterization of these strains by multiplex PCR, using six markers specific MIRU-VNTR, considering the com-plex A (4,26,40 loci) and complex B (loci 10,16,31) and conducted genotyping by capillary electrophoresis. Then we assessed the variability of genotypes between strains of the 8 departments and established the basis for the construction of phylogenetic trees with the populations studied.
The genotype strains of M. tuberculosis obtained and their genetic structures established by MIRU allelicfrequencies analyzed, indicates an independent evolu-tion of strains of M. tuberculosis in the departments of Oruro, La Paz, Santa Cruz, Chuquisaca and Potosi, Oruro department being more related to the control strain (H37Rv), on the other hand, in the departments of Cochabamba and Beni, strains show a close evolutionary pattern, which is indicative of a migration between these departments.
Phylogenetic analysis based on the an-tibiotic resistance profile of M. tuberculosis strains shows a differential evolution pro-cess in each of the tested strains, being La Paz department strains mostly related to the characteristics of the control strains.
KEY WORDS
Mycobacterium tuberculosis, Molecular Epidemiology, Genotyping, Mycobacterium interspersed repetitive units (MIRU), multiplex PCR, alleles, capillary electrophoresis, phylogenetic tree, evolution.
INTRODUCCION
De acuerdo con últimos informes de la Organización Mundial de la Salud (OMS) 9,4 millones de personas en el mundo resultaron infectadas con TB entre los años 2010 -2011 y 1,7 millones murieron por esta causa, entre las que se incluyen 380.000 personas con TB asociada a VIH. La mayoría de los casos en las Américas se dan en Haití, Surinam, Bolivia, Guyana y Perú (OMS,2012-Wright, A. 2009). Bolivia es el tercer país más afectado en el continente Americano. De acuerdo a los datos de la OMS, el país presenta 20.200 casos de TB con una incidencia de 110 nuevos casos de baciloscopía positiva por 100.000 habitantes (Wright, A. 2009)
Por muchos años, el Programa Nacional de Tuberculosis en nuestro país se ha basado en la epidemiología clásica para determinar ciertos parámetros de control de este mal. Sin embargo, en la actualidad, con el advenimiento de la era de la Genética nuestro entendimiento acerca de la dinámica de transmisión ha sido aclarado gracias a la genotipificación de cepas de M. tuberculosis que permite profundizar significativamente aspectos de la transmisión de la enfermedad, además de conocer la diversidad genética de las cepas, estructura poblacional y sobretodo la distribución geográfica de las mismas, que adjunta a la epidemiología clásica constituyen una gran herramienta tanto para la vigilancia como para un control adecuado de la TB, sin olvidarnos que además estos estudios se aplican a la práctica clínica.
En los últimos años el aumento en las migraciones de países endémicos hacia países industrializados, así como el aumento de los casos de VIH, han contribuido a la re- emergencia de esta enfermedad en países en los cuales este estaba erradicado. Es por ello que el control eficiente de la enfermedad debe ser logrado no solo a través de sistemas de Vigilancia Nacional sino a través de sistemas de vigilancia epidemiológica internacionales capaces de monitorear con precisión las tendencias epidémicas a nivel global.
En este estudio proponemos la evaluación de un sistema optimizado para la genotipificación de M. tuberculosis mediante el análisis de loci genómicos que contienen secuencias con repeticiones en tándem (VNTRs). Usando este enfoque las cepas pueden ser tipificadas por códigos correspondientes al número de VNTRs en diferentes loci que contienen elementos genéticos llamados unidades repetitivas interespaciadas de mycobacterium (MIRU), creando bases de datos electrónicas que pueden ser fácilmente intercambiadas y compartidas entre diferentes laboratorios por lo cual se constituye en un método altamente apropiado para estudios epidemiológicos a nivel mundial.
MATERIALES Y MÉTODOS
Cepas utilizadas en el estudio
En el marco del acuerdo Interinstitucional entre la FCFB e INLASA; se solicito al Laboratorio Nacional de Referencia de Tuberculosis, se nos proporcione de la colección (cepario); 85 cepas disponibles cómo resultado de los aislamientos realizados durante los años 2009-2010. Las cepas fueron aisladas en medio de Lowenstein Jensen, procedentes de muestras clínicas enviadas para pruebas de susceptibilidad a antituberculosos, de diferentes departamentos de Bolivia.
Para poder contar con el número de cepas a estudiar, fue necesario realizar un artificio que garantice una selección aleatoria, cuya limitante se originaba en el número de cepas a "poder analizar" que estaba en función de la limitante económica, El número de muestras por departamento obedeció a alguna determinante aleatoria, la que a su vez se justifique con algún indicador y puedan hacerse estimaciones proporcionales a datos de incidencia.
El procesamiento de las muestras se llevo a cabo en los Laboratorios de la Unidad de Bioquímica Molecular del IIFB (Facultad de Ciencias Farmacéuticas y Bioquímicas) y en Laboratorios del Centro de Investigación Genética del Instituto de Investigaciones Técnico Científicas de la Universidad Policial.
Obtención de ADN genómico
El sistema de tipificación automatizado de MIRU-VNTR se desarrolló usando colonias de M. tuberculosis tratadas con ebullición y purificadas mediante la optimización de un método químico consistente en un kit de obtención de ADN genómico Wizard Genomic DNA Purification (PROMEGA).
Amplificación de MIRU- VNTRs - Multiplex PCR
Se realizó la caracterización de las cepas de M. tuberculosis, utilizando seis marcadores MIRU-VNTR específicos, considerándose los complejos A (loci 4,26,40) y complejos B (loci 10,16,31) de acuerdo al protocolo y cebadores propuestos por Supply et al. (Supply, 2001)
Los oligonucleótidos usados para la PCR correspondieron a las regiones flanqueantes del loci polimórfico MIRU-VNTR identificado en el genoma M.tuberculosis H37Rv
(Mazars,2001 - Van Embden,1993), la misma que se utilizó en este estudio para comparar los diferentes perfiles obtenidos, para cada mezcla de PCR multiplex, un primer de cada oligonucleótido fue marcado con un fluorocromo diferente.
El programa del termociclador para las dos reacciones multiplex fue el mismo. La PCR se llevo a cabo usando un termociclador (Hybaid, Ashford, Great Britain), comenzando con un paso de desnaturalización de 15 minutos a 95°, seguido de 35 ciclos de 1 min a 94°c, 1min a 59°C y 30 seg a 72°C. Los controles negativos estuvieron constituidos por mezclas de reacción sin ADN de mycobacterium (Van Embden, 1993).
Análisis de Minisatélites
Los fragmentos de MIRU VNTRs obtenidos fueron sometidos a electrofo-resis capilar en el Analizador Genético AB3130. Posteriormente, estos fragmentos, marcados con los fluoróforos FAM, TET y HEX, se analizaron a través del programa GeneMapper, lográndose la identificación de los alelos respectivos para cada marcador, en cada cepa. La asignación de alelos se realizó una vez determinados los pesos en comparación con el marcador de peso LIZ y la bibliografía descrita (Supply, 2001).
RESULTADOS
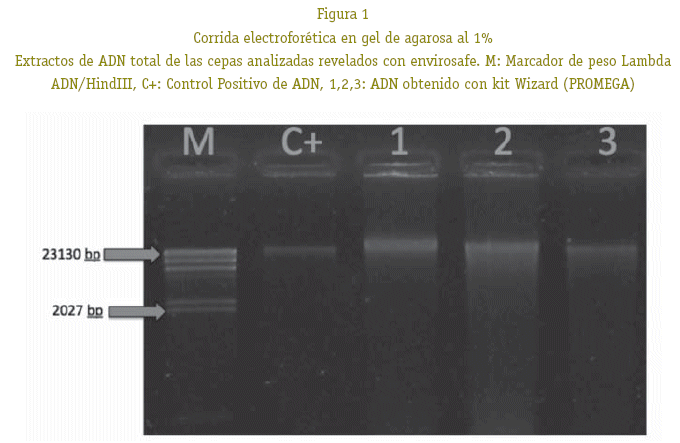
La optimización del método de extracción y purificación de ADN, Wizard Genomic Purification, permitió la obtención de fragmentos de ADN en calidad y cantidad suficiente para los ensayos subsecuentes (Fig.1).
Identificación de alelos de MIRUS VNTRs
La amplificación de los complejos A y B propuestos por Supply y colaboradores (Mazars,2001) permitió la obtención de 6 fragmentos específicos cuyo peso varía de cepa a cepa de acuerdo a sus características de polimorfismo (Fig. 2).
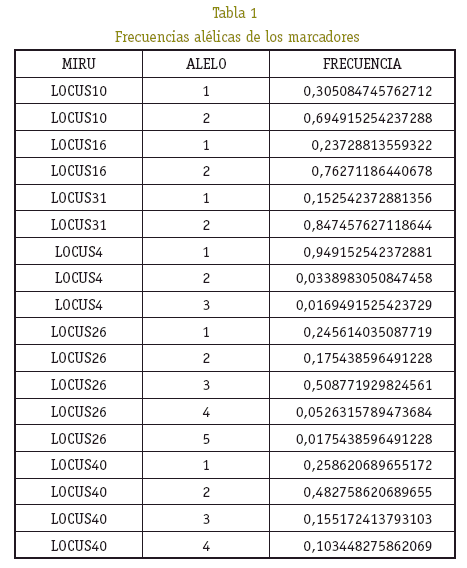
Así, el número de alelos obtenidos en la población total para los seis lo-cus evaluados fue de 18, presentando cada uno de ellos al menos dos variantes (Tabla 1).
Análisis de la estructura genética de los MIRU-VNTRs
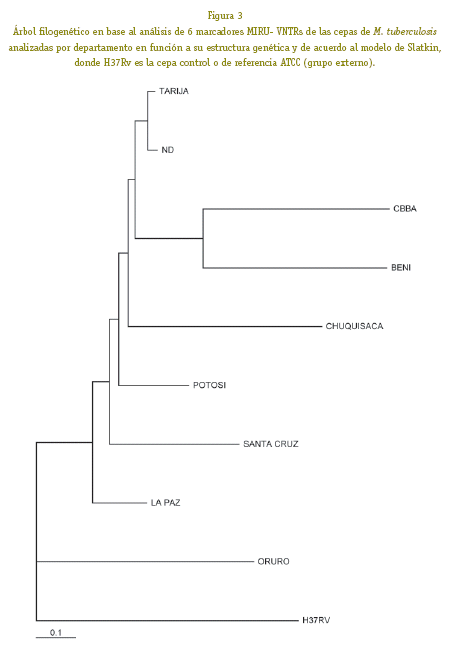
En esta investigación preliminar, el análisis de los alelos obtenidos en los seis marcadores VNTRs analizados en las 85 cepas estudiadas, permitió el establecimiento de su estructura genética a través de sus frecuencias alélicas, las cuáles, traducidas a distancia genética permitieron la construcción de un árbol filogenético a través del cuál es posible analizar la relación de las cepas entre los diferentes departamentos en función a la cepa control (H37Rv) (Fig. 3).
Por otro lado, analizando la relación filogenética de las cepas estudiadas en función a su perfil de resistencia a antibióticos (Fig.4), se observa que las cepas con menor resistencia se encuentran en el departamento de La Paz, mientras que las resistentes en Potosí y Cochabamba.
DISCUSIONES
La evaluación de un sistema optimizado de tipificación de 6 loci MIRU-VNTR propuesto en este estudio representa una aproximación bastante eficaz a la genotipificación de alta resolución de aislamientos de M. tuberculosis. Este utiliza una PCR multiplex con primers marcados con fluorocromos para analizar 2 dos grupos de 3 loci de MIRU-VNTR simultáneamente y provee un sistema automatizado necesario actualmente para la producción y manejo rápidos de los datos de genotipificación.
Datos bibliográficos revelan que la tipificación de MIRU-VNTR es 100% reproducible, sensible y específico para aislamientos del complejo M. tuberculosis, rendimiento que no ha sido alcanzado por ningún otro método utilizado con propósitos de tipificación, incluyendo RFLP IS6110, probado en las mismas condiciones(Cohn,1998). Es más, el poder de discriminación de la tipificación por MIRU-VNTR rindió mejor que otros métodos basados en RFLP u otros métodos basados en tipificación por PCR-IS6110- independiente (Cohn,1998- Frothingham,1998- Kremer,1999).
Enfocándonos plenamente a lo que se refiere a la identificación de los ale-los MIRU-VNTR, definimos que la menor frecuencia se presentó en el alelo 3 del locus 4, mientras que la mayor en el alelo 1 locus 4. En general, los seis loci estudiados son informativos para la identificación filogenética de los linajes de M. tuberculosis. Sin embargo, hay loci que parecen haber tenido una tasa evolutiva baja, o donde recientemente se están presentando procesos evolutivos, como es el caso de los locus 4 y 31, pues de no haberse reportado en ellos los alelos 1 y 2, respectivamente, estos no serían de gran utilidad para el establecimiento del lineas evolutivas (tabla 1).
Así, los alelos y las frecuencias encontradas muestran que los seis loci analizados presentan la suficiente variabilidad para distinguir cepas no relacionadas y estabilidad clonal para identificar una misma cepa y seguir la cadena de transmisión, aspectos de gran importancia de acuerdo a lo planteado por Supply et al. (Supply, 2001 - Supply,2006). Es en base a esto, que nuestro árbol (fig. 3), muestra que las cepas provenientes de la población orureña son las más relacionadas a la cepa control (cepa sensible a todo el espectro de antibióticos utilizados para el tratamiento de la tuberculosis), siendo su estructura genética única, pues no muestran relación con otras cepas del país, lo que podría indicar que en esta población, M. tuberculosis no está sometida a flujo génico o que no existen migrantes con otro genotipo de cepas.
Por otro lado, las cepas de La Paz, Santa Cruz, Potosí, Chuquisaca, Beni, Cochabamba y Tarija, se encuentran en un clado diferente al de Oruro, siendo las cepas genéticamente más diferentes, aquéllas pertenecientes al departamento de Tarija. Asimismo, las cepas de Beni y Cochabamba muestran una estrecha relación, lo que puede ser indicativo de un proceso de migración entre estos departamentos que llevó a una evolución conjunta de sus cepas de M. tuberculosis. En suma, en este árbol también es posible identificar a las cepas de La Paz en clado distinto al de los restantes departamentos. Sin embargo, es necesario mencionar que, al ser éste un estudio preliminar, pues solo involucra seis loci, la variación entre cepas puede incrementarse en más del 50%, lo que no necesariamente indicaría que los clados conformados se modificarían, pues ya se ha demostrado que los MIRU tienen una evolución convergente (Supply,2006).
En el análisis de la relación filogenética comparando la filogenia obtenida a partir del perfil de resistencia con aquélla establecida a través de la estructura genética dada por el análisis de MIRU-VNTRs (fig 4), se observó que no existe una relación a priori entre resistencia y evolución de las cepas en los distintos departamentos, lo que podría indicar que no existe un efecto de los antibióticos en la evolución a este nivel. Sin embargo, para rastrear los eventos evolutivos independientes en las distintas cepas en los 8 departamentos de Bolivia que puedan ligarse a estudios epidemiológicos y tal vez, más certeramente al perfil de resistencia de estas cepas, es necesaria la evaluación de estas poblaciones con aproximadamente 18 loci. Pues de acuerdo a (Supply et al - 2001- Monteserin, 2013) una comparación y evaluación precisa puede realizarse sólo a este nivel.
Finalmente concluimos que la aplicación del sistema MIRU-VNTRs en cepas de M. tuberculosis provenientes de ocho departamentos del país, permite la identificación y tipificación de las mismas, estableciendo sus linajes, mismos que permiten rastrear las cepas de mayor resistencia en el territorio nacional. Estos resultados demuestran la capacidad de nuestros laboratorios e investigadores para desarrollar y aplicar esta tecnología y la factibilidad, a través del Programa Nacional de Tuberculosis, para llevar a cabo este tipo de estudios que son de gran interés en nuestro medio, de manera que su análisis objetivo pueda coadyuvar al Programa en el establecimiento de políticas de control e información. Es en este sentido, que concluida exitosamente esta evaluación, se analizaron los 6 loci propuestos para la genotipificación de este microorganismo. Estudios futuros, mayor número de muestras y una tipificación de mayor complejidad son necesarios para establecer un patrón regionalizado, de manera que nos permita asignar el origen en aislamientos provenientes de pacientes.
AGRADECIMIENTOS
Agradecemos la colaboración del Dr. Antonio Flores Serna, por su apoyo logístico y coordinación administrativa en el proyecto del que es parte la presente investigación.
A la Dra. Mirtha Camacho y todo el equipo de profesionales y colaboradores del Laboratorio de Referencia Nacional de Control de la Tuberculosis (IN-LASA), por su apoyo incondicional al facilitarnos las cepas para este estudio.
A La Dra. María Teresa Alvarez responsable de la Unidad de Bioquímica Molecular del IIFB, donde se llevo a cabo el proyecto, por su amistad, lealtad y apoyo en todo momento
A las Lic. Silvana Limache Valderrama, Lic. Julia Molina Orihuela, Lic. Dina Quispe Mamani y la Lic.Daniela Arteaga Voight, quienes coadyuvaron al desarrollo del trabajo de laboratorio, así como al análisis y revisión de esta investigación.
REFERENCIAS
Cohn L. The use of restriction fragment length polymorphism (RFLP) analysisfor epidemiological studies of tuberculosis in developing countries. IntJTuber Lung Dis. 1998;2:16-26. [ Links ]
Frothingham, R., Meeker-O'Connell,WA. (1998). Genetic diversity in the Mycobacterium tuberculosis complex based on variable numbers of tandem DNA re-peats. Microbiology, 144 (pt5),1189-96. [ Links ]
Kremer, K. & Van Soolingen, D. (1999). Com-parison of methods based on different molecular epidemiological markers for typing of Mycobacterium tuberculosis complex strains: interlaboratory study of discriminatory power and reprodu-cibility. Journal of Clinical Microbiology, 37(8), 2607-18. [ Links ]
Mazars, E., Lesjean, S. & Banuls, A. High-re-solution minisatellite-based typing as a portable approach to global analysis of Mycobacterium tuberculosis molecular epidemiology.(2001) Proc Natl Acad Sci, 98(2), 1901-6. [ Links ]
Organizacion Mundial de la Salud (2012). Global tuberculosis control: epidemio-logy strategy financing. Geneva. [ Links ]
Supply, P., Allix, C., Lesjean, S., Savine, E., Kremer, K., Locht, C.(2001) Automated High-Throughput Genotyping for Study of Global Epidemiology of Mycobacterium tuberculosis Based on Mycobacte-rial Interspersed Repetitive Units. Journal of Clinical Microbiology, 2001: 39 (10), 3563-71. [ Links ]
Supply, P., Allix, C., Lesjean, S., Cardoso-Oelemann, M.,Ru sch-Gerdes, S., Willery, E.,.........Van Soolingen, D. (2006) Proposal for Standardization of Opti-mized Mycobacterial Interspersed Repetitive Unit-Variable-Number Tandem Repeat Typing of Mycobacterium tuberculosis. Journal of Clinical Microbiology, 44 (12), 4498 - 4510. [ Links ]
Van Embden J, Cave M, Crawford J, et al. (1993) Strain identification of Mycoba-terium tuberculosis by DNA fingerprin-ting: Recommendations for a standar-dized methodology. Journal of Clinical Microbiology, 31(2): 406-9. [ Links ]
Wright, A., Zignol, M., & Van Deun, A.(2009). Epidemiology of antituberculosis drug resistance: an updated analysis of the Global Project on Anti-Tuberculosis Drug Resistance Surveillance. Lancet, 373,1861-73. [ Links ]
Monteserin,J., Camacho M., Barrera, L. & Palomino, JC. (2013).Genotypes of Mycobacterium tuberculosis in patients at risk of drug resistance in Bolivia. Infection, Geneticsand Evolution, 17, 195-201. [ Links ]












 uBio
uBio