Journal of the Selva Andina Research Society
versión On-line ISSN 2072-9294
J. Selva Andina Res. Soc. vol.10 no.1 La Paz 2019
Artículo Original
Análisis de la diversidad de papas (Solanum spp.) con marcadores moleculares microsatélite de los distritos de Secclla y Santo Tomás de Pata (Huancavelica) y Santillana (Ayacucho)
Diversity analysis of potato (Solanum spp.) with microsatellite molecular markers of Secclla and Santo Tomas de Pata (Huancavelica) and Santillana (Ayacucho) districts
Tenorio-Bautista Saturnino Martín*, De La Cruz Marcos Ruggerths Neil
Facultad de Ciencias Agrarias. Universidad Nacional de Huancavelica. Jr. Victoria Garma No 275 y Jr. Hipólito Unánue No 280. Ciudad Universitaria de Paturpampa s/n. Paturpampa-Perú
Tel:+51 067451551
*Dirección de contacto: Facultad de Ciencias Agrarias. Universidad Nacional de Huancavelica. Jr. Victoria Garma No 275 y Jr. Hipólito Unánue No 280. Ciudad Universitaria de Paturpampa s/n. Paturpampa-Perú
Tel:+51 067451551
Saturnino Martín Tenorio-Bautista
E-mail address marte1901@hotmail.com
Historial del artículo.
Recibido julio, 2018.
Devuelto noviembre 2018
Aceptado diciembre, 2018.
Disponible en línea, febrero, 2019.
Resumen
La investigación se realizó con los objetivos de estudiar los perfiles moleculares de la diversidad genética, comparar la variabilidad y distancias genéticas de los marcadores moleculares de papas (Solanum spp.) colectados en campo de cultivo de agricultores de 12 comunidades de los distritos de Secclla, Santo Tomás de Pata y Santillana. Las 128 accesiones fueron cultivadas en bolsas polietileno con dos repeticiones cada una. El muestreo de los foliolos jóvenes se realizó a los 48 días después de la siembra. La extracción del ADN se procedió por el método de CTAB, copias de fragmentos del ADN se obtuvo mediante PCR, que fueron separados por PAGE. En el análisis molecular se empleó 8 pares de marcadores microsatélites. Los resultados se analizaron por los estadísticos, coeficiente Simple Matching, NTSYS 2.10p, método de agrupamiento UPGMA para estimar el polimorfismo y el AMOVA se calculó usando el programa Arlequín (versión 3.0). El PIC promedio fue 0.238, señala la existencia de polimorfismo. La distancia y similitud genética, las poblaciones que exhiben mayor relación genética comprenden, Santillana y Santo Tomás de Pata, denotando que las accesiones de estas poblaciones presentan mayores características en común. El AMOVA muestra que la variabilidad se encuentra dentro de poblaciones (91.55%), y la diferenciación genética entre las tres poblaciones es moderada (FST = 0.08448).
Palabras clave: Diversidad genética de Solanum spp. Microsatélites, Ayacucho-Huancavelica, polimorfismo de papa, marcadores SSR.
Abstract
The research was carried out with the objectives of studying the molecular profiles of genetic diversity, comparing the variability and genetic distances of the molecular markers of potatoes (Solanum spp.) which were collected in farmers' field from 12 communities of the Secclla, Santo Tomás de Pata and Santillana districts. The 128 accessions were cultivated in polyethylene bags with two repetitions each. The sampling of the young leaflets was carried out 48 days after sowing. DNA extraction was performed by CTAB method, copies of DNA fragments were obtained by PCR, which were separated by PAGE. In the molecular analysis, 8 pairs of microsatellite markers were used. The results were analyzed by the statistics, Simple Matching coefficient, NTSYS 2.10p, UPGMA grouping method to estímate the polymorphism and the AMOVA was calculated using the Arlequín program (versión 3.0). The average PIC was 0.238, indicating the existence of polymorphism. The distance and genetic similarity, the populations that exhibit the greatest genetic relationship include Santillana and Santo Tomás de Pata, denoting that the accessions of these populations have greater common characteristics. The AMOVA shows that the variability is within populations (91.55%), and the genetic differentiation between the three populations is modérate (FST = 0.08448).
Key words: Genetic diversity of Solanum spp., Microsatélite, Ayacucho-Huancavelica, potato polymorphism, SSR markers.
Introducción
La papa es cultivada en los Andes desde más de siete mil años, según investigaciones recientes, el origen de la papa, - Solanum tuberosum-, se centra en la parte norte de lago Titicaca.1 Esta región fue testigo silencioso del paso de culturas andinas, que forjaron a los primeros agricultores, combinando características genéticas y variabilidad ambiental, pudieron domesticar a la papa silvestre (PS), logrando una gran colección que dio lugar a variedades de amplia adaptabilidad ambiental, que hoy se constituyen en un alimento básico, y el cuarto alimento a nivel mundial.2
En el catálogo de variedades de papa nativa (PN) de Huancavelica-Perú, describen un total de 144 variedades que representan una cuarta parte del total de variedades de PN de esta región, muestra que mantiene una impresionante diversidad de variedades nativas, estudios preliminares indican que cuenta con 500 a 600 variedades.3
Investigaciones sobre el origen de las papas domesticadas señalan que hubo discrepancias entre los investigadores que abordaron el tema, principalmente postularon dos teorías: La primera sostiene que es posible la existencia de dos centros de domesticación, como fue descrito por Vavilov en 19514 que sostiene que deben existir dos centros de origen, uno principal, situado en el macizo andino peruano-boliviano y el otro secundario, que comprende el sur de Chile y Chiloé. La segunda teoría planteada por Spooner et al.1, al analizar el perfil genético de las papas cultivadas y especies silvestres relacionadas mediante AFLP nucleares, llegaron a la conclusión que la PN cultivada tiene un origen único o monofilético en una región extensa del sur de Perú y norte de Bolivia, su cultivo se dispersó por el norte hasta Colombia y por el sur hasta Chile y Chiloé. En los últimos años, estudios genéticos, bioquímicos y moleculares han apoyado la teoría de la existencia de un solo centro de domesticación ubicado en la región peruana-boliviana.4,1
Los marcadores moleculares presentan ventajas que permiten revelar diferencias existentes entre accesiones como producto de la variabilidad en el proceso evolutivo, que permitan asociar variaciones genómicas, características heredables de interés agronómico y de calidad, que resulten importantes dentro de un programa de mejoramiento. Los microsatélites ofrecen varias ventajas, entre ellas su comportamiento codominante, multialelismo, nivel de polimorfismo, simplicidad de trabajo, bajo requerimiento de cantidad y calidad de ADN, distribución en el genoma y reproducibilidad.5,6
Muchas de las variedades de PN han sido identificadas por las características morfológicas, citogenéticas y agronómicas, entre otras cualidades, para corroborar estos estudios, se debe identificar a través de los marcadores moleculares microsatélites que representan una de las tecnologías más importantes en el análisis de los genomas, permitiendo asociar variaciones genómicas, características heredables de interés agronómico y de calidad que permitan dar respuesta a la creciente exigencia de nuevas variedades, que resulten importantes dentro de un programa de mejoramiento con resistencia a heladas, sequías, cambio climático, plagas y enfermedades, alta producción, aceptación gastronómica y altos niveles nutrivos.7 Otro aspecto importante al momento de elegir una técnica molecular, es su capacidad para trabajar con gran cantidad de datos y establecer, mediante análisis estadísticos estimaciones de diversidad en y dentro las poblaciones en estudio.8
Los objetivos del presente trabajo fueron: Determinar los perfiles moleculares de la diversidad genética, comparar la variabilidad, estimar las distancias genéticas entre los marcadores moleculares microsatélites de la diversidad genética y establecer las variaciones moleculares entre las poblaciones a través del AMOVA de papas (Solanum spp.), procedentes de los distritos de Secclla, Santo Tomás de Pata y Santillana.
Materiales y métodos
En los meses de mayo y junio de 2017, época de cosecha, fueron colectados entre 6 a 8 tubérculos de cada variedad en campo de agricultores, fueron guardadas en bolsas de papel previamente codificados y puestos los nombres de cada accesión, para la siembra se escogió tubérculos sanos que fueron fotografiados con cámara semiprofesional (NIKON D7"100 AF-SDX NIKKOR 18-105mm) antes de la siembra (figura 1).
La colección total procedió de doce comunidades productoras de PN, cuatro comunidades por cada distrito. La investigación se llevó a cabo con 128 accesiones de papas (Solanum spp.) procedentes del distrito de Santillana (49), Santo de Tomás de Pata (40) y Secclla (39).
La siembra de las 128 accesiones se realizó el 18 de julio de 2017 en bolsas polietileno con dos repeticiones en condiciones similares, en la localidad de Acuchimay distrito de San Juan Bautista provincia de Huamanga región Ayacucho, entre las coordenadas geográficas 13°10'6.62" Sur, 74°13'21.98" Oeste a 2791 msnm, temperatura media mensual de julio y agosto 2017 de 16.3 °C. La colección de muestras para la extracción de ADN se realizó a los 48 días después de la siembra (03-09-17). Una vez colectadas las hojas, fueron empaquetadas en papel toalla debidamente rotuladas y conservadas a una temperatura de 4 °C, puestas en caja de tecnopor se transportaron al laboratorio de Biología Molecular de la Universidad Nacional de San Cristóbal de Huamanga (UNSCH). La extracción del ADN se realizó el 04 de setiembre de 2017.
La extracción de ADN se realizó de hojas tiernas, frescas, mediante el método de CTAB.9 La calidad y concentración de ADN se determinó a través de la fluorescencia producida por el complejo Bromuro de Etidio-ADN, mediante uso de luz ultravioleta, se visualizó en foto documentador UVsolo TS imaging system. Para cuantificar el ADN total se utilizó espectrofotómetro NANODROP (ND-1000), a 260 y 280 nm para evaluar su pureza, luego se realizó diluciones con agua libre de nucleasas, hasta alcanzar una concentración final de 5 ng/![]() l de ADN, y almacenarlas a -20 °C.
l de ADN, y almacenarlas a -20 °C.
Antes de iniciar con el PCR se realizó una gradiente de temperatura de 48-62 °C, cada primer o marcadores microsatélites fue probado a cinco temperaturas diferentes de anillamiento (annealing) para cada primer microsatélite de acuerdo a las accesiones de papa, verificando el tamaño esperado en pares de bases (pb) en los productos microsatelítales amplificados. En base a la calidad de los productos de amplificación obtenidos y presentar mayor polimorfismo, se seleccionaron 8 iniciadores microsatelites para ser utilizados en el análisis con todas las accesiones10, los primers seleccionados se detalla a continuación.

La reacción en cadena de la polimerasa (PCR) se llevó a cabo en un termociclador Eppendorf, la electroforesis en geles de poliacrilamida (PAGE), se preparó 100 mL de solución de poliacrilamida al 6 % (acrilamida, bis-acrilamida, 19:1), urea 5 M. Se agregó 545 ![]() L de persulfato de amonio al 10 % y 109
L de persulfato de amonio al 10 % y 109 ![]() L de TEMED. La solución se colocó entre dos placas de vidrio de la cámara, siendo el tamaño del gel 28 x 40 cm, y su grosor 0.4 mm, se colocó el peine, una vez solidificado el gel se extrajo el peine para formar los pocillos, con una jeringa hipodérmica se cargaron 6
L de TEMED. La solución se colocó entre dos placas de vidrio de la cámara, siendo el tamaño del gel 28 x 40 cm, y su grosor 0.4 mm, se colocó el peine, una vez solidificado el gel se extrajo el peine para formar los pocillos, con una jeringa hipodérmica se cargaron 6 ![]() L corridos en una cámara vertical de electroforesis de secuenciamiento, usando como gradiente de corrida TBE 1X y 0.5X, para el ánodo y cátodo, respectivamente. Una vez terminada se sometió a la fijación, tinción y revelado del gel.
L corridos en una cámara vertical de electroforesis de secuenciamiento, usando como gradiente de corrida TBE 1X y 0.5X, para el ánodo y cátodo, respectivamente. Una vez terminada se sometió a la fijación, tinción y revelado del gel.
Las bandas reveladas fueron registradas visualmente con ayuda de un transiluminador de luz blanca, se leyeron las bandas consecutivamente empezando desde la primera accesión del primer al final del carril. Las bandas se numeraron en orden ascendente, tomando como referencia su respectivo peso molecular, y se codificaron en una matriz binaria donde "1" indicó presencia y "0" ausencia.
El patrón para determiner el tamaño de bandas se utilizó "GeneRuler 100bp Plus DNA Ladder #SM0321", según los primers seleccionados diseñado para dimensionamiento y cuantificación aproximada de anchos. Rango de ADN de doble cadena en PAGE. La escalera está compuesta por catorce fragmentos de ADN individuales purificados por cromatografía (en pares de bases): 3000, 2000, 1500, 1200, 1000, 900, 800, 700, 600, 500, 400, 300, 200, 100. Que se adquiere junto con los primers de establecimientos de genética molecular o importados (GeneRuler 100 bp Plus DNA Ladder, #SM0321 2012)12.
Los datos se analizaron con los paquetes estadísticos de coeficiente simple matching, con NTSYS 2.10p. El dendrograma de relaciones entre las variédades se construyó usando coeficientes de similitud de Jaccard y el método de agrupamiento UPGMA. Para estimar parámetros de polimorfismo a nivel de poblaciones con la matriz de datos de presencia/ausencia se usó el programa POPGEN32.13,14 Se cuantificó la variación presente entre las poblaciones, mediante el AMOVA (por sus siglas en inglés Analysis of Molecular Variance), usando el programa Arlequin (versión 3.0) con la opción "pairwise differences" (diferencias por parejas). En este sofware, los datos moleculares binarios se consignaron como locus.15 El coeficiente de diferenciación genética (FST) cuantifica la diferenciación u homogenización entre distintas poblaciones en el cultivo de la papa.13
Resultados
Las bandas reveladas de las 128 muestras como resultado del proceso de tinción fueron registradas visualmente e ingresados a una hoja de cálculo Microsoft Excel 2010, los datos se analizaron en los paquetes estadísticos, para estimar parámetros de polimorfismo (PIC) a nivel de poblaciones con la matriz de datos de presencia/ausencia de bandas, distancia y similitud genética, el dendrograma y AMOVA.
Discusión
La gran diversidad de PN en las zonas de estudio reportadas se debe a la presencia de reproducción sexual (semilla botánica) y asexual (tubérculos semilla), que facilita el flujo de genes influyendo a la diversidad genética. Por experiencias profesionales la diversidad de papas silvestres en la Sierra del Perú (Huancavelica y Ayacucho) es endémica, la teoría que reconoce la existencia de un centro de domesticación comprendida por la región sur del Perú y norte de Bolivia, es la más aceptada por ser apoyada por estudios genéticos, bioquímicos y moleculares.4,1
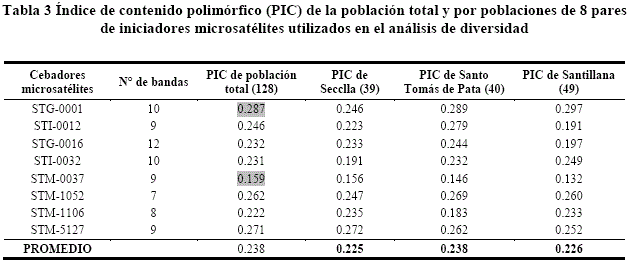
El más alto valor del iniciador STG-0001 fue 0.287, cuyo valor indica que el PIC es informativo, que señala la existencia de variabilidad genética entre las 128 accesiones, mientras el cebador STM-0037 es menos informativo de polimorfismo, con valor 0.159, en relación al promedio 0.238 siendo este un indicador del polimorfismo (tabla 3). El PIC es importante porque estima la eficiencia de cada marcador para determinar la variabilidad genética (polimorfismo) en un locus dado en cada una de las accesiones que se evaluó se encuentra las diferencias genéticas entre las accesiones en estudio. Cadima et al.16, reportaron valores del PIC en rangos 0.13 a 0.87. Cadima-Fuentes et al.17 señalan valores de PIC por microsatélite de 0.13 a 0.85, nuestros resultados concuerdan con las investigaciones referidas.
Mientras Nuñez-Linares18 en análisis de la variabilidad genética de ocas cultivadas (Oxalis tuberosa Mol.) de la región Cajamarca, obtubieron valores de PIC que varían de 0.11 a 0.17, en tanto Soto et al.19 al realizar una investigación con 79 variedades de PN de cinco regiones del Perú (Ayacucho, Cajamarca, Cusco, Huancavelica y Puno) reportan PIC entre 0.55-0.85, y Ghebreslassie et al.20 PIC 0.51-0.91, valores que no concuerdan con nuestros resultados. Las poblaciones que presentan mayor relación genética, comprenden a Santillana y Santo Tomás de Pata siendo la distancia y similitud genética de 0.015 y 0.986, respectivamente. Las accesiones de papa de estas poblaciones presentan mayores características genéticas en común. Santo Tomás de Pata y Secclla presentan distancias y similitudes de 0.025 y 0.976, y entre poblaciones de Santillana y Secclla alcanzando distancia y similitud de 0.024 y 0.975.
Las accesiones de Secclla son muy diferentes genéticamente (tabla 4). Los estudios evolutivos realizados concuerdan con los resultados obtenidos4,1. Pérez Diaz21 del total de 273 accesiones de papa evaluados con siete iniciadores moleculares calculando similitud describió en cuatro poblaciones diferencias, el grupo Colección alcanzó 94.2% (0.942), Chiloé con 92.6% (0.926), Chonos tuvo 97.6% (0.976) y Europea, 95% (0.95). Otros autores reportan resultados de coeficiente de similitud genética promedio 98.35% (0.9835).22 También determinaron el índice de matriz de distancia genética que oscila entre 0.011 y 0.048.20 Valores que corroboran los resultados obtenidos. Asi valores entre 0.112-0.179, ligeramente superiores a los descritos.23
La importancia de determinar la similitud genética entre individuos de una población, es para realizar correctamente los análisis de agrupamiento y diversidad. No existe un coeficiente de similitud universal. El análisis de agrupamiento de relaciones entre accesiones de papa se realizó utilizando el coeficiente de similitud de Simple Matching y el método de agrupamiento de UPGMA (Unweighted Pair Group Method using Arithmetic Averages) del análisis de 74 marcadores microsatélites, se agruparon en once grupos las 128 accesiones de papas (Solanum spp.) procedentes de los distritos de Santillana provincia de Huanta región Ayacucho y de los distritos de Santo Tomás de Pata y Secclla de la provincia de Angaraes región Huancavelica, a 0.70 unidades ultramétricas. La variabilidad genética de las accesiones analizadas manifiesta proceder de poblaciones polimórficas con tendencia de ser estrechos formando grupos familiares por distrito y dentro de los distritos, se tuvo solamente dos duplicados (figura 2). El primer grupo formado por trece accesiones, la primera accesión corresponde a Santillana (SA) y doce restantes a Santo Tomás de Pata (TP). En el grupo II señalamos 87 accesiones, esto es las primeras 25 variedades están distribuidas por las tres poblaciones, del 26 al 39 forman un grupo local las papas de Santillana, del 40 al 51 prevalecen de Secclla (SE), del 52 al 69 destacan nuevamente de Santillana, del 70 al 76 sobresalen de Secclla, del 77 al 87 predominan papas procedentes de Santo Tomás de Pata. Se revela que se agrupan en grupos locales con algunas accesiones que probablemente hayan sido introducidos dentro de estas poblaciones. El grupo III predominantemente es representado por accesiones de Secclla, en los grupos del IV al IX sobresalen Santo Tomás de Pata. Finalmente en los grupos X y XI destacan papas de Secclla, y que están las papas amargas y semisilvestres que se supone que vienen a ser los ancestros de las papas nativas y mejoradas. El dendrograma representa la diversidad del individuo con respecto a los demás miembros de la muestra. Ponce Almeri et al. 24 al evaluar el agrupamiento desde el punto de vista regional, reportó una tendencia moderada de agrupamiento de acuerdo a la región geográfica de procedencia del centro, sur y norte del Perú, señalan que las variedades mejoradas se agrupaban en un extremo y ciertas entradas que se sobrepasaban con el grupo de las nativas. Con microsatélites, en un dendrograma se describe una estructura genética clara y definida, se pueden observar que la población se divide básicamente en dos grandes grupos A y B, a un coeficiente de similaridad de 0.26, el análisis de conglomerados permitió visualizar las diferencias y relaciones genéticas entre especies y dentro de las especies en cuanto a diversidad16. Alvarado Aliaga25, señala un dato muy importante que coincide con los resultados obtenidos, de que en la parte final del dendrograma se agrupan los cultivares de papas amargas y al ser agrupadas las ponen aisladas en la parte final, siendo estas las más distantes en similaridad. Orona Castro26, menciona que las relaciones genéticas observadas en el dendrogramsa para cada sistema de marcador, en ambas técnicas, las variedades permanecieron estrechamente relacionadas bajo los dos sistemas de marcadores. En la técnica de SSR, una mejor discriminación entre genotipos, la línea procedente de Argentina fue la más divergente. Milbourne et al.27, manifiesta que la técnica de SSR es muy eficiente en este tipo de investigaciones, debido a que es capaz de identificar variación en sitios determinados. Spooner et al.28, los cultivares modernos se ubican en un extremo del dendrograma. Todos los autores mencionandos coinciden con los resultados obtenidos.
La AMOVA una técnica utilizada para estudiar las variaciones moleculares en el interior de una especie, en el presente trabajo se utilizó para comprobar el porcentaje entre poblaciones y variación individual dentro de las poblaciones, fueron tratadas a través del análisis de varianza molecular (AMOVA).15 Las muestras se recogieron de tres distritos geográficamente distintos, este método señala que existe una variación entre los integrantes de cada población (91.55%), que indica que existe grupos familiares o subpoblaciones en el interior de cada población y el 8.45% restante se debe al componente de varianza genética entre poblaciones (tabla 5), el índice de fijación (FST) es de 0.08448 (tabla 2), indica que la diferenciación genética es moderada entre las tres poblaciones. Estos resultados refuerzan con la distancia y similitud genética, entre las poblaciones donde se indica que las tres poblaciones presentan mayores características en común, además los resultados obtenidos, señalan que hay evidencia sobre la existencia de la diversidad genética de Solanum spp., variabilidad, distancia y similitud genética entre los marcadores moleculares microsatélites. Ponce Almeri et al.24, respalda nuestros resultados, al agrupar papas de distintas localidades por regiones geográficas, la principal fuente de variación genética (89.47%) sea debido a las diferencias dentro de las localidades de cada región. De Han et al.29, trabajando con PN de Huancavelica, al determinar el AMOVA entre dos regiones geográficamente distanciadas del centro y sur de Huancavelica, señala que la principal fuente de variación genética estaba dentro de las poblaciones locales. Onamu & Legaría30, en estudio de diversidad genética entre variedades de papa (S. tuberosum L.) cultivadas en México, en el AMOVA indican que la mayor variación se debe a poblaciones dentro de grupos, lo que significa que entre variedades hay diferencias. Anoumaa et al.23, al analizar la diversidad genética papa S. tuberosum L., en el AMOVA 97% de variación se debió a la diversidad dentro de grupos. Ghebreslassie et al.20, en la diversidad genética en cultivares de patata (S. tuberosum) con SSR, el 92% de la variación fue observada dentro de la población. Huaraca Meza31, en Lepidium meyenii Walp), con cinco combinaciones de iniciadores de AFLP, con AMOVA en 95.71%, la variación genética se encuentra entre las accesiones dentro de los distritos.
En síntesis del presente trabajo de investigación se deduce: que los perfiles moleculares varían de acuerdo a los iniciadores microsatélites que señalan mayor o menor polimorfismo según poblaciones, porque las muestras son diferentes en accesiones y ploidia. La técnica de marcadores moleculares microsatélites son eficientes en el análisis de la diversidad genética de accesiones de papas (Solanum spp.), formando en cada población de estudio grupos locales de variedades semisilvestres, nativas y las mejoradas. Las poblaciones que presentan mayor relación genética comprenden a Santillana-Santo Tomás de Pata, cuya distancia genética es 0.015, las accesiones de estas poblaciones presentan mayores características en común; las asociaciones de Santo Tomás de Pata-Secclla y Santillana-Secclla, presentan distancias genéticas promedio de 0.0245 que muestran diferencias genéticamente. La AMOVA determinó las variaciones moleculares en las poblaciones, el 91.55% de variación ocurre dentro de las poblaciones y el 8.45% restante se debe al componente de varianza genética entre poblaciones. El cálculo del estadístico FST=0.08448 (grado de diferenciación génica entre las poblaciones) muestra que existe una diferenciación genética moderada entre poblaciones.
Conflictos de intereses
Los autores expresan que no existen conflictos de interés
Agradecimientos
Nuestro agradecimiento a la Universidad Nacional de Huancavelica, Universidad Nacional de San Cristóbal de Huamanga, por cedernos sus instalaciones, equipos y materiales de laboratorio en la parte experimental del presente trabajo y a los agricultores de las zonas de estudio que conservan insitu la diversidad genética de las papas (Solanum spp.).
Aspectos éticos
La aprobación de la investigación por el Comité de Ética, Vicerrectorado de Investigación de la Universidad Nacional de Huancavelica - Perú, siguió las pautas establecidas para este comité.
Literatura citada
1. Spooner DM, Mcleant K, Ramsay G, Waugh R, Bryan GJ. A single domestication for potato based on multilocus amplified fragment length polymorphism genotyping. Proc Natl Acad Sci USA 2005;102(41): 14694-9. DOI: https://dx.doi.org/10.1073/pnas.0507400102 [ Links ]
2. Iriarte V, Condori B, Parapo D, Acuña D. Catálogo etnobotánica de papas nativas del Altiplano Norte de La Paz-Bolivia [Internet]. Cochabamba: Fundación Proinpa; 2009. 142 p. Recuperado a partir de: https://www.proinpa.org/tic/index.php?option=com_content&view=article&id=13:catalogo-etnobotanico-de-papas-nativas-del-altiplano-norte-de-la-paz-bolivia&catid=15&Itemid=115 [ Links ]
3. Centro Internacional de la Papa & Federación Departamental de Comunidades Campesinas de Huancavelica. Catálogo de variedades de papa de Huancavelica-Perú [Internet]. Huancavelica: Centro Internacional de la Papa; 2006. Recuperado a partir de: https://cipotato.org/wp-content/uploads/2014/08/_003524.pdf [ Links ]
4. Hawkes JG. Origins of cultivated potatoes and species relationships. In: Bradshaw JE, Mackay GR, editors. Potato genetics. CAB International: Oxford, UK; 1994. p. 3-42. Disponible en: http://agris.fao.org/agris-search/search.do?_recordID=GB9416538 [ Links ]
5. Colman SL, Monti MC, Divito SB, Digilio A, Feingold SE. Marcadores funcionales asociados al endulzamiento inducido por frío en papas nativas de Argentina. Rev Latinoam Papa 2009;15(1):61-5 [ Links ]
6. Navarro C, Bolaños LC, Lagos TC. Caracterización morfoagronómica y molecular de 19 genotipos de papa guata y chaucha (Solanum tuberosum L. y Solanum phureja Juz Et Buk) cultivados en el departamento de Nariño. Rev Cienc Agr 2010;27(1):27-39. [ Links ]
7. Ghislain M, Rodríguez F, Núñez J, Naik P, Huamán Z, Bonierbale M. Comparison of genetic diversity of potato varieties from India and South America [Internet]. San Diego: Poster presented at Plant and Animal Genome; 2001. Recuperado a partir de: http://www.cipotato.org [ Links ]
8. Roca Infante LA. Análisis de la diversidad genética de papas nativas de la zona suroeste del departamento de Junín mediante el uso de marcadores moleculares microsatélites [tesis de Licenciatura]. [Lima]: Universidad Nacional Agraria La Molina; 2015. Recuperado a partir de: http://repositorio.lamolina.edu.pe/handle/UNALM/1885 [ Links ]
9. Doyle J, Doyle JL. Isolation of DNA from small amounts of plant tissues. BRL Focus, modified at the Forest Biotechnology Lab at NCSU 1990;12:13-5. [ Links ]
10. Oetting WS, Lee HK, Flanders DJ, Wiesner GL, Sellers TA, King RA. Linkage analysis with multiplexed short tándem repeat polymorphisms using infrared fluorescence and M13 tailed primers. Genomics 1995;30(3):450-8. DOI; https://dx.doi/org/10.1006/geno.1995.1264 [ Links ]
11. Ghislain M, Nuñez J, del Rosario Herrera M, Pignataro J, Guzman F, Bonierbale M. et al. Robust and highly informative microsatellite-based genetic identity kit for potato. Mol Breeding 2009;23(3):377-83. DOI: https://dx.doi.org/10.1007/s11032/008/9240/0 [ Links ]
12. Thermo Scientific. GeneRuler 100 bp Plus DNA Ladder [Internet]. Product infomation. Recuperado a partir de: http://www.molecularlab.it/public/data/sar99/2015715164227_MAN0013008_GeneRuler_100bp_Plus_DNALadder_50ug_UG.pdf [ Links ]
13. Nei M. Estimation of average heterozygosity and genetic distance from a small number of individuals. Genetics 1978;89(3):583-90. [ Links ]
14. Hart D, Clark A. Molecular population genetics. En: Avise J, Hamrick J, editors. Principles of population genetics. Sinauer Associates: Massachusetts; 1997. p.423. [ Links ]
15. Excoffier L, Laval G, Schneider S. Arlequin (versión 3.0): An integrated software package for population genetics data analysis. Evol Bioinform Online 2005;1:47-50. [ Links ]
16. Cadima A, Veramendi S, Gabriel J. Uso de marcadores moleculares microsatélite para el análisis de la diversidad genética de papa nativa de Bolivia. J Selva Andina Res Soc 2013;4(1):18-30. [ Links ]
17. Cadima-Fuentes X, Veramendi S, Ángulo A. Comparación de niveles de diversidad genética de papa entre Centros de Agrobiodiversidad y la Colección Nacional de Bolivia. Rev Latinoam Papa 2017;21(1):73-92. [ Links ]
18. Nuñez Linares E. Análisis de la variabilidad genética de las ocas cultivadas (Oxalis tuberosa Mol.) de la región Cajamarca [tesis de Licenciatura]. [Lima]: Universidad Nacional Agraria La Molina; 2015. Recuperado a partir de: http://repositorio.lamolina.edu.pe/handle/UNALM/1885 [ Links ]
19. Soto J, Medina T, Aquino Y, Estrada R. Diversidad genética de papas nativas (Solanum spp.) conservados en cultivares nativos del Perú. Rev Perú Biol 2013;20(3):215-22. [ Links ]
20. Ghebreslassie B, Githiri S, Mehari T, Kasili R, Ghislain M, Magembe E. Genetic diversity assessment of Farmers and Improved potato (Solanum tuberosum) Cultivars from Eritrea using Simple Sequence Repeat (SSR) markers. Afr J Biotechnol 2016;15(35):1883-91. DOI: http://dx.doi.org/10.5897/AJB2016.15237 [ Links ]
21. Pérez Diaz JR. Evaluación de la diversidad genética de papas nativas (Solanum tuberosum L. ssp. tuberosum Hawkes) silvestres y cultivadas del sur de Chile, mediante el uso de marcadores microsatélites [tesis de Licenciatura]. [Lima]: Universidad Austral de Chile; 2004. Recuperado a partir de: http://cybertesis.uach.cl/tesis/uach/2004/fap438e/pdf/fap438e.pdf [ Links ]
22. Hong L, Huachun G. Using SSR to evalúate the genetic diversity of potato cultivars from Yunnan province (SW China). Acta Biol Cracov Ser Bot 2014;56(1):16-27. DOI: http://dx.doi.org/10.2478/abcsb-2014-0003 [ Links ]
23. Anoumaa M, Yao NK, Kouam EB, Kanmegne G, Machuka E, Osama SK, et al. Genetic diversity and core collection for potato (Solanum tuberosum L.) cultivars from Cameroon as Revealed by SSR markers. Am J Potato Res 2017;94:449-63. DOI: https://dx.doi.org/10.1007/s12230-017-9584-2 [ Links ]
24. Ponce Almeri R, Herrera M, Ramírez P. Caracterización molecular de las variedades de papas cultivadas (Solanum spp.) más importantes del Perú mediante el uso de microsatélites [tesis de Licenciatura]. [Lima]: Universidad Nacional Mayor de San Marcos; 2013. Recuperado a partir de: http://cybertesis.unmsm.edu.pe/handle/cybertesis/913 [ Links ]
25. Alvarado Aliaga CA. Caracterización fenotípica y molecular de la diversidad genética de papas cultivadas por su tolerancia al endulzamiento en frío [tesis de Licenciatura]. [Lima]: Universidad Nacional Mayor de San Marcos; 2008. Recuperado a partir de: http://cybertesis.unmsm.edu.pe/handle/cybertesis/913 [ Links ]
26. Orona Castro F. Caracterización de germoplasma sobresaliente de papa Solanum tuberosum L. en México mediante RADP y SSR [tesis Doctoral]. [Nuava León]: Universidad Autónoma de Nuevo León; 2004. Recuperado a partir de: http://eprints.uanl.mx/5818/ [ Links ]
27. Milbourne D, Meyer R, Bradshaw JE, Baird E, Bonar N, Provan J, et al. Comparison of PCR-based markers systems for the analysis of genetics relationships in cultivated potato. Mol Breed 1997;3:127-36. DOI: https://dx.doi.org/10.1023/A:1009633005390 [ Links ]
28. Spooner DM, Nuñez J, Trujillo G, Herrera MR, Guzman F, Ghislain M. Extensive simple sequence repeat genotyping of potato landraces supports a major reevaluation of their gene pool structure and classification. Proc Natl Acad Sci USA 2007;104(49):19398-403. DOI: https://dx.doi.org/10.1073/pnas.0709796104 [ Links ]
29. De Haan S, Núñez J, Bonierbale M, Ghislain M. Multilevel Agrobiodiversity and conservation of andean potatoes in central Perú: Species morphological, genetic, and spatial diversity. Mt Res Dev 2010;30:222-31. DOI: https://dx.doi.org/10.1659/mrd-journal-d-10-00020.1 [ Links ]
30. Onamu R, Legaría Solano JP. Diversidad genética entre variedades de papa (Solanum tuberosum L.) cultivadas en México usando marcadores RAPD e ISSR. Rev Mex Cienc Agríc 2014;5(4):561-75. [ Links ]
31. Huaraca Meza F. Variabilidad genetica de maca (Lepidium meyenii Walp.) de la Meseta de Bombón-Junín, Perú [tesis de Maestría]. [Lima]: Universidad Nacional Agraria La Molina; 2015. Recuperado a partir de: http://repositorio._Lamolina.edu.pe/handle/UNALM/2834 [ Links ]












 uBio
uBio