Journal of the Selva Andina Research Society
versión On-line ISSN 2072-9294
J. Selva Andina Res. Soc. vol.8 no.1 La Paz 2017
Comunicación corta
Huellas digitales de cepas de Acinetobacter baumannii procedentes de pacientes hospitalizados en la Caja Petrolera de Salud de Obrajes, mediante el método de Pulsed Field Gel Electrophoresis (PFGE), La Paz, Bolivia. Marzo 2015
Huellas digitales de cepas de Acinetobacter baumannii procedentes de pacientes hospitalizados en la Caja Petrolera de Salud de Obrajes, mediante el método de Pulsed Field Gel Electrophoresis (PFGE), La Paz, Bolivia. Marzo 2015
García-Rada Giovanni2*, Damiani Esther1, Copa Helen1, Ruiz Ericka1, Santalla José2, Revollo Carmen1, Mehlis Ana1, Molina Ivón3, Mamani Anselma3
1Laboratorio de Referencia Nacional en Bacteriología Clínica (LRNBC)-Instituto de Laboratorios de Salud (INLASA)
2Centro de Investigación de Genética Molecular (CIGMO)-INLASA.
3Laboratorio de Bacteriología de la Caja Petrolera de Salud en Obrajes, La Paz.
*Dirección de contacto: 1Laboratorio de Referencia Nacional en Bacteriología Clínica (LRNBC)-Instituto de Laboratorios de Salud (INLASA). Rafael Zubieta No. 1889, (lado Hospital del Niño) Zona de Miraflores. Tel +591-71992621.
Giovanni Garcia Rada
E-mail address : giovanni.rodrigo.garcia@gmail.com
Historial del artículo.
Recibido agosto, 2015.
Devuelto mayo 2015.
Aceptado octubre, 2016.
Disponible en línea, febrero, 2017.
Resumen
Acinetobacter baumannii, a nivel mundial es considerado un microorganismo oportunista presente en varios casos de infecciones intrahospitalarias. En el Hospital de Obrajes, de la Caja Petrolera de Salud, realizaron el aislamiento de 4 cepas de Acinetobacter baumannii en diferentes pacientes y diferentes salas, confirmado mediante pruebas bioquímicas. Luego, se utilizó la técnica de PFGE para la identificación de huellas digitales genómicas utilizando enzima de restricción ApaI.
Palabras clave: PFGE, huellas digitales genómicas, clones, infección intrahospitalaria.
Abstract
Acinetobacter baumannii, worldwide is considered an opportunistic microorganism, present in several cases of hospital-acquired infections. In the Caja Petrolera de Salud of Obrajes Hospital was made the isolation of four Acinetobacter baumannii strains. Identification was confirmed by biochemical tests. Then, the PFGE molecular technique was applied for the identification of genomic fingerprints using Apa I restriction enzyme.
Key words: PFGE, genomic fingerprints, clones, nosocomial infection.
Introducción
Las bacterias del género Acinetobacter son bacilos o cocobacilos gram negativos, muchas veces dispuestos en parejas. No fermentan la glucosa y son aerobios estrictos, inmóviles, catalasa positivos y oxidasa negativos. Crecen bien en todos los mediosde cultivo de rutina, siendo su temperatura óptima de crecimiento de 33 a 35° C. (Marcos 1999)
Debido a la simplicidad en sus requerimientos de crecimiento y a la capacidad para usar una gran variedad de fuentes de carbono a través de diversas vías metabólicas, A. baumannii puede ser hallado en múltiples medios animados e inanimados; así, puede ser aislado en material hospitalario, como aparatos de ventilación mecánica, catéteres, líquido de diálisis peritoneal y una amplia variedad de instrumentos. (Bergogne-Bérézin et al. 1987)
Por otra parte, A. baumannii puede formar parte de la flora normal de la piel de los adultos sanos (especialmente las manos) y puede colonizar la cavidad oral, faringe e intestino, constituyendo éstos unos reservorios epidemiológicos muy importantes en brotes nosocomiales. (Patterson et al. 1991)
Debido a la simplicidad en sus requerimientos de crecimiento y a la capacidad para usar una gran variedad de fuentes de carbono a través de diversas vías metabólicas, A. baumannii puede ser hallado en múltiples medios animados e inanimados; así, puede ser aislado en material hospitalario, como aparatos de ventilación mecánica, catéteres, líquido de diálisis peritoneal y una amplia variedad de instrumentos. (Marcos Ma et al. 1993).
Por otra parte, A. baumannii puede formar parte de la flora normal de la piel de los adultos sanos (especialmente las manos) y puede colonizar la cavidad oral, faringe e intestino, constituyendo éstos unos reservorios epidemiológicos muy importantes en brotes nosocomiales. (Patterson et al. 1991)
En los últimos años ha ocurrido un importante incremento de las infecciones nosocomiales por A. baumannii, siendo responsable de infecciones graves como sepsis, neumonía y meningitis. No es infrecuente que algunas de estas infecciones nosocomiales aparezcan en forma de brotes. (Marcos Ma et al. 1993).
Las unidades más afectadas son las de cuidados intensivos y quemados, donde el uso masivo de antibióticos puede seleccionar la aparición de cepas multirresistentes. (Lortholary et al. 1995)
La introducción de la tecnología molecular del PFGE (Electroforesis en Geles en Campo Pulsante, en español) ha permitido la identificación de patrones monoclonales en brotes, como es el caso de las infecciones intrahospitalarias. También brinda información en la confirmación de la transmisión intrahospitalaria, incluso interhospitalaria de las bacterias asociadas a infecciones en servicios de salud. (Villalón et al. 2011)
Por lo anteriormente expresado, los objetivos de esta investigación fueron: i) Estandarizar la técnica de PFGE para la determinación de patrones monoclonales de Acinetobacter baumannii. ii) Identificar los patrones monoclonales de las cepas de Acinetobacter baumannii aislados de un brote mediante PFGE.
Materiales y métodos
Cuatro aislamientos de Acinetobacter baumannii procedentes del Hospital Caja Petrolera de Salud, Obrajes, de pacientes hospitalizados, fueron remitidos al INLASA, al Laboratorio de Referencia Nacional en Bacteriología Clínica (LRNBC), para confirmar su identificación y pruebas adicionales.
Estas bacterias fueron sembradas en agar Mac Conkey (BRITANIA) por agotamiento, para trabajar con colonias monoclonales. Después de 24 horas una colonia fue resembrada en agar nutritivo (Britania) por método de siembra en superficie, con el fin de obtener una cantidad masiva de la bacteria clonada.
Este último cultivo fue utilizado para confirmar la especie mediante pruebas bioquímicas (Mac Faddin, 2da ed). Otra parte del mismo fue utilizado para la extracción de ADN para PFGE.
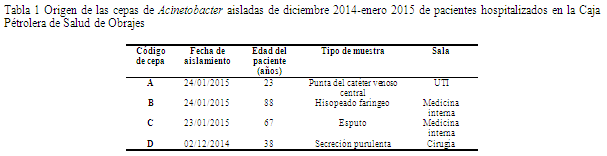
Los datos referentes a los pacientes y sus muestras (Tabla 1)
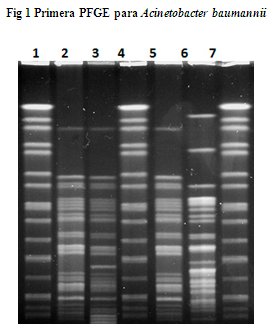
Una primera electroforesis fue realizada en equipo CHEF DR III, (Figura 1). Las condiciones fueron las siguientes: Equipo Chef DR III. Voltaje: 6V/cm, pulso inicial: 1 segundo, pulso final 35 seg, Tiempo: 23.4 h, Temperatura: 14 °C, buffer TBE 0.5X pH 8. Carriles 1, 4 y 7, cepa H9812 (Salmonella enterica Ser. Braenderup ATCC BAA 664TM) sometida con enzima XbaI (THERMO) (patrón de migración). Carriles 2, 3, 5 y 6, cepas de Acinetobacter codificadas respectivamente como A, B, C y D, digeridas con enzima ApaI (PROMEGA). Este protocolo estaba basado en el propuesto por la ARPAC (Bannerman et al. 1995).
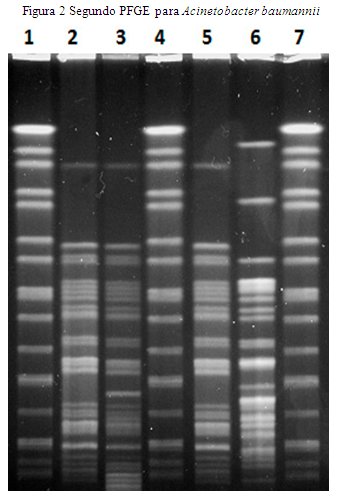
Una segunda PFGE fue realizada cambiando algunos parámetros (Pulso final: 30 segundos, duración: 18 horas. Los demás parámetros se mantienen sin cambios).
Las numeraciones, coinciden con el orden de la primera electroforesis (Figura 2).
El patrón de bandas obtenido en la segunda electroforesis fue introducido en el programa BIONUMERICS ver.6.6.
Resultados
En la primera electroforesis, muchas bandas de ADN migraron hasta salir del gel, debido al tiempo. El protocolo original de la ARPAC recomienda 28 horas (Villalón et al. 2011). Sin embargo, lo detuvimos a las 23.4 horas. Si hubiéramos esperado las 28 horas, habría mayor cantidad de bandas perdidas. Por esa razón fue modificado el protocolo reduciendo de 28 horas a 18 horas, y cambiando de 35 segundos de tiempo final de pulso, a 30 segundos. De esta manera es posible diferenciar las dos electroforesis obtenidas con los parámetros diferentes en las dos fotos de este artículo.
Las bandas son más definidas en la segunda electroforesis, y podemos distinguir un total de 3 clones diferentes (Figura 2), en lugar de dos, como se aprecia en la figura 1.
A simple vista, los carriles 2 y 5, correspondientes a las cepas A y C, de dos salas diferentes (UTI y Medicina Interna, respectivamente) proceden de un mismo clon. La cepa C es 1 día más antigua que la A, en lo que respecta a la fecha de aislamiento. Esto implicaría que la fuente de esta cepa es la muestra de esputo del paciente C, luego, al día siguiente aparece su clon en el paciente A de la sala de UTI en una muestra de punta de catéter venoso central.
El paciente de la cepa B (carril 3), ya no es portador del mismo clon, pero podemos inferir que se trata de un descendiente de la cepa C, que aparece un día después en la misma sala (Medicina Interna).
La cuarta cepa (D), aislada posiblemente de Medicina Interna, aislada 50 días antes, correspondiente también a la especie Acinetobacter baumannii, por el patrón de bandas observado en el PFGE, es un clon diferente a estas 3 nuevas.
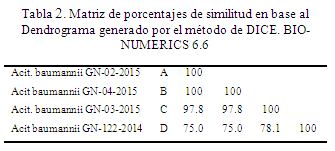
El dendrograma generado puede ser apreciado en la Figura 3. Claramente, este estadístico agrupa un par de cepas representando a un solo clon, luego presenta a la cepa B con un patrón diferente con una similitud del 97.8% valor que permite considerar que es un descendiente de la cepa C.
También es posible apreciar la similitud de bandas con respecto a la cepa D, de 50 días antes del aislamiento de las otras tres. Corresponde significativamente a un clon diferente. El porcentaje de similitud es del 78% con respecto a las cepas A y C ; y de un 75% con la cepa B.
La matriz de porcentajes de similitud, está representada en la tabla 2.
El perfil de sensibilidad a antimicrobianos es presentado en la siguiente tabla:
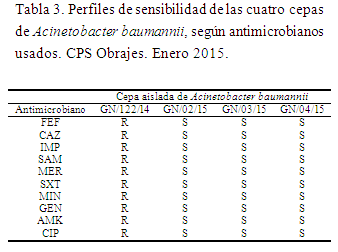
En base a los perfiles de sensibilidad. La cepa más antigua es resistente a todos los antimicrobianos probados. Las otras restantes son sensibles a todos los antimicrobianos.
Discusión
Un clon compartido en dos salas, Medicina Interna y UTI, según la información del laboratorio de la Caja Petrolera de Salud, es identifica Vdo. Luego, aparece otro clon, que por el porcentaje de similitud es descendiente de las otras dos. Posiblemente el paciente de la cepa C está infectada con una bacteria que ya tiene más tiempo de residencia en el hospital, de ahí que aparece una cepa fuertemente relacionada genómicamente (cepa B), aislada el mismo día del aislamiento de la cepa A. Por último, la cepa D, aislada 50 días antes, es un clon diferente a tres aislamientos posteriores, lo que refleja que no hubo conservación de esta cepa en el hospital. Sin embargo, esto debe ser tomado como hipótesis, dado que el número de aislamientos de la especie Acinetobacter baumannii es de apenas 4 al momento.
No obstante, es correcto decir que el clon de la cepa D, no es el mismo que los dos clones que aparecieron 50 días después.
Los perfiles de sensibilidad a los antimicrobianos confirman la lejanía filogenética presentada en el dendrograma de esta cepa con respecto a las tres que fueron aisladas en enero de 2015.
Por su parte la cepa B en base al patrón de resistencia que presenta, indica fuertemente que tiene relación con las cepas A y D, ya que el perfil es el mismo en las tres.
Por último. La ruta hipotética de contagios indica dos posibles focos de diseminación de Acinetobacter. Una primera que apareció el 2 de diciembre de 2014, que infectó a un paciente en Cirugía. El patrón PFGE de Acinetobacter de este paciente es aislado y por tanto no tuvo mayores contagios dentro de los pacientes de este estudio.
Un segundo foco, portador de la misma especie, pero correspondiente a otro clon apareció un mes después, el 23 de Enero de 2015. La sala de compromiso fue de Medicina interna. Esta cepa un día después aparece en un paciente la sala de UTI. Corresponde al mismo patrón, por tanto es la misma cepa. No obstante un paciente en la sala de Medicina Interna que aparece infectado con la misma especie, revela por PFGE que se trata de un clon ligeramente diferente, con una similitud del 97.8% según el dendrograma elaborado por UPGMA método DICE, en el software BIONUMERICS 6.6. esto indica que posiblemente el mismo foco que infectó al paciente del día anterior, fue el que contagió a este otro paciente, porque la bacteria debe estar por lo menos 4 meses para generar un cambio de disposición genómica que permita detectar una diferencia por PFGE. Lo mencionado podemos esquematizarlo en la Figura 2.
La aplicación de la técnica de PFGE, presenta en este informe la importancia de su utilización en el campo de las infecciones intrahospitalarias. La interpretación de que entre dos aislamientos correspondientes a la misma especie, cuando esto ocurre en mismo hospital en fechas cercanas es empírica. Es necesaria la demostración científica de este contagio. Una herramienta poderosa para determinar esto es PFGE. Así pudimos establecer las rutas de contagio entre estas cuatro cepas correspondientes a una misma especie. Sorprende, por ejemplo que dos clones diferentes se encuentren en la misma sala, demostrando que la fuente de contagio no fue directa entre ambos, o por lo menos no parece serlo. También sorprende que un mismo clon aparece en dos salas diferentes, revelando una infección directa entre ambos pacientes.
Este es un plan para ofrecer este servicio en el futuro. Sin embargo, aún falta poner a punto las especies Klebsiella pneumoniae y Pseudomonas aeruginosa como primera línea de avance.
Conflictos de intereses
Los autores declaran que no tienen conflictos de interés con la presente investigación.
Agradecimientos
Los autores agradecen a los pacientes hospitalizados que proveyeron las muestras para realizar el presente trabajo de investigación.
Literatura citada
Bannerman TL, Hancock GA, Tenover FC, Miller JM. Pulsed‐field gel electrophoresis as a replacement for bacteriophage typing of Staphylococcus aureus. J Clin Microbiol. 1995; 33: 551-5. [ Links ]
Bergogne-Bérézin E, Joly-Guillou ML, Vieu JF. Epidemiology of nosocomial infections due to Acinetobacter calcoaceticus. J Hosp Infect. 1987; 10(2):105-13. [ Links ]
Bionumerics version 6.6: New F Eatures Copyright By Applied Maths Nv. July 2011.
Lortholary O, Fagon JY, Hoi AB, Slama MA, Pierre J, Giral P, et al. Nosocomial acquisition of multiresistant Acinetobacter baumannii: risk factors and prognosis. Clin Infect Dis. 1995; 20(4): 790-6. [ Links ]
Marcos Mª Angeles, Acinetobacter Baumannii. Departamento de Microbiología y Parasitología. Hospital Clínico. Universidad de Barcelona. 1999. [ Links ]
Marcos Ma, Vila J, Jiménez de Anta Mt. Epidemiología de las infecciones por Acinetobacter baumannii. Enferm Infec. Microbiol Clin 1993; 11:29-33. [ Links ]
Patterson JE, Vecchio J, Pantelick EL, Farrel P, Mazon D, Zervos MJ, et al. Association of contaminated gloves with transmission of Acinetobacter calcoaceticus var. anitratus in an intensive care unit. Am J Med. 1991; 91(5):479-83. [ Links ]
Villalón P, Valdezate S, Medina-Pascual MJ, Rubio V, Vindel A, Saez-Nieto JA. Clonal Diversity of Nosocomial Epidemic Acinetobacter baumannii Strains Isolated in Spain. J Clin Microbiol. 2011; 49(3): 875-82. [ Links ]
______________












 uBio
uBio