Servicios Personalizados
Revista
Articulo
Indicadores
Links relacionados
Compartir
Revista Científica Ciencia Médica
versión impresa ISSN 2077-3323
Rev Cient Cienc Méd vol.23 no.1 Cochabamba 2020
ARTÍCULO DE REVISIÓN
COVID-19: ASPECTOS VIROLOGICOS Y PATOGENESIS
COVID-19: VIROLOGICAL ASPECTS AND PATHOGENESIS
Bismar Jorge Gutierrez Choque 1, Carla Jimena Aruquipa Quispe 2.
1 Doctor of Medicine, Facultad de Medicina, Universidad Mayor de San Andrés.
2 Estudiante de Quinto año de Medicina, Facultad de Medicina, UMSA.
Correspondencia a: Bismar Jorge Gutierrez Choque
Correo electrónico: thebismar12@hotmail.com
Procedencia y arbitraje: no comisionado, sometido a arbitraje externo.
Recibido para publicación: 9 de abril 2020
Aceptado para publicación: 30 de junio 2020
Citar como: Gutiérrez Choque BJ, Aruquipa Quispe CJ. COVID-19: aspectos virologicos y patogenesis. Rev Cient Cienc Med 2020; 23(1): 77-86
RESUMEN
El nuevo coronavirus 2019, posteriormente conocido como SARS-CoV-2 ha generado una nueva pandemia y hoy se reúnen esfuerzos para su control. Se hacen investigaciones de la estructura genómica del virus y de sus mecanismos patogénicos, primero describiendo su estructura para así comprender su función, también en relación a su unión con los receptores que se expresan en los órganos más afectados.
La relación estructural que se encuentra en los Dominios de Unión de SARS-CoV-2, son compatibles con los dominios de unión de SARS-CoV. Hay diferencias en algunos segmentos de la cadena, pero esto no parece alterar la letalidad del virus.
El uso de técnicas de criomicroscopía hizo posible reconocer estructuras de la superficie viral y hacer una descripción de su unión al receptor ECA2. Además de esta manera se puede fijar objetivos con potencial terapéutico.
Palabras clave: Coronavirus, Virus del SARS, Infección.
ABSTRACT
The new coronavirus 2019, later known as SARS-CoV-2, has generated a new pandemic and today efforts are being made to control it, investigations are made of the genomic structure of the virus and also of pathogenic mechanisms, first describing its structure and thus its function, as well as in relation to its union with the receptors that are expressed in its most affected organs.
The structural relationship found in the SARS-CoV-2 Binding Domains are compatible with the SARS-CoV binding domains. There are differences in some segments of the chain, but this does not seem to alter the lethality of the virus. The use of cryomicroscopy techniques helped to recognize viral surface structures and to describe their binding to the ECA2 receptor. In addition, in this way, targets with therapeutic potential can be set.
Keywords: Coronavirus, SARS virus, Infection.
INTRODUCCIÓN
El nuevo coronavirus 2019 (2019-nCoV), luego denominado SARS-CoV-2, causante de la Enfermedad por Coronavirus 2019 (COVID-19), ha generado una nueva pandemia a nivel mundial, ocasionando temor e incertidumbre para todas las personas y sobre todo para el personal de salud2. Poco se sabe aún de los mecanismos moleculares de este nuevo virus y como afecta a los seres humanos desde el punto de vista fisiopatológico. Se estudia la relación de la Enzima Convertidora de Angiotensina tipo 2 en la patogénesis de la enfermedad, en su entrada a los neumocitos y la comparación de los Dominios y Motivos de unión, mediante modelos moleculares y asociaciones filogenéticas con SARS-CoV. Describir la estructura genómica viral, permitirá entender el funcionamiento y su interacción con receptores u otras moléculas asociadas a este patógeno, por tal razón mediante criomicroscopía se intenta describir toda su estructura3,4.
En esta revisión intentaremos mostrar los acercamientos que con gran esfuerzo intentan describir la patogenia que desencadena el SARS-CoV-2.
Metodología de búsqueda de artículos
Se realizó una investigación bibliográfica informática de la literatura en los motores de búsqueda PubMed, ScienceDirect, SciELO y Google Academics durante el periodo de enero a marzo del 2020, utilizando las siguientes palabras clave "Coronavirus", "2019-nCoV", "2019 Novel Coronavirus", "COVID-19" y SARS-CoV-2. La búsqueda generó 10480 artículos en inglés, fueron seleccionados artículos con nivel de evidencia II-1, II-2 y II-3; incluyendo artículos de revisión bibliográfica, reportes epidemiológicos, estudios experimentales, en los últimos 5 años. También se incluyeron artículos que fueron base para la descripción de los coronavirus, con una antigüedad de publicación superior a los 10 años, que son fundamentales por ser inéditos. Se excluyeron cartas al editor, análisis de los estudios por un solo autor y con datos inconclusos. No se encontraron artículos de revisión en español sobre aspectos virológicos con relación a la patogenia del SARS-CoV-2.
DESARROLLO
En los últimos días del mes de diciembre, del año 2019 se informó de un grupo de pacientes con Neumonía de origen desconocido que compartían el factor de exposición al Mercado Mayorista de Mariscos en Wuhan, China5.
Se identificaron muchos casos de Neumonía por el entonces conocido como Nuevo Coronavirus 2019 (2019-nCoV) en Wuhan, una ciudad de la provincia Hubei. En un principio su propagación fue limitada a sus países vecinos, como Tailandia, Japón, Corea del Sur y otros. Sin embargo ha alcanzado países de Europa y el continente Americano, donde se han confirmado casos en EEUU, Canadá, Centroamérica y países de Sudamérica. El 30 de enero de 2020 la OMS declara Emergencia de Salud Pública de importancia internacional. Esto sucede si un evento plantea una amenaza a la Salud Pública para otros países a través de la propagación global de enfermedades, y que, potencialmente requiere una respuesta internacional coordinada 6.
Posteriormente el 11 de marzo de 2020 la OMS declara pandemia, al evaluar la expansión globalizada de este coronavirus. La gran cantidad de pacientes infectados, su gravedad y los niveles alarmantes de inacción, que mencionó el máximo representante de esta organización, obliga a tomar medidas internacionales7.
Los países de América Latina ya tienen una gran cantidad de pacientes con COVID-19. Hay una gran preocupación por países de escasos recursos, donde su capacidad para enfrentar esta pandemia es muy limitada 6,7.
Características epidemiológicas
Los parámetros epidemiológicos han sido estudiados en diferentes momentos y con una gran variabilidad. Se han publicado varios valores del número reproductivo básico (R0) para el nuevo coronavirus de Wuhan, el SARS-CoV-2, que van desde 1,4 hasta 5,58. Según la OMS se está produciendo una transmisión de persona a persona y se presentó una estimación preliminar de R0 de 1.4-2.5. La amplificación se ha producido en un centro de atención médica. De los casos confirmados, se informa que el 25% son graves. La fuente aún se desconoce (muy probablemente un reservorio animal) y el alcance de la transmisión de persona a persona aún no está claro9,10.
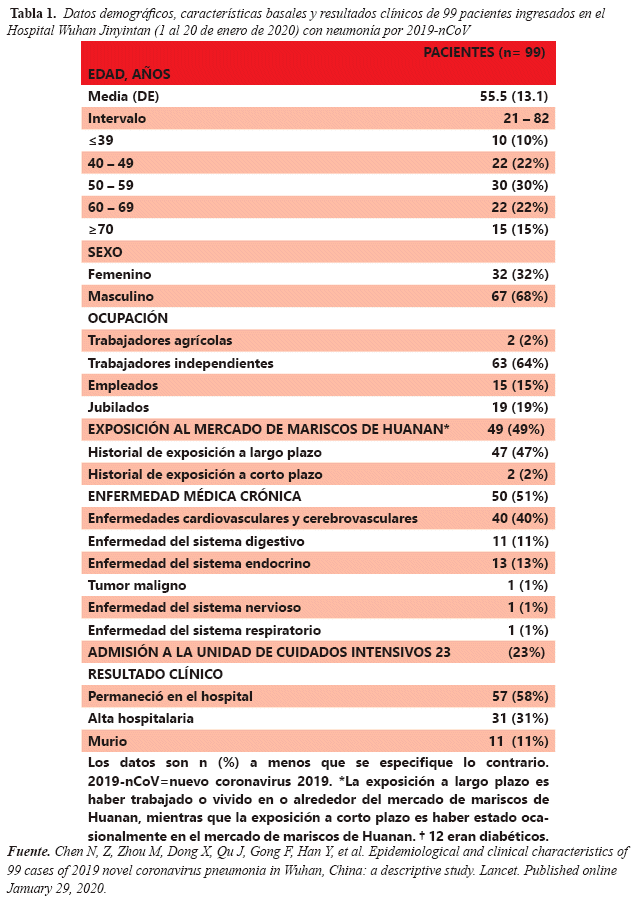
Zhang L. et.al. Realizaron un estudio retrospectivo de un solo centro, donde reclutaron pacientes del 1 al 20 de enero del 2020, en el Hospital Jinyintan en Wuhan, China, especializado en formas infecciosas. Todos los pacientes ingresados que fueron diagnosticados con Neumonía por SARS-CoV-2 según la guía provisional de la OMS se inscribieron en este estudio. 99 pacientes con COVID-19 fueron incluidos en este estudio.
En total 49% de pacientes tenían antecedente de exposición al Mercado Mayorista de Mariscos de Huanan. La edad promedio de los pacientes fue de 55,5 años (DE+/-13,1), incluidos 67 hombres y 32 mujeres. Se confirmó COVID-19 mediante RT-PCR. De los pacientes incluidos al estudio 51% tenían enfermedades crónicas8.(Tabla 1).
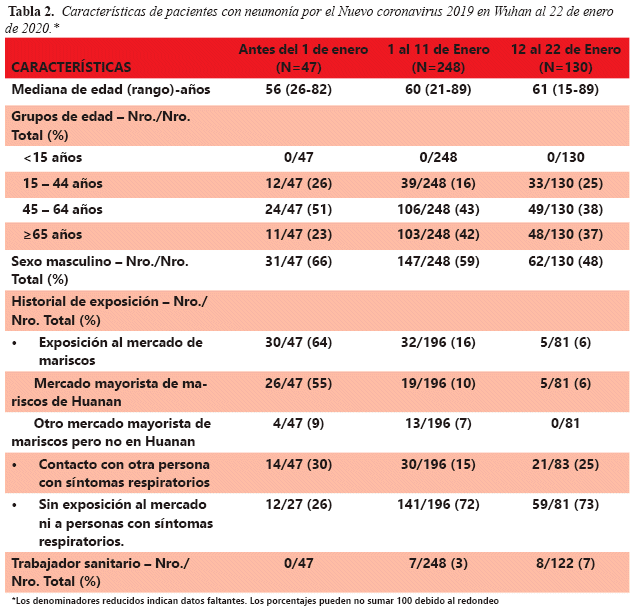
Li Q. et.al. Recopilaron información sobre características demográficas, historial de exposición y la línea de tiempo de la enfermedad de 425 casos confirmados que se habían informado antes del 22 de enero de 2020. La edad promedio de los pacientes con COVID-19 fue de 59 años y el 56% eran hombres. No hubo casos en menores de 15 años. Se examinaron los datos sobre exposiciones entre 10 casos confirmados y se estimó que el periodo de incubación promedio fue de 5,2 días (intervalo de confianza [IC] del 95%, 4,1 a 7,0); el percentil 95 de la distribución fue de 12,5 días (IC de 95%, 9,2 a 18)11. (Tabla 2).
Coronavirus: estructura genómica y replicación viral
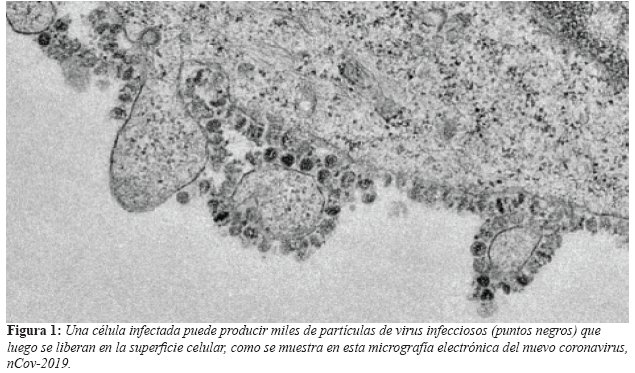
Los Coronavirus de los humanos se han clasificado como una subfamilia de la Familia Coronaviridae, del orden Nidovirales. Son Viriones con envoltura más o menos esféricos de 120 a 160 nm de diámetro. Su nombre se debe a sus proyecciones características de "Corona" en su superficie de aproximadamente 20 nm de largo12. (Figura 1).
Son virus de RNA monocatenario de polaridad positiva, son sensibles al calor y a los solventes lipídicos. Antes del brote de SARS, se reconoció que el coronavirus infectaba a muchas especies diferentes, particularmente animales domésticos y de laboratorio, su extrema diversidad en la naturaleza no se apreciaba completamente12.
Aunque se sabía que las enfermedades graves solo ocurrían en los animales, en los humanos se pensaba que los virus causaban solo una infección respiratoria aguda que generalmente era leve. Todo esto cambio con el reconocimiento de que el SARS fue causado por un coronavirus (SARS-CoV). Muy recientemente, se ha descubierto que el Coronavirus del Síndrome respiratorio de Oriente Medio (MERS-CoV), un nuevo virus de linaje C del Betacoronavirus humano, está infectando a los humanos en los países del medio oriente12,13.
El genoma del SARS-CoV-2 es un RNA monocatenario de polaridad positiva con un extremo 5' metilado y un extremo 3' poliadenilado, con 29903 bases (~ 30 kb). El RNA genómico se utiliza como plantilla, para traducir directamente la Poli-Proteína (pp) 1a/1b, que codifica proteínas no estructurales para formar un Complejo de Replicasa-Transcriptasa (CRT) en vesículas de doble membrana. Posteriormente, un conjunto de RNA subgenómico (sgRNA) son sintetizados por el CRT en una forma de transcripción discontinua. Estos RNAm subgenómicos poseen una secuencia común 5' iniciador y 3' terminal. La terminación de la transcripción y la posterior adquisición de una RNA iniciador se produce en las secuencias reguladoras de la transcripción (SRT) ubicados entre los Marcos Abiertos de Lectura (del inglés, Open Reading Frames) (ORF's). Estos RNA subgenómicos de cadena negativa sirven como plantillas para la producción de RNAmsubgenómicos12,13.
Los genomas y subgenomas del SARS-CoV-2 contienen al menos 6 ORF's. El primer ORF (ORF 1a/b), aproximadamente dos tercios de la longitud del genoma, codifica 16 proteínas no estructurales (nsp 1-16), excepto los Gammacoronavirus que carecen de nsp-1. Hay un desplazamiento de marco-1 entre ORF1a y ORF1b, lo que lleva a la producción de dos polipéptidos: pp1a y pp1b. Estos polipéptidos serán procesados por la proteasa similar a la quimiotripsina codificada viralmente (3CLpro) o la proteasa principal (Mpro) y una o dos proteasas similares a la papaína (PLP) en 16 nsp. Otros ORF en un tercio del genoma del extremo 3' terminal, codifica al menos cuatro proteínas estructurales principales: espiga (S), membrana (M), envoltura (E) y proteínas de la nucleocápside (N). Además de estas cuatro proteínas estructurales, diferentes CoV codifican proteínas estructurales y accesorias, como la proteína HE, proteína 3a/b, proteína 4a/b y otros. Todas las proteínas estructurales y accesorias se traducen a partir de RNA subgenómicos del CoV12,13,14,15. Se realizaron análisis por secuenciación genética que mostró que el SARS-CoV-2 posee una estructura genómica típica de coronavirus y pertenece al grupo Betacoronavirus que incluye Bat-SARS-like (SL)-ZC45, Bat-SL ZXc21, SARS-CoV y MERS-CoV12,13.
Funciones de las proteínas estructurales y no estructurales en la replicación de los Coronavirus
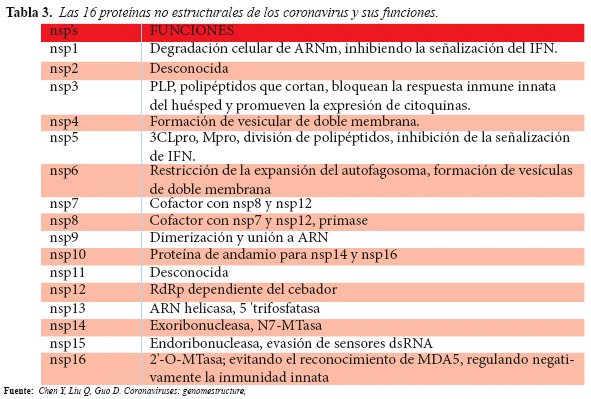
La mayoría de las proteínas no estructurales nsp1-16 han sido reportadas por sus roles específicos en la replicación del CoV. Sin embargo, las funciones de algunas nsp son desconocidos o no se entienden completamente. Se resumen las funciones de los 16 nsp's en la tabla 313.
Cuatro proteínas estructurales son esenciales para el ensamblaje de viriones y la infección de CoV: (1) Los homotrímeros de las proteínas S constituyen la espiga en la superficie de las partículas virales y es la clave para la unión viral al receptor del huésped. (2) La proteína M tiene tres dominios transmembrana y da forma a los viriones, promueve la curvatura de la membrana y se une a la nucleocápside. (3) La proteína E juega un papel en el ensamblaje y liberación del virus, y es necesaria para la patogénesis. (4) La proteína N contiene dos dominios, ambos pueden unirse al genoma de ARN del virus a través de diferentes mecanismos. Se informa que la proteína N puede unirse a la proteína nsp3 para ayudar a unir el genoma al complejo replicasa-transcriptasa (CRT) y empaquetar el genoma encapsidado en viriones. La proteína N también es un antagonista del interferón y el represor viral codificado (VSR) de RNA de interferencia (RNAi), que benefician la replicación viral13.
Patogénesis de la COVID-19
El coronavirus está distribuido ampliamente en humanos, otros mamíferos y aves, causando enfermedades respiratorias, entéricas, hepáticas y neurológicas. El rango de huéspedes y el tropismo tisular muestran mucha variación entre los diferentes CoV13.
Antes del 2019, solo había seis especies de CoV, que afectaban a los humanos y podían ocasionar enfermedades respiratorias: HCoV-229E, HCoV-OC43, HCoV-NL63 y HKU1. Estas especies causan síntomas de resfriado común en individuos inmunocompetentes. Sin embargo hay otras dos cepas de origen zoonótico que pueden ocasionar enfermedad grave en los humanos: el SARS-CoV, que fue el agente causal de los brotes de Sindrome Respiratorio Agudo Severo en 2002 y 2003 en la provincia de Guangdong, China. MERS-CoV que fue el patógeno responsable de los brotes de enfermedades respiratorias graves en 2012 en el medio Oriente13,14.
El nuevo coronavirus SARS-CoV-2, que pertenece a los betacoronavirus según el análisis del genoma, también puede infectar el tracto respiratorio inferiory causar neumonía, pero en general, los síntomas son más leves que el SARS y el MERS13.
Estudios anteriores han descubierto varios receptores a los que se unen diferentes coronavirus, como la Enzima Convertidora de Angiotensina tipo 2 (ECA2) para SARS-CoV y CD26 para MERS-CoV16,17.
Un modelo molecular mostró similitud estructural entre los dominios de unión al receptor de SARS-CoV y el nuevo coronavirus del 2019, SARS-CoV-2. Por lo tanto, se sugiere que SARS-CoV-2 mediante su proteína S podría usar la ECA2 como receptor, a pesar de la presencia de mutaciones de aminoácidos en el dominio de unión al receptor. Las similitudes de secuencia generales entre la espiga del SARS-CoV-2 y la espiga del SARS-CoV (aislado de humanos, civetas o murciélagos) son de alrededor del 76% a 78% para la proteína completa, alrededor del 73% a 76% para su Dominio de Unión al Receptor (del inglés Receptor BindingDomain [RBD]) y del 50% a 53 % para el Motivo de Unión al Receptor (del inglés Receptor BindingMotif [RBM]). En comparación, el MERS de coronavirus humano (MERS-CoV) y el coronavirus HKU4 tipo murciélago comparten similitudes de secuencia más bajas en sus espigas, RBD o RBM, y sin embargo reconocen el mismo receptor de dipeptidil peptidasa 4 (DPP4). Por lo tanto, las similitudes de secuencia entre las espigas SARS-CoV-2 y SARS-CoV sugieren la posibilidad de que compartan el mismo receptor ECA2. Es importante destacar que, en comparación con SARS-CoV RBM, SARS-CoV-2 RBM no contiene ninguna eliminación o inserción (excepto una inserción de un residuo en un bucle alejado de la región de unión a ECA2), proporcionando evidencia adicional de que SARS-CoV-2 usa ECA2 como su receptor. Además, entre los 14 residuos que entran en contacto con la ECA2 en el RBD, 9 están completamente conservados en SARS-CoV-2 y 4 están parcialmente conservados entre SARS-CoV-2 y SARS-CoV de humanos, civetas y murciélagos. Una última pieza de evidencia sólida que respalda a ECA2 como receptor para SARS-CoV-2 rodea los cinco residuos en SARS-CoV-2 RBM que se sometieron a selecciones naturales en SARS-CoV y desempeñaron papeles críticos en la transmisión entre especies de SARS-CoV (es decir, residuos 442, 472, 479, 480, 487 en SARS-CoV RBD). Primero, el residuo 493 en SARS-CoV-2 RBD (correspondiente al residuo 479 en SARS CoV) es una glutamina. Un SARS-CoV RBD previamente diseñado es óptimo para unirse a la ECA2 humano. El residuo 479 en SARS-CoV RBD se encuentra cerca del punto de acceso de unión al virus Lys31 (es decir, punto de acceso-31) en ECA2 humano. El punto de acceso 31 consiste en un puente de sal entre Lys31 y Glu35 sepultado en un ambiente hidrofóbico. En la civeta SARS-CoV RBD (año 2002), el residuo 479 es una lisina, que impone interferencia estérica y electrostática con el Punto de acceso-31. En el SARS-CoV RBD humano (año 2002), el residuo 479 se convierte en una asparagina. La mutación K479N elimina la interacción desfavorable en la interfaz RBD/ ECA2 humana, mejora la unión viral a la ECA2 humana y desempeña un papel crítico en la transmisión de la civeta al ser humano de CoV de SARS. Es importante destacar que Gln493 en SARS-CoV-2 RBD es compatible con punto de acceso-31, lo que sugiere que SARS-CoV-2 es capaz de reconocer ECA2 humano e infectar células humanas15,16,17,18.
Segundo, el residuo 501 en SARS-CoV-2 RBD (correspondiente al residuo 487 en SARS-CoV) es una Asparagina. Según el análisis estructural anterior, el residuo 487 en el SARS-CoV-1 se encuentra cerca del punto de enlace de unión al virus Lys353 (es decir, punto de acceso-353) en la ECA2 humano. El punto de acceso-353 consiste en un puente de sal entre Lys353 y Asp38 también sepultado en un ambiente hidrofóbico. En civetas SARS-CoVRBD (año 2002), el residuo 487 es una Serina, que no puede proporcionar un soporte favorable para el punto de acceso-353. En el SARS-CoV humano aislado en el año 2002, el residuo 487 es una Treonina, que fortalece la estabilidad estructural del punto de acceso-353. La mutación S487T agrega la interacción favorable en la interfaz RBD/ ECA2 humana, mejora la unión viral a la ECA2 humana y desempeñó un papel crítico en la transmisión de SARS-CoV de persona a persona. En el SARS-CoV humano aislado en el año 2003, el residuo 487 es una Serina y no hubo transmisión de humano a humano para esta cepa de SARS-CoV. Asn501 en SARS-CoV-2 RBD proporciona más soporte para el punto de acceso-353 que Ser487, pero menos queThr487. Este análisis sugiere que SARS-CoV-2 reconoce el ECA2 humano de manera menos eficiente que el SARS-CoV humano (año 2002), pero más eficientemente que el SARS-CoV humano (año 2003). Por lo tanto, al menos al considerar las interacciones ECA2-RBD, SARS-CoV-2 ha adquirido cierta capacidad de transmisión de humanos a humanos15,16,17,18.
Tercero, los residuos 455, 486 y 494 son leucina, fenilalanina y serina en SARS-CoV-2 RBD, respectivamente (correspondientes a los residuos 442, 472 y 480 en SARS-CoV RBD, respectivamente). En base al análisis estructural anterior, estos tres residuos en SARS-CoV RBD juegan un papel significativo, aunque no tan marcado como los residuos 479 y 487, en la unión de ECA2. Más específicamente, el Tyr442 del SARS-CoV RBD humanos y de la civeta proporciona interacciones desfavorables con el punto de acceso-31 en ECA2 humano (este residuo ha mutado a Phe442 en el RBD optimizado); Leu455 de SARS-CoV-2 RBD proporciona interacciones favorables con el punto de acceso-31, mejorando así la unión viral a ECA2 humano. Leu472 del SARS-CoV RBD humano y de civeta proporciona un soporte favorable para el punto de acceso-31 en ECA2 humano a través de interacciones hidrofóbicas con el residuo ECA2 Met82 y varios otros residuos hidrofóbicos (este residuo ha mutado a Phe472 en el RBD optimizado); Phe486 del SARS-CoV-2 RBD proporciona aún más soporte para el punto de acceso-31, por lo tanto, también mejora la unión viral a ECA2 humano. Asp480 del SARS-CoV RBD humano y de la civeta proporciona un soporte favorable para el punto de acceso-353 en ECA2 humano a través de una tirosina vecina (este residuo permanece como un aspartato en el RBD optimizado); Ser494en el SARS-CoV-2 RBD aún brinda soporte positivo para el punto de acceso-353, pero el soporte no es tan favorable como lo proporciona el Asp480. En general, Leu455, Phe486 y Ser494 de SARS-CoV-2 RBD admiten que SARS-CoV-2 reconoce ECA2 humano e infecta células humanas15,16,17,18.
Estos análisis se basan en el modelo de las interacciones SARS-CoV-2 RBD/ECA2, fuertemente fundamentadas en una serie de estructuras de nivel atómico de SARS-CoV aisladas de diferentes huéspedes en diferentes años. Ciertamente, hay otros factores que afectan la infectividad y la patogénesis de SARS-CoV-2 y deberán investigarse. Sin embargo, estos estudios estructurales de una década sobre el SARS-CoV han demostrado firmemente que el reconocimiento del receptor por el SARS-CoV es uno de los determinantes más importantes de su transmisión entre especies y transmisión de persona a persona, una conclusión que ha sido confirmada por diferentes líneas de investigación. Uno de los objetivos a largo plazo de estos estudios estructurales previos sobre el SARS-CoV fue construir un marco iterativo a nivel atómico de interacciones virus-receptor que facilite la vigilancia epidemiológica, prediga el uso de receptores específicos de la especie e identifique posibles huéspedes animales y animales modelos de enfermedades humanas. Estos estudios proporcionan una prueba amplia de este marco reiterativo, brindando a las comunidades de investigación básica, traslacional y de salud pública ideas predictivas que pueden ayudar a estudiar y combatir esta epidemia global de SARS-CoV-215,16,17,18.
La expresión del receptor ECA2 también se encuentra en muchos tejidos extrapulmonares, incluidos el corazón, los riñones, el endotelio y el intestino19.
Receptor ECA2: biología y su potencial terapéutico
Entender los mecanismos de infección es fundamental para plantearnos terapias específicas contra el coronavirus. Los inhibidores de la ECA constituyen la primera línea del tratamiento antihipertensivo. El descubrimiento de su homologo, ECA2, ha desencadenado un interés sustancial, sin embargo se evidencio que ECA2, no convertía Ang-I en Ang-II, ni fue inhibido por IECA. Se notó de inmediato una gran diferencia en la especificidad del sustrato, al saber, que ECA2 actuaba como una carboxipeptidasa eliminando un solo aminoácido del extremo C de los sustratos susceptibles, mientras que ECA actúa como una carboxi-dipeptidasa (más concretamente, peptidil-dipeptidasa), eliminando un Dipéptido C-terminal. ECA2 hidroliza Ang I, en Ang (1-9) en lugar de Ang II (Ang-(1-8)). Estudios en modelos comparativos, demostraron su especificidad distintiva al revelar diferencias en los sitios activos de ECA y ECA2. La sustitución de un aminoácido en ECA2 impide estéricamente la entrada del penúltimo aminoácido sustrato en el sitio activo, eliminando asi la actividad de peptidil-dipeptidasa similar a ECA. También se demostró que ECA2 tiene mayor acción catalítica para la hidrolisis de Ang-II (400 veces) en comparación con Ang-I. Solo bajo condiciones de concentraciones elevadas de Ang-I (como en paciente con tratamiento con IECA) es probable que la conversión de Ang-I a Ang-(1-9) por ECA2 sea de alguna importancia fisiológica. El producto principal de la actividad catalítica de ECA2 es Ang-(1-7), un péptido vasodilatador20.
Wan y col. informaron que el residuo 394 (glutamina) en el dominio de unión al receptor de SARS-CoV-2 (RBD), correspondiente al residuo 479 en SARS-CoV, puede ser reconocido por la lys-31 en el receptor ECA2 humano. Se presumía que SARS-CoV-2 podía tener la misma afinidad que SARS-CoV por el receptor ECA221.
Después de la secuenciación genómica de SARS-CoV-2, se realizó inmediatamente la estructuración de una imagen tridimensional de su glicoproteína espicular S. La imagen se logró por medio de criomicroscopia electrónica, gracias a esta imagen con resolución de 3,5 Å se confirma que esta proteína se acopla a la ECA2 de las células humanas con mayor afinidad que la del SARS-CoV. La secuencia de la proteína S del SARS-CoV-2 coincide al 98% con la proteína S del coronavirus Bat-RaTG13, con la gran diferencia de que en el sitio de escisión se encuentra un segmento de cuatro aminoácidos compuestos por :arginina-arginina-alanina-arginina en lugar de una sola arginina (R). Después de una comparación realizada entre los 61 genomas completos de SARS-CoV-2 se evidencio que solo hay 9 aminoácidos diferentes entre todos ellos y que estas variantes no afectan la letalidad del virus22,23,24,25.
DISCUSIÓN
Desde los orígenes de esta enfermedad que se ha propagado por todo el mundo, describir sus características virológicas y clínicas, han ido desde el hallazgo de una Neumonía de Origen desconocido en la Ciudad de Wuhan que compartía el factor de exposición a un mercado de Mariscos. Posteriormente se identificó a un betacoronavirus que comparte características de infección, parecidas al SARS-CoV, por lo cual se le denomino SARS-CoV-25,6. Ya declarada la pandemia por la OMS, se ha puesto en marcha la descripción patogénica de esta enfermedad y encontrar una vacuna se ha convertido en una carrera6,7,9.El potencial de inefectividad del SARS-CoV-2 es alto, no todos los infectados desarrollan una enfermedad grave, sin embargo, a él se han asociado algunos factores de riesgo, que aumentan la mortalidad de esta población7,8. Según la revisión realizada el periodo de incubación puede abarcar hasta un promedio de 12,5 días, esto debe ir en relación a la carga viral detectable por RT-PCR11.
Los dos tercios del genoma del coronavirus, perteneciente a ORF1a/b corresponde a la expresión de proteínas funcionales, sin embargo solo un tercio del genoma codifica a proteínas estructurales, de estas, la que tiene mayor importancia para describir la patogénesis es la proteína de Spike. Después de realizado un modelo molecular y hallar similitudes estructurales en los dominios de unión al receptor de SARS-CoV y el SARS-CoV-2, se puede presumir que a pesar de las mutaciones de aminoácidos en los dominios de unión, SARS-CoV-2 no contiene ninguna eliminación o inserción en el lugar de unión, lo que es evidencia clara de que SARS-CoV-2 utiliza a ECA2 como receptor para su unión. Hay cinco residuos en SARS-CoV-2 RBM que son fundamentales para la unión a ECA2, los residuos 442, 472, 479, 480 y 487; tiene relación con los dominios de unión de la misma línea filogenética que el SARS-CoV15,16,17.
La compatibilidad del punto de acceso-31 con la Gln493, es una interacción clave para reconocer ECA2 humano e infectar células humanas16,17,18.
Los estudios de asociación de la ECA2 y SARS-CoV-2, es tema de investigación para no solo explicar la patogenia, también para buscar otros enfoques terapéuticos potenciales que se basan en su afinidad por la ECA2. Estos ensayos también sirvieron para explicar su asociación con la mortalidad elevada en pacientes con Hipertensión y Diabetes15,19,20.
Trabajos previos sobre la patogénesis del SARS-CoV hace que ACE2 sea un objetivo terapéutico racional y científicamente validado para la pandemia actualdeCOVID-19. La disponibilidad de ACE2 recombinante fue el impulso para reunir un equipo multinacional de intensivistas, científicos y biotecnología para iniciar rápidamente un ensayo piloto de rhACE2 en pacientes con COVID-19 grave19,20.
CONCLUSIÓN
La Enfermedad por Coronavirus 2019 llego a ser una pandemia. En un mundo desde lo alto del antropocentrismo, vemos como los microorganismos nos dan grandes lecciones de adaptación a este medio hostil. La aparición de formas más complejas de vida, no conlleva consigo la desaparición de formas simples; lo que ha producido una reacomodación simbiótica. Esto ha permitido a los sistemas tener mayor resiliencia. Aunque el conocimiento de las bases de esta enfermedad emergente es todavía muy incompleto, en los últimos años la investigación de este tipo de enfermedades se ha centrado en el contagio y el "salto" del patógeno desde el animal al hombre, en lo que influyen multitud de circunstancias dependientes de ambos y de la interfaz, en lo que se va progresando.
En un principio una zoonosis que la transmitían los murciélagos y posteriormente una transmisión de persona a persona, la cual ha llevado a un aumento exponencial de casos reportados de COVID-19.
Desde el punto de vista biológico, nos interesa conocer la estructura del coronavirus para determinar la función que tiene cada uno de sus componentes en la patogenia de la enfermedad. Se logró de esa manera, evidenciar que SARS-CoV-2 tiene mayor afinidad por ECA2, que el SARS-CoV previamente descrito.
Aun debemos poner todos nuestros esfuerzos para poder controlar la transmisión en la población, además de un tratamiento dirigido contra el virus y una vacuna, de la que hasta hoy todavía se esperan los resultados finales.
REFERENCIAS
1. Gao GF. From “A”IV to “Z”IKV: attacks from emerging and re-emerging pathogens. Cell 2018; 172:1157-1159. Disponible en: DOI: 10.1016/j.cell.2018.02.025.
2. Weiss SR, Leibowitz JL. Coronavirus pathogenesis. Adv Virus Res 2011; 81:85-164. Disponible en: DOI: 10.1016/B978-0-12-385885-6.00009-2.
3. Lu H, Stratton CW, Tang YW. Outbreak of pneumonia of unknown etiology in Wuhan, China: The mystery and the miracle. J Med Virology. 2020; 92 (4): 401-402. Disponible en: DOI: 10.1016/S0140-6736(20)30211-7.
4. Cui J, Li F, Shi ZL. Origin and evolution of pathogenic coronaviruses. Nat Rev Microbiol 2019; 17: 181-92. Disponible en: DOI: 10.1038/s41579-018-0118-9.
5. Zhu N, Zhang D, Wang W, Li X, Yang B, Song J, et.al. A Novel Coronavirus from Patients with Pneumonia in China, 2019. N Engl J Med. Published online January 25, 2020. Disponible en: DOI: 10.1056/NEJMoa2001017.
6. World Health Organization: Declares Public Health Emergency for Novel Coronavirus; Medscape - Jan 30, 2020. Disponible en: https://www.medscape.com/viewarticle/924596. [ Links ]
7. World Health Organization: Coronavirus disease 2019 (COVID-19) Situation Report – 52. Reported on March 12, 2020. Disponible en: https://www.who.int/emergencies/diseases/novelcoronavirus-2019/situation-reports.
8. Chen N, Zhou M, Dong X, Qu J, Gong F, Han Y, et.al. Epidemiological and clinical characteristics of 99 cases of 2019 novel coronavirus pneumonia in Wuhan, China: a descriptive study. Lancet. Published online January 29, 2020. Disponible en: DOI: 10.1016/S0140-6736(20)30211-7.
9. World Health Organization: Statement on the meeting of the International Health Regulations (2005) Emergency Committee regarding the outbreak of novel coronavirus (2019-nCoV). Disponible en: https://www.who.int/news-room/detail/23-01-2020-statement-on-the-meetingof-the-international-health-regulations-(2005)-emergency-committee-regarding-the-outbreakof-novel-coronavirus-(2019-ncov).
10. Read JM, Bridgen JR, Cummings D, Ho A, Jewell CP. Novel coronavirus 2019-nCoV: early estimation of epidemiological parameters and epidemic predictions. medRxiv. 2020; 1(2): 1-8. Disponible en: DOI: 10.1101/2020.01.23.20018549.
11. Li Q, Guan X, Wu P, Wang X, Zhou L, Tong Y, et.al. Early Transmission Dynamics in Wuhan, China, of Novel Coronavirus–Infected Pneumonia. N Engl J Med. Published online January 29, 2020. Disponible en: DOI: 10.1056/NEJMoa2001316.
12. Monto A.S., Cowling B.J., Peiris J.S.M. (2014) Coronaviruses. In: Kaslow R., Stanberry L., Le Duc J. (eds) Viral Infections of Humans. Springer, Boston, MA. Disponible en: DOI: 10.1007/978-1-4899-7448-8_10.
13. Chen Y, Liu Q, Guo D. Coronaviruses: genome structure, replication, and pathogenesis. J Med Virol. 2020: 1 – 6. Disponible en: DOI: 10.1002/jmv.25681.
14. Lu R, Zhao X, Li J, Niu P, Yang B, Wu H, et.al. Genomic characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding. Lancet. Published online January 29, 2020. Disponible en: DOI: 10.1016/S0140-6736(20)30251-8.
15. Wan Y, Shang J, Graham R, Baric RS, Li F. Receptor recognition by novel coronavirus from Wuhan: An analysis based on decade-long structural studies of SARS. J Virol; Accepted Manuscript Posted Online 29 January 2020. Disponible en: DOI: 10.1128/JVI.00127-20.
16. Li F Li W Farzan M Harrison SC Structure of SARS coronavirus spike receptor-binding domain complexed with receptor. Science. 2005; 309: 1864-1868. Disponible en: DOI: 10.1126/science.1116480.
17. Lu G Hu Y Wang Q et al. Molecular basis of binding between novel human coronavirus MERSCoV and its receptor CD26. Nature. 2013; 500: 227-231. Disponible en: DOI: 10.1038/nature12328.
18. Wu KL, Peng GQ, Wilken M, Geraghty RJ, Li F. Mechanisms of Host Receptor Adaptation by Severe Acute Respiratory Syndrome Coronavirus. Journal of Biological Chemistry. 2012; 287:8904-8911. Disponible en: DOI: 10.1074/jbc.M111.325803.
19. Zhang H, Penninger JM, Li Y, Zhong N, Slutsky AS. Angiotensin‑converting enzyme 2 (ACE2) as a SARS‑CoV‑2 receptor: molecular mechanisms and potential therapeutic target. Intensive Care Med. (2020). Disponible en: DOI: 10.1007/s00134-020-05985-9.
20. Clarke NE, Turner AJ. Angiotensin-Converting Enzyme 2: The First Decade. International Journal of Hypertension. 2012; Article ID 307315, 12 pages. Disponible en: DOI: 10.1155/2012/307315.
21. Wrapp D, Wang N, Corbett KS, Goldsmith JA, Hsieh CL, Abiona O, et.al. Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation. Science. 2020; 367: 1260–1263. Disponible en: DOI: 10.1126/science.abb2507.
22. Yan R, Zhang Y, Li Y, Xia L, Guo Y, Zhou Q. Structural basis for the recognition of the SARSCoV-2 by full-length human ACE2. Science. 2020: eabb2762. Disponible en: DOI: 10.1126/science.abb2762.
23. Walls AC, Tortorici MA, Bosch BJ, Frenz B, Rottier PJ, DiMaio F. Cryo-electron microscopy structure of a coronavirus spike glycoprotein trimer. Nature. 2016; 531: 114-119. Disponible en: DOI: 10.1038/nature16988.
24. Yan R, Zhang Y, Guo Y, Xia L, Zhou Q. Structural basis for the recognition of the 2019-nCoV by human ACE2. bioRxiv. 2020: preprint 956946. Disponible en: DOI: 10.1101/2020.02.19.956946.
25. Villatoro FR. La estructura 3D de la glicoproteína espicular del coronavirus SARSCoV-2. NAUKAS. 2020 [citado el 24 de febrero de 2020]. Disponible en: https://francis.naukas.com/2020/02/24/la-estructura-3d-de-la-glicoproteina-espicular-del-coronavirus-sarscov-2/. [ Links ]