Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Acta Nova
On-line version ISSN 1683-0789
RevActaNova. vol.2 no.1 Cochabamba Dec. 2002
Actualidad Científica
El nuevo servidor Latinoamericano de Biología Molecular de la UCB: bo.expasy.org
Reynaldo Vargas Altamirano1, Javier Rojas Balderrama1, Christine Hoogland2, Elisabeth Gasteiger2, Ivan Ivanyi2, Amos Bairoch2, Ron D. Appel2, Denis Hochstrasser3
1 Instituto de Investigación en Informática Aplicada
Universidad Católica Boliviana
Cochabamba, Bolivia
2 Swiss Institute of Bioinformatics
Geneva, Switzerland
3 Laboratoire Central de Chimie Clinique
Hôpitaux Universitaires de Genève
Geneva, Switzerland
Resumen
ExPASy es un servidor de biología molecular que provee acceso a información en proteómica a través de un conjunto de herramientas de análisis y bases de datos dedicadas. Este servidor, desarrollado por el Instituto Suizo de Bioinformática (SIB), es pionero en su clase y actualmente se ha convertido en una de las referencias más consultadas por centros de investigación e industrias de biotecnología a nivel mundial. Sitios mirrors han sido implementados alrededor del mundo, en instituciones académicas y de investigación, para ofrecer un acceso eficiente a los centros de investigación y desarrollo en biología molecular geográficamente distribuidos.
La Universidad Católica Boliviana, mediante el Instituto de Investigación en Informática Aplicada (IIIA), en colaboración con el SIB, ha implementado un nuevo servidor mirror http://bo.expasy.org para la región latinoamericana que ha sido puesto a disposición de la comunidad científica a principios de noviembre 2002.
Este artículo tiene por objetivo presentar el contenido del sitio mirror y sus posibles aplicaciones para la investigación y la industria.
1. Introducción
El Instituto de Investigación en Informática Aplicada (http://iiia.ucbcba.edu.bo) ha sido nombrado el mirror oficial para Latinoamérica de ExPASy (Expert Protein Analysis System) [3], servidor de proteómica desarrollado por el Instituto Suizo de Bioinformática (http://www.sib-isb.ch). De esta manera se ha logrado un resultado importante del convenio suscrito entre ambas instituciones, en beneficio de la comunidad científica de latinoamerica y el mundo.
2. Instituto de Investigación en Informática Aplicada (IIIA)
El IIIA de la Universidad Católica Boliviana es un instituto dedicado a la investigación y desarrollo de aplicaciones informáticas. Entre los objetivos del Instituto están: impulsar la investigación en informática con el fin de responder a las crecientes necesidades de tecnología de la información; impulsar la transferencia de tecnología en informática a las empresas y universidades; colaborar con industrias e instituciones nacionales e internacionales a través de proyectos de investigación aplicada; establecer convenios y acuerdos con universidades extranjeras para el intercambio de estudiantes y docentes. Las actividades del IIIA apoyan campos académicos y profesionales bajo auspicios y convenios nacionales e internacionales.
Estos objetivos han permitido al IIIA participar en un proyecto de investigación y desarrollo tan importante como ExPASy (ver figura 1), facilitando a los investigadores y educadores en Bolivia, Latinoamérica e incluso el mundo entero, el acceso a una colección de herramientas de análisis y bases de datos dedicadas a la proteómica. El hecho de tener un servidor mirror de estas características se convierte en una realidad a partir de un esfuerzo notable, puesto que para su ejecución han sido necesarios contactos, infraestructura, soporte y desarrollo conjunto entre el IIIA, el SIB y otras instituciones. Es así que el servidor mirror ExPASy es administrado íntegramente por el IIIA y forma parte de la red latinoamericana de Bioinformática para América Latina y el Caribe (LacBioNet, http://www.lacbionet.org) [4].
Finalmente, es importante destacar que este proyecto de investigación y desarrollo (I+D) ha logrado introducir, en nuestra región, una nueva área de especialidad no solo para los informáticos e ingenieros de sistemas sino también para biólogos, bioquímicos y médicos: la Bioinformática [2].
3. La Bioinformática
El siglo XXI ha sido nombrado el siglo de la informática y la biotecnología, dado el gran interés en sus aplicaciones para la mejora de la calidad de vida de los habitantes de la tierra. Grandes centros de investigación alrededor del mundo han invertido recursos humanos y tecnología de punta para la realización de proyectos como el "Human Genome Project"[6] para el secuenciamiento completo del genoma humano. Actualmente, los resultados de estas investigaciones han generado enormes cantidades de información, produciendo bases de datos complejas que necesitan el concurso de especialistas en una nueva disciplina denominada Bioinformática [2].
La Bioinformática estudia las técnicas y herramientas computacionales para resolver y sistematizar los problemas ligados al manejo de la información de biología molecular permitiendo almacenar, recuperar, procesar, analizar, buscar, comparar e identificar la composición y estructura de las moléculas biológicas. En este sentido, el bioinformático realiza tareas diversas y variadas incluyendo:
Adquisición, almacenamiento y visualización de datos.
Procesamiento, análisis y gestión de datos de secuencias de proteínas.
Generación, integración, ensamblaje de secuencias.
Predicción de estructura de proteínas.
Software para alineamiento, comparación y clasificación de secuencias de proteínas.
Desarrollo de Bases de Datos que contengan datos y documentos, con acceso privado o público a través del Internet o de redes privadas.
Automatización del secuenciamiento y la experimentación.
Predicción de funciones de secuencias genéticas.
Desarrollo de motores de búsqueda en bases de datos estructuradas y no estructuradas.
Determinación y predicción de las estructuras primaria, secundaria y terciaria de las macromoléculas.
Precisamente, los bioinformáticos tienen una formación híbrida entre la informática y la biología que les permite entender los principios de la biología molecular y desarrollar herramientas informáticas para tratar este tipo de problemas. Las aplicaciones de esta nueva disciplina representan un gran interés científico y económico en distintas áreas de investigación y producción incluyendo aplicaciones farmacéuticas, industrias agrícolas y de alimentos, aplicaciones en medicina y otras.
4. Servidor ExPASy
El Servidor ExPASy (http://www.expasy.org) es un servidor Web que provee servicios a la comunidad dedicada al estudio e investigación de las Ciencias de la Vida [1]. Permite a sus usuarios acceder a una gran variedad de bases de datos especializadas en distintos rubros enfocados a lo que se denomina proteómica1 [7]. Además, presta servicios a través de un conjunto de herramientas de software de análisis y consulta. El servidor ExPASy, que inició sus actividades en 1993 en el Hospital Universitario de Ginebra (http://www.hcuge.ch), es uno de los servidores pioneros en su género. Posteriormente, ha sido desarrollado por el SIB por un equipo multidisciplinario y funciona ininterrumpidamente desde Agosto de 1993, alcanzando en noviembre del 2002, a 227 millones de consultas por Internet a través de 2.9 millones de computadoras desde 185 países diferentes. De esta manera, ExPASy se ha convertido en una de las referencias más grandes a nivel mundial [5] en el área de la proteómica, sirviendo a varios centros de investigación, empresas dedicadas a la biotecnología, universidades, hospitales y personas particulares interesadas. A continuación presentaremos una descripción resumida de los recursos disponibles en ExPASy.
4.1. Base de Datos
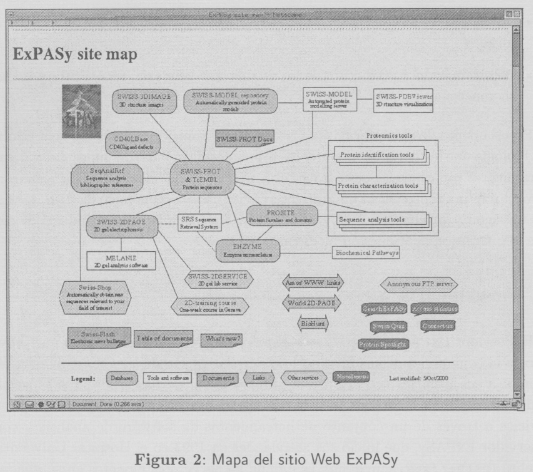
ExPASy aloja varias bases de datos que han sido parcial o totalmente desarrolladas por el SIB, constituyendo el corazón del sitio Web (ver figura 2), entre las que se destacan:
La base de conocimientos SWISS-PROT. Es una base de datos de secuencias de proteínas cuidadosamente elaborada, proporciona anotaciones de alta calidad (descripción de la función de una proteína, estructura, referencias, etc.) con un mínimo de redundancia y un alto nivel de integración con las otras bases de datos.
SWISS-2DPAGE. Contiene información de proteínas identificadas en geles de Electroforesis Bidimensional que son mapas de concentraciones proteicas.
PROSITE. Base de datos de dominios y familias de proteínas. PROSITE contiene patrones y especificaciones que ayudan a la identificación de familias de proteínas conocidas y nuevas secuencias que puedan pertenecer a éstas.
SWISS-3DIMAGE. Incluye imágenes de alta calidad con anotaciones de macro-moléculas biológicas con estructura tridimensional conocida.
SWISS-MODEL. Base de datos de modelos estructurales de proteínas generados automáticamente.
Una variedad de opciones de acceso y formatos de presentación están disponibles. Estas opciones permiten a los usuarios mostrar y recuperar subconjuntos específicos de las bases de datos. Para complementar estas opciones se tiene también implementado un servidor llamado SRS que permite búsquedas complejas hechas combinando campos de las diferentes bases de datos.
Un gran número de documentos (manuales de usuarios, notas de releases, índices, nomenclaturas, documentos técnicos) están disponibles en ExPASy y han sido complementados con una variedad de referencias en línea. Todas las bases de datos disponibles en ExPASy están siendo extensivamente referenciadas por otras bases de datos de biología molecular u otros recursos afines. Por ejemplo, SWISS-PROT es referenciada a través del Internet por más de 50 bases de datos diferentes alrededor del mundo. SWISS-PROT es actualizado con una frecuencia de aproximadamente dos semanas. Las otras bases de datos son periódicamente periódicamente.
Cada base de datos de ExPASy (los datos, información y documentación asociada) puede ser localmente copiada mediante servicio de FTP anónimo. Por otra parte, se tiene el cuidado de distribuir los archivos de datos para poder construir una base de datos completa y no-redundante.
Gracias al hardware disponible es posible ejecutar búsquedas avanzadas de similitud de alta velocidad en las bases de datos de proteínas contenidas en ExPASy.
El uso de todas las bases de datos es gratuito para usos académicos. Sin embargo, se ha implementado un sistema de suscripción anual con costo para usos comerciales. La empresa GeneBio (http://www.genebio.com) está encargada de la administración y comercialización de las licencias comerciales. Los recursos obtenidos son usados para poder mantener en funcionamiento estas bases de datos, actualizarlas, extenderlas y mejorar su calidad y servicios.
4.2. Herramientas de software para el análisis
El SIB ha desarrollado una gran colección de herramientas, basadas en tecnología Internet, que son usadas para poder acceder o mostrar resultados de consultas hechas a las bases de datos o analizar secuencias de proteínas o datos de proteómica originados por experimentos de espectrometría de masas. Estas herramientas pueden ser consultadas en su totalidad desde ExPASy y se dividen en once categorías: identificación y caracterización de proteínas, traducción de DNA a secuencias de proteínas, búsquedas de similitud, búsquedas de patrón y de perfil, predicción post-translacional, topologías de predicción, análisis de estructuras primarias, predicción de estructuras secundarias, estructuras terciarias, alineamiento de secuencias y análisis de textos biológicos.
Una característica muy importante de las herramientas de software (como PeptIdent, TagIdent, MultiIdent, PeptideMass, FindPept o FindMod) es que utilizan las anotaciones de las entradas de SWISS-PROT para considerar modificaciones post-translacionales así como variantes de secuencias para realizar predicciones.
Algunas de las herramientas (SWISS-MODEL, Swiss-Shop o AAComSim) reportan sus resultados por correo electrónico mientras que otras muestran resultados directamente en línea incluyendo datos y gráficos.
Todas estas herramientas son listadas en una página de ExPASy que también ofrece enlaces a otros muchos programas de análisis de secuencias de proteínas disponibles en otros sitios.
4.3. ExPASy como un portal a otros recursos de Ciencias de la Vida
El volumen de información disponible en Internet ha cambiado completamente el acceso a la información y la manera en que los científicos y profesionales procesan los datos biológicos. Esto ha extendido considerablemente el uso de los servidores, creando muchas oportunidades y posibilidades de servicios pero también ha traído consigo nuevos problemas. Una de las dificultades más críticas es la de distinguir los recursos de información actualizada y útiles de los sitios que proveen información de baja calidad, desactualizada y errónea. Para solucionar parcialmente este problema, se han desarrollado una serie de listas y herramientas ubicadas también en ExPASy:
Amos' WWW links page. Es una lista que contiene enlaces a una gran cantidad de recursos útiles para las ciencias de la vida. Esta lista esta organizada en un cierto número de secciones que corresponde a temas específicos y es actualizada constantemente.
W0RD-2DPAGE. Es una lista de todos los servidores WWW de bases de datos conocidas relacionadas a la técnica de Electroforesis Bidimensional.
BioHunt. Es Un servicio de búsqueda de información de biología molecular. Actualmente BioHunt indexa más de 20.000 documentos.
2DHUNT. Es un índice especializado de sitios relacionados a la técnica de Electroforesis Bidimensional.
4.4. Otras características interesantes de ExPASy
ExPASy contiene además de toda la información descrita previamente otro conjunto de información útil, interesante o recreacional:
Biochemical pathways: una versión indexada del póster de Boehringer Mannheim' s' Biochemical Pathways que puede ser accedida gráficamente y permite a los usuarios navegar por medio de una representación de rutas metabólicas y enlazado a la base de datos ENZIME.
DeepView: una aplicación que se ejecuta en distintas plataformas y que ofrece un amplio rango de opciones de visualización y manipulación de estructuras de proteínas.
LALNVIEW: una aplicación multiplataforma que permite una visualización gráfica de alineamiento de pares de secuencias de proteínas.
2-D PAGE: conjunto extenso de información concerniente a Electroforesis Bidimensional que incluye la descripción completa de los protocolos experimentales como también de una revisión de Melanie, que es un software de análisis y procesamiento de imágenes de geles.
Protein Spotlight: un suplemento periódico enfocado en proteínas o grupos de proteínas.
Además, los usuarios pueden tomar una pequeña pausa, visitando el Swiss-Quiz o los Swiss-Jokes. Con Swiss-Quiz uno tiene la oportunidad de ganar algunos chocolates suizos después de responder satisfactoriamente un examen en el campo de la biología molecular. Swiss-Jokes provee acceso a una colección de chistes y aforismos en áreas de las ciencias de la vida y computación.
5. Desarrollos y aportes del IIIA
A partir de enero de 2000 se han realizado varios aportes al servidor ExPASy en el IIIA. En primer lugar se ha realizado la migración y consolidación total del servidor sobre una plataforma Linux a partir de la plataforma Sun Solaris y SGI Irix (Silicon Graphics). En este sentido se han adecuado todas las funciones y herramientas a la nueva plataforma Red Hat Linux. Para el lanzamiento oficial del nuevo sitio mirror, se han realizado pruebas de validación y ajustes para que el servidor ExPASy sea publicado mundialmente como sitio mirror latinoamericano conforme a las normas vigentes y establecidas por el SIB. Este lanzamiento ha sido realizado a principios del mes de noviembre de 2002. El nuevo servidor instalado en los ambientes del Instituto tiene las siguientes características:
Dos procesadores Pentium Xeon de 1.8 GHz.
106 GBytes de Memoria de Almacenamiento.
1 GByte de RAM.
Conexión Internet de 512 Kbps.
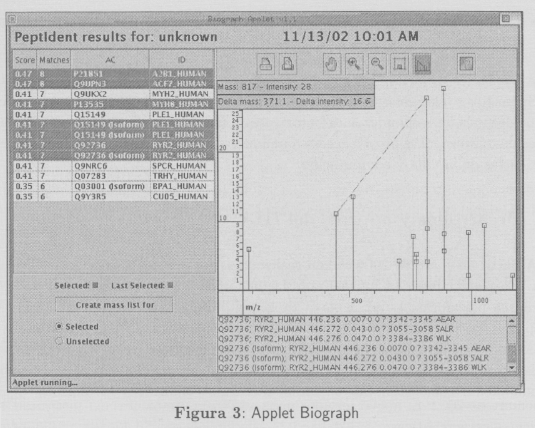
Paralelamente se han realizado desarrollos de nuevas herramientas que han sido completamente implementadas en el IIIA e incorporadas en el sitio ExPASy. Uno de los desarrollos más sobresaliente es Biograph que es una herramienta de visualización y manipulación de espectros de masa correspondientes a secuencias proteicas. El desarrollo de esta nueva herramienta fue realizado en el lenguaje JAVA bajo forma de un applet usando la tecnología Java Web Start (http://java.sun.com/products/javawebstart). Las funcionalidades de esta herramienta incluyen el análisis de espectros de masa, reportes, gestión de grupos de espectros, zooming, printing. Esta aplicación es principalmente usada por los químicos y biólogos en varias aplicaciones biotecnológicas incluyendo la búsqueda y creación de nuevos medicamentos farmacéuticos y terapéuticos. La figura 3 ilustra esta aplicación gráfica que puede ser manipulada a través de un navegador.
6. Conclusión
El Instituto de Investigación en Informática Aplicada de la Universidad Católica Boliviana ha preparado un nuevo servidor mirror de biología molecular: el servidor latinoamericano ExPASy. Los servicios y la información contenida este servidor están libremente disponibles a partir de noviembre del 2002 para toda la comunidad científica interesada en proteómica. Este nuevo servidor incluye nuevas herramientas de análisis desarrolladas en la UCB como la aplicación Biograph Web de análisis de espectros de masa de secuencias de proteínas.
7. Agradecimientos
Los agradecimientos van dirigidos al Prof. Dr. Ron D. Appel del Instituto Suizo de Bioinformática y al Prof. Dr. Denis Hoschtrasser del Laboratorio Central de Química Clínica del Hospital Universitario de Ginebra, por el apoyo científico y financiero a este trabajo; van dirigidos también a la Universidad Católica Boliviana, en especial al Dr. Hans van den Berg.
Notas
1En la actualidad la secuencia completa de varios genomas, incluyendo el humano, es conocida. Sin embargo, el entendimiento de por lo menos medio millón de proteínas humanas codificadas en alrededor de 30000 genes es todavía una larga y dura travesía por recorrer. Es así que una nueva disciplina, la proteómica (PROTEina complementada a genOMA), ha emergido recientemente, para descifrar los mecanismos bioquímicos y fisiológicos de procesos multivariables a nivel molecular. La proteómica puede ser definida, como la comparación cuantitativa y cualitativa bajo diferentes condiciones para descifrar los componentes biológicos relacionados a este campo.
Referencias
[1] Ron D. Appel, Amos Bairoch, y D. F Hochstrasser. A new generation of information retrieval tools for biologist: the example of the ExPASy WWW server. Trends Biochem. Sci., (19):258-260, 1994.
[2] T.K. Attwood y D.J. Parry-Smith. Introduction to Bioinformatics. Addison Wesley Longman Higher Education, Essex, 1999.
[3] Amos Bairoch, Elisabeth Gasteiger, Alexandre Gattiker, Christine Hoogland, Corinne Lachaize, Khaled Mostaguir, Ivan Ivanyi, y Ron D. Appel. The ExPASy proteome WWW server in 2002. Reporte técnico, Swiss Institute of Bioinformatics, November, 2002.
[4] Wim Degrave. A bioinformatics network for latin america an the caribbean. http://biolac.unu.edu/spanish/proyecto3.htm, 2002.
[5] Junhyong Kim. Computers are from Mars, organisms are from Venus. Computer, 35(7), July, 2002. [ Links ]
[6] U.S. Department of Energy Office of Science, Office of Biological and Environmental Research, y Human Genome Program. Human Genome Project Information. http://www.ornl.gov/hgmis/. [ Links ]
[7] M. R. Wilkins, K. L. Williams, R. D. Appel, y D.F Hochstrasser, editores. Proteome Research: New Frontiers in Functional Genomics. Principies and Practice. Springer, Berlin, 1997.