Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO  uBio
uBio
Share
Cuadernos Hospital de Clínicas
Print version ISSN 1562-6776
Cuad. - Hosp. Clín. vol.59 no.Especial La Paz 2018
ARTICULOS ORIGINALES
Asociación genética entre los loci hla-drb1 y hla-dqb1 con la susceptibilidad a padecer lupus eritematoso sistémico
Genetic association between the loci hla-drb1 and hla-dqb1 with susceptibility to suffer from systemic lupus erythematosus
Cabrera-Calzadilla S1, Sosa-Tordoya L F1, 2, Terán de Baudoim M de A3, 4, Plata-Cornejo R4
1Instituto de Servicios de Laboratorio de Diagnostico e Investigación en Salud (SELADIS), Facultad de Ciencias
Farmacéuticas y Bioquímicas (FCFB), Universidad Mayor de San Andrés (UMSA)
2Docente Investigador del Instituto SELADIS, Facultad de Ciencias Farmacéuticas y Bioquímicas, UMSA
3Docente de la Facultad de Medicina, Cátedra de medicina III, Capítulo de nefrología, UMSA
4 Instituto de Nefrología BOL SRL.
Autor para correspondencia: Dr. Sergio Cabrera Calzadilla, scabrerac88@gmail.com Dirección: Av. Saavedra N° 2224 (Instituto SELADIS) Piso 6, Ciudad Nuestra Señora de La Paz, Departamento de La Paz, Bolivia(591 -2 612448)
RECIBIDO: 03/07/2018 ACEPTADO: 28/09/2018
ABSTRACT
Immunogenetics research provides information on genetic markers associated with autoimmune diseases such as systemic lupus erythematosus (SLE), you can then observe certain risk factors or protection to the disease in a given population. To determine the genetic association between polymorphisms ofthe Major Histocompatibility Complex loci represented by the HLA-DRB1 and HLA-DQB1 with susceptibility to SLE.
METHODOLOGY: We worked with 85 lupus patients and 85 patients without the disease; Human DNA was obtained from peripheral blood, PCR-SSP low and high resolution molecularly performed to establish the loci HLA-DRB1 and HLA-DQB1. Allele frequencies, which were associated with both samples using the Odds Ratio at a level of significance of 5% were determined.
RESULTS: Results of PCR-SSP low-resolution HLA show that no predisposing allele plays a role, we observed that HLA-DRB1*04 allele has a protective role OR=0.49 (p=0.03). The PCR-SSP results of high resolution show that the HLA-DRB1*03:01 alleles (OR=18.3; p=0.007), DRB1*04:04 (OR=4.2; p=0.009), DRB1*09:01 (OR=18.3; p=0.007), HLA-DQB1*03:03 (OR=18.8; p=0.006) and DQB1 *02:01 (OR=21.2; p=0.003) are risk factors. We demonstrated that HLA-DRB1 *08:02 alleles (OR=0.42; p=0.003) and HLA-DQB 1*04:02 (OR=0.50; p=0.02) are ofa protective nature.
CONCLUSIONS: The alleles representing LES risk in the study sample are HLA-DRB1*03:01, *04:04, *09:01 and HLA-DQB1*03:03, *02:01. The alleles having a protective character to the disease are HLA-DRB 1 *08:02 and HLA-DQB1 *04:02.
KEYWORDS: Human Leukocyte Antigen, Systemic Lupus Erythematosus, Genetics Association, HLA-DRB1, HLA-DQB1.
RESUMEN
La investigación en Inmunogenética brinda información acerca de marcadores genéticos asociados con enfermedades autoinmunes, como el Lupus Eritematoso Sistémico (LES), se puede observar entonces ciertos factores de riesgo o protección hacia la enfermedad en una población determinada.
OBJETIVO: Determinar la asociación genética entre los polimorfismos del Complejo Principal de Histocompatibilidad (CPH) representados por los loci HLA-DRB1 y HLA-DQB1 con la susceptibilidad a LES.
METODOLOGÍA: Se trabajó con 85 pacientes lúpicos y 85 pacientes sin la enfermedad; se obtuvo DNA humano a partir de sangre periférica, se realizó un PCR-SSP de baja y alta resolución para tipificar molecularmente a los loci HLA-DRB1 y HLA-DQB1. Se determinó las frecuencias alélicas, las cuales fueron asociadas con ambas muestras mediante el uso del Odds Ratio, a un nivel de significancia del 5 %.
RESULTADOS: Los resultados del PCR-SSP de baja resolución muestran que ningún alelo HLA tiene un rol predisponente, se observó que el alelo HLA-DRB1*04 presenta un rol protector OR=0,49 (p=0,03).
Los resultados por PCR-SSP de alta resolución muestran que los alelos HLA-DRB1*03:01 (OR=18,3; p=0,007), DRB1*04:04 (OR=4,2; p=0,009), DRB1*09:01 (OR=18,3; p=0,007), HLA-DQB1*03:03 (OR=18,8; p=0,006) y DQB1*02:01 (OR=21,2; p=0,003) son factores de riesgo. Se evidenció que los alelos HLA-DRB1*08:02 (OR=0,42; p=0,003) y HLA-DQB1*04:02 (OR=0,50; p=0,02) son de carácter protector.
CONCLUSIONES: Los alelos que representan riesgo de padecer LES en la muestra estudiada son HLA-DRB1*03:01, 04:04, 09:01 y HLA-DQB1*03:03, 02:01. Los alelos que tiene un carácter protector a la enfermedad son HLA-DRB1*08:02 y HLA-DQB1*04:02.
PALABRAS CLAVE: Antígeno Leucocitario Humano, Lupus Eritematoso Sistémico, Asociación Genética, HLA-DRB1, HLA-DQB1.
INTRODUCCIÓN
El LES en una enfermedad autoinmune sistémica con manifestaciones clínicas heterogéneas debido a que diferentes órganos, tejidos y células son dañadas por la acción auto-anticuerpos, depósito complejos inmunitarios y activación del complemento. En la mayoría de los pacientes se evidencia la presencia de auto-anticuerpos años antes de que aparezca el primer signo clínico. Si bien las personas de cualquier edad, grupo étnico y género son susceptibles a la enfermedad, el noventa por ciento de los pacientes son mujeres en edad reproductiva.
Se ha planteado como posible etiología del LES, la confluencia de factores genéticos, ambientales y hormonales. Los factores genéticos son de suma importancia para determinar la susceptibilidad al LES y la complejidad en las manifestaciones clínicas de la enfermedad1; es por esto que se ha estudiado y propuesto un gran número de genes candidatos a predisponentes de la enfermedad, entre los cuales los genes del CPH representan los loci genómicos más fuertemente asociados al riesgo de padecer LES2. En la región del CPH, los genes del antígeno leucocitario humano (HLA), tienen importantes funciones en la activación y regulación del sistema inmune (SI), han sido asociados con el desarrollo del LES; entre ellos se encuentran los genes del HLA-DRB1 y HLA-DQB1, que forman parte del CPH clase II.
Muchos estudios han determinado que entre los factores genéticos, el grupo étnico reviste importancia en la susceptibilidad a LES, y el polimorfismo genético de los genes asociados puede variar en las diferentes poblaciones del mundo. Como ejemplo en la población afro descendiente se ha observado una mayor predisposición a LES3.
Por lo antes expuesto, se hace necesario caracterizar genéticamente los pacientes con LES en diferentes regiones o poblaciones del mundo.
Varios estudios realizados en el genoma humano de familias que viven con LES han identificado un gran número de regiones que pueden contener genes de susceptibilidad.
La búsqueda de genes que predisponen a una persona a desarrollar LES se ha hecho a través de los estudios de asociación de genes candidatos y el análisis de la frecuencia alélica de los loci HLA clase II, que han tenido un éxito mensurable en las últimas décadas.
La identificación de genes candidatos y el conocimiento de las influencias genéticas nos permiten comprender la fisiopatología de esta enfermedad autoinmune. No existen estudios sobre la asociación de los polimorfismos HLA de clase II en pacientes bolivianos con LES, este es el pionero de muchos otros análisis que deben realizarse, esto es muy importante ya que existe heterogeneidad genética debido al genoma mestizo que la gran mayoría de la población boliviana tiene.
Los resultados del presente estudio brindan un apoyo en la toma de decisiones para el diagnóstico y seguimiento clínico del LES, como también aplicar protocolos de medicina preventiva en familiares de pacientes con LES. Asimismo, se podrá comparar si los alelos encontrados con susceptibilidad a LES son los mismos que se han reportado en otros estudios hechos a nivel Latinoamericano y Mundial.
El presente estudio pretende determinar la asociación de los polimorfismos alélicos de los loci genéticos HLA-DRB1 y HLA-DQB1 con la susceptibilidad a LES en una muestra atendida en el Instituto SELADIS de la UMSA.
METODOLOGÍA
El presente estudio de casos y controles, involucro a 85 pacientes confirmados clínicamente con LES y 85 pacientes sin la enfermedad, todos fueron atendidos en inmediaciones del Instituto SELADIS - FCFB - UMSA. Se obtuvo 4 mL de sangre periférica con EDTA-K3, se realizó la extracción de DNA humano de acuerdo al protocolo propuesto por el kit Wizard® Genomic DNA Purification (PROMEGA, USA), conservando las muestras rehidratadas a 4°C y las muestras de DNA seco a -20°C.
La cuantificación de DNA se realizó por el método de espectrofotometría UV, obteniéndose un rango de concentración de DNA entre 100 a 200 ng/uL. Las relaciones de D.O. (densidad óptica) en las muestras diluidas leídas a 260/280 nm estuvieron entre 1,8 y 1,9 considerándose así, la concentración y calidad de DNA extraído aptos para su estudio.
Posteriormente se realizó la tipificación de los genes del Complejo Principal de Histocompatibilidad mediante la técnica de PCR-SSP (del inglés, Secuence Specific Primer)de baja y alta resolución, según el protocolo propuesto por los kits de tipificación HLA AllSet+TM Gold SSP (Invitrogen Corporation, USA)tanto para los genes HLA-DRB1 y HLA-DQB1.
Se realizó la corrida electroforética horizontal en gel de agarosa al 2 %, se utilizó el colorante de ácidos nucleicos SYBR® Gold (10000x) (Molecular Probes, USA). Pasado el tiempo de corrida los geles fueron revelados con la ayuda de un transiluminador de luz azul SafeImagerTM 2.0 (Invitrogen, USA) y todos los resultados fueron documentados mediante fotografías.
Se obtuvieron las frecuencias alélicas tanto por las técnicas de PCR-SSP de baja y alta resolución para ambos grupos muestrales.
MUESTRA
Tamaño de la muestra
El tamaño de la muestra fue definido por conveniencia, estaba sujeto a los criterios de inclusión y exclusión del estudio y a la capacidad de financiamiento del mismo, obteniendo de esa manera a un 75 % de pacientes que provienen de la ciudad de La Paz y el restante al interior y exterior del país, considerándolo de esta manera como una muestra de pacientes mixta con alta proporción de población paceña. Estos mismos acudieron al Laboratorio de Histocompatibilidad e Inmunogenética del Instituto de Servicios de Laboratorio de Diagnóstico e Investigación en Salud (SELADIS) durante el periodo entre noviembre de 2012 a febrero de 2014.
Se incorporaron al estudio 85 pacientes lúpicos (CASOS) y a 85 pacientes sin la enfermedad (CONTROLES). Una vez realizado el PCR-SSP de baja resolución y conocidos los alelos de riesgo o protección, se construyó un nuevo grupo de estudio constituido por50 pacientes casos y 30 pacientes controles para la tipificación por PCR-SSP de alta resolución, todo esto debido al presupuesto y financiamiento asignado al presente estudio.
ESTADÍSTICA
- Software
La sistematización, análisis y cálculos de los datos estadísticos se realizaron con la ayuda de los paquetes estadísticos SSPS 20.0 Versión de prueba, GenAlEx 6.501 y Case-Control StudyTable (versión1/12).
- Parámetros Inferenciales
Los estimadores de grado de asociación que se utilizaron fueron el Chi-cuadrado de Pearson y el Odds Ratio (O.R.), con sus correspondientes intervalos de confianza. El cálculo se realizó a partir de las frecuencias antes mencionadas obtenidas a partir del grupo de pacientes caso y control. Se utilizó un 5% de significancia.
ÉTICA
Todos los pacientes que formaron parte del presente estudio fueron informados acerca de los objetivos, alcance, riesgos y beneficios del mismo, procediéndose con el registró del consentimiento informado según lo estipulado en el aval ético, otorgado por la Comité de Ética en Investigación de la UMSA. Los pacientes no pagaron ningún monto económico por la realización de las pruebas serológicas o moleculares.
RESULTADOS
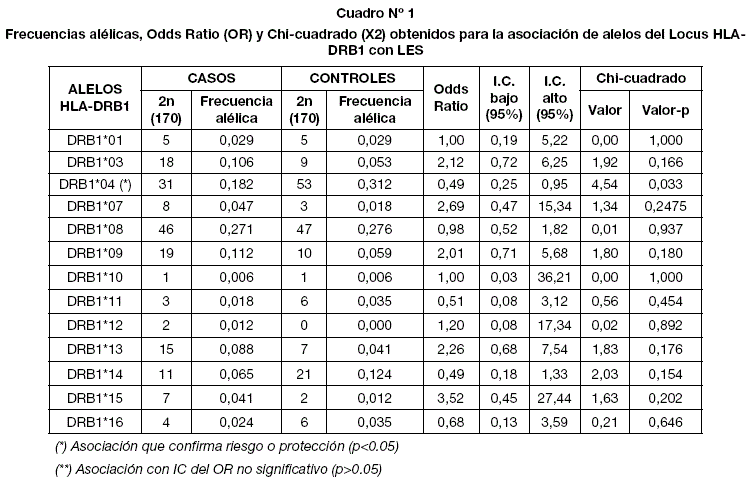
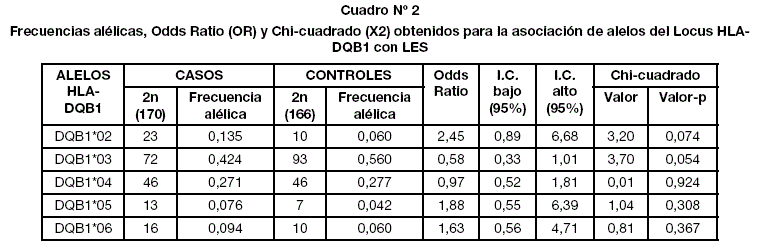
A partir de las frecuencias alélicas obtenidas de los dos grupos muestrales estudiados se pudo realizar el análisis de la asociación genética por cada alelo de los genes estudiados. Dentro el análisis por PCR-SSP de baja resolución se puede apreciar (Cuadro 1) al alelo HLA-DRB1*04 con un OR de 0,49(p=0,033), que se muestra como un alelo de protección para el LES. Asimismo, ningún alelo DQB1 presentó resultados estadísticamente significativos de asociación genética (Cuadro 2).
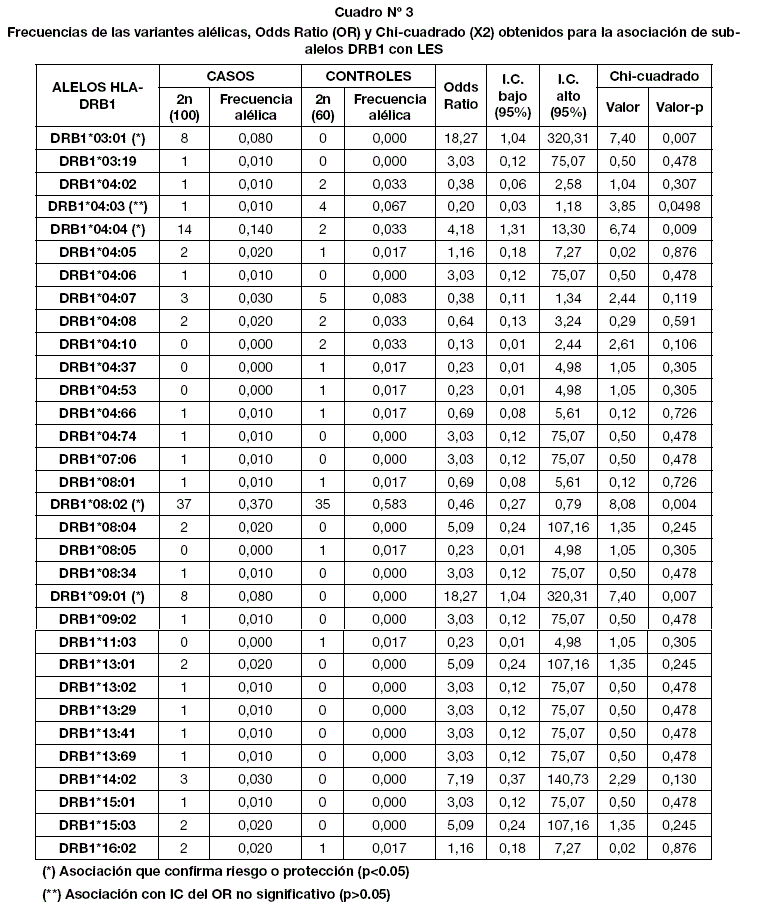
Para el análisis por métodos de PCR-SSP de alta resolución se seleccionó a los pacientes que poseían los alelos HLA-DRB1*04, HLA-DRB1*08, HLA-DQB1*03 y HLA-DQB1*04 este último por los antecedentes de riesgo a la enfermedad. El análisis de las variantes alélicas por alta resolución muestra en el caso del locus DRB1 (Cuadro 3) que los alelos HLA-DRB1*03:01, HLA-DRB1*04:04, HLA-DRB1*09:01, presentan un OR=18,27 (p=0,007), OR = 4,18 (p=0,009) y OR = 18,27 (p=0,007) respectivamente, asociándose así como alelos de riesgo a padecer el Lupus Eritematoso Sistémico en la población estudiada siendo los alelos HLA-DRB1*03:01 y HLA-DRB1*09:01 los más importantes.
También se pudo evidenciar que el alelo HLA-DRB1*08:02 presenta un OR = 0,46 (p=0.004), asociándose como alelo de protección contra el Lupus Eritematoso Sistémico en la población estudiada.
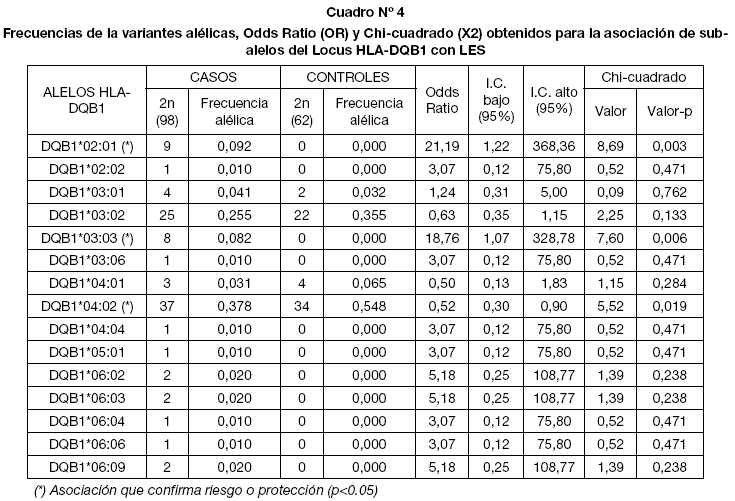
En el caso del locus DQB1 (Cuadro 4), los alelos HLA-DQB1*03:03 y HLA-DQB1*02:01 OR=18,76 (p=0,006) y OR = 21,19 (p=0,003) se asocian como alelos que generan riesgo a padecer LES, siendo el más importante HLA-DQB1*02:01. Además se puede evidenciar que el alelo HLA-DQB1*04:02 con un OR =0,52 (p=0,019), es un alelo que brinda protección a sufrir LES.
DISCUSIÓN
El lupus eritematoso sistémico es una enfermedad de etiología muy compleja en la que los factores genéticos son parte de la etiopatogenia, este aspecto ha sido demostrado por estudios genéticos de alta complejidad como el análisis GWAS4 en el que se reporta a un conjunto de genes candidatos, en este estudio se identificaron 29 loci genéticos que muestran asociación con LES, dentro de estos se encuentran los loci HLA-DRB1 y HLA-DQB1.
También la asociación genética se ve reflejada en estudios de agregación familiar, en un meta-análisis5, de un total de 44 artículos seleccionados se demostró que las enfermedades autoinmunes prevalecen en las familias, siendo las más importantes la agregación de la enfermedad tiroidea autoinmune, seguida por lupus eritematoso sistémico y la artritis reumatoide.
La complejidad genética del LES tiene probablemente un gran paralelismo con su gran heterogeneidad en las manifestaciones clínicas y presencia de auto-anticuerpos. Este hecho implica la necesidad del análisis de los factores genéticos que determinan la expresión de la enfermedad en diferentes grupos más homogéneos con el fin de obtener resultados más concluyentes. Los hallazgos de este trabajo así lo apoyan.
Los resultados del presente estudio reflejan a los alelos HLA-DRB1*03:01, *04:04, *09:01, DQB1*03:03, *02:01 como asociados positivamente a LES (factores de riesgo), y los alelos HLA-DRB1*08:02 y DQB1*04:02 asociados negativamente (factores protectores). A nivel internacional, se indica que los HLA DRB1*15, DRB1*16, DRB1*03 y DQB1*02 son los más importantes al asociarse positivamente a LES2, 6, tal como lo describe un estudio de casos y controles realizado en México por Thibault y cols. (2008) donde encontraron una asociación positiva de los alelos DRB*03:01, *15:01 y *14:01 en pacientes lúpicos; en el Reino Unido el grupo de investigadores de Mc Hugh (2006), aseveran lo anteriormente mencionado, añadiendo al alelo DQB1 *02:01 como factor de riesgo a padecer LES y al alelo DQB1*03:03 como factor de protección, este último no concuerda con los resultados obtenidos en el presente estudio, debido a que este alelo se expresa como factor de riesgo; por ello la importancia de realizar estos estudios en diferentes poblaciones o regiones demográficas.
En un meta-análisis realizado en Europa7 se establece como alelos de riesgo a HLA-DRB1*03:01, HLA-DRB1*08:01, y HLA-DQA1*01:02, además dos SNPs de la región HLA, rs8192591 y rs2246618, el estudio de los polimorfismos de los SNPs abre un nuevo campo de acción en la investigación de enfermedades autoinmunes.
Se puede evidenciar que2 el antígeno HLA DR2 (alelos DRB1*15 y DRB1*16) está fuertemente asociado a LES. Sin embargo, en la población estudiada en el presente estudio la frecuencia alélica de estos fue baja, para pacientes lúpicos 0.041 (DRB1*15) y 0.024 (DRB1*16), y para controles 0.012 (DRB1*15) y 0.035 (DRB1*16); lo que conllevó a revisar un estudio de frecuencias alélicas del gen HLA en nuestra población.
En población boliviana en una muestra de 542 pacientes, Cruz (2011) estableció que los alelos DRB1*15 (frecuencia de 0.024) y DRB1*16 (frecuencia de 0.037) son alelos de baja frecuencia de expresión en nuestra población. En ese estudio se encontró que los alelos más frecuentes son HLA-DRB1*04 (0.244), HLA-DRB1*08 (0.172), HLA-DRB1*14 (0.105) y HLA-DRB1*09 (0.101 )8; por lo tanto, los resultados del presente estudio no contradicen lo antes mencionado, aunque de todos modos, estos alelos son dos veces más frecuente en pacientes lúpicos con respecto a la frecuencia general encontrada en nuestra población. Sin embargo, considero que esto puede ser de importancia, ya que es muy probable que estos alelos (DRB1*15 y DRB1*16) no estén asociados como factores de riesgo a lupus en nuestra población debido a su baja frecuencia; se aclara de que estos alelos revisten importancia como factores de riesgo en caso de pacientes de procedencia caucásica, asiática o africana.2, 9, 10
Un amplio estudio llevado a cabo por11 reveló que en Malasia los alelos HLA-A*11:01, *11:02, DQB1*05, DQB1*03:01, *03:04, DRB1*07:01 se asocian significativamente con LES en los malasios; mientras que en China los alelos DRB1*16:01 -*16:06 y DQB1*05 fueron significativamente mayores en pacientes con LES. La investigación reveló que estos dos locus DRB1 y DQB1 pueden ser específicos y representativos para los dos grupos poblacionales y que DQB1 *05 podría ser el alelo común de susceptibilidad en la población estudiada. Sin embargo, lo que indica el presente estudio es que los alelos DQB1*03:03 y *02:01 son de riesgo y el alelo DQB1*04:02 de protección, considerando que sea muy probable que el origen de nuestro árbol antropológico sea de Asia, estos resultados genéticos indican, una vez más, que el CPH al ser muy polimórfico es importante analizarlo en diferentes regiones demográficas.
En un meta-análisis12 que integró estudios realizados en pacientes latinoamericanos con LES (2 estudios de Brasil, 7 de México, 1 de Colombia y 1 con Méxicano-Americanos), se encontraron en los pacientes los siguientes alelos del HLA-DQB1, en México: HLA-DQB1*03:03, HLA-DQB1*04:02 y HLADQB1*05:01; en mexicano-americanos: HLA-DQB1*04:02; en Brasil: no se encontraron; en Colombia: HLA-DQB1*03:02 y HLA-DQB1*05:01. Dicho análisis reflejó que el alelo DQB1*03:03 encontrado en población mexicana, se correlaciona con los resultados obtenidos en el presente estudio, por el contrario, DQB1*04:02 en población mexicano-americana se muestra como alelo de riesgo, mientras que en población boliviana este se manifiesta como alelo de protección.
Otro alelo que resalta en el presente estudio es el DRB1*09:01, obteniéndose correlación con la población nipona, Shimane y cols. encontraron a losalelosHLA-DRB1*15:01,*09:01,*08:02y*04:01 altamente asociados a LES, y particularmente el alelo HLA-DRB1*09:01 también se encuentra en la artritis reumatoide, lo que conjuntamente se denomina rhupus.13
En el presente estudio se puede demostrar con claridad la importancia de la metodología por alta y baja resolución, ya que en el análisis de baja resolución, el alelo HLA-DRB1*04 se lo considera como de protección, pero en el análisis de alta resolución, se aclara el panorama, ya que la variante alelica HLA-DRB1*04:04 se manifiesta como riesgo, en cambio la variante alelica HLA-DRB1*04:03 se manifiesta como un alelo de protección contra el LES.
CONFLINTO DE INTERESES
Los autores declaramos no presentar ningún conflicto de interés para la realización y/o publicación del presente artículo.
AGRADECIMIENTOS
A nuestras familias, el esfuerzo que se realiza es impulsado por nuestras queridas familias.
A la UMSA, ya que sin el aporte del recurso económico mediante el IDH, nada de esto se hubiera realizado.
Al personal del Laboratorio de Histocompatibilidad e Inmunogenética del Instituto SELADIS, Facultad de Medicina y Personal de la UAGRM; Dr. Jose Luis Choquehuanca, Dr. Leo Medrano, Dr. Gonzalo Yanarico, Dr. Edwin Condori, Dra. Wendy Cutipa, Dra. Ana María Aracena; gracias por el apoyo, aporte logístico y sobre todo por la amistad brindada.
A los pacientes miembros del grupo ASBOLUP, ya que sin su interés por participar en el presente estudio no se podría beneficiar científicamente nuestra población.
REFERENCIAS
1. Ramos PS, Brown EE, Kimberly RP, Langefeld CD. Genetic Factors Predisposing to Systemic Lupus Erythematosus and Lupus Nephritis. Semin Nephrol. 2011:1-17. [ Links ]
2. Castaño N, Lina M, Diaz G, Pineda R, Rojas A, Anaya J. Meta-analysis of HLA-DRB1 and HLA-DQB1 polymorphisms in Latin American patients with systemic lupus erythematosus. Autoimmunity Reviews. 2008:322-30. [ Links ]
3. Ruiz-Narvaez E, Fraser P, Palmer J, Cupples A, Reich D, Wang Y, et al. MHC region and risk of systemic lupus erythematosus in African-American women. Hum Genet. 2011:1-17. [ Links ]
4. Thibault Flesher D, Sun X, Behrens T, Graham R, Criswell L. Recent advances in the genetics of systemic lupus erythematosus. Expert Rev Clin Immunol. 2010:461-79. [ Links ]
5. Cárdenas-Roldán J, Rojas-Villarraga A, Anaya J-M. How do autoimmune diseases cluster in families? A systematic review and meta-analysis. BMC Medicine. 2013:1-22. [ Links ]
6. Khaled M, Yousr M, Saloua F, Rafika M. The involvement of HLA - DRB1*, DQA1*, DQB1 * and complement C4A Loci in diagnosing systemic lupus erythematosus among Tunisians. Ann Saudi Med. 2004:24(1): 31-5. [ Links ]
7. Morris D, Taylor K, Fernando M, Nititham J, Alarcon-Riquelme M, Barcellos L, et al. Unraveling Multiple MHC Gene Associations with Systemic Lupus Erythematosus: Model Choice Indicates a Role for HLA Alleles and Non-HLA Genes in Europeans. The American Journal of Human Genetics. 2012:91, 778-93. [ Links ]
8. Cruz Quispe A. Determinación del polimorfismo alélico, genotípico, haplotípico de los antígenos HLA clase I y clase II, en la población de donantes y receptores de órganos, atendidos en el laboratorio de Histocompatibilidad e Inmunogenética del Instituto SELADIS. La Paz: UMSA; 2011. [ Links ]
9. Aristizábal B, González Á, Pérez-Cuevas B, Florez V, Martínez I C. Respuesta inmune innata. Autoinmunidad y Enfermedad Autoinmune. Medellin, Colombia: CORPORACIÓN PARA INVESTIGACIONES BIOLÓGICAS; 2005. p. 19-28. [ Links ]
10. Farabosco P, Gorman J, Cleveland C, Kelly J, Fisher S, Ortmann W. Meta-analysis of genome-wide linkage studies of systemic lupus erythematosus. Genes Immun. 2006:7:609-14. [ Links ]
11. Chia Chai H, Elvira Phipps M, Heng Chua K. Genetic Risk Factors of Systemic Lupus Erythematosus in the Malaysian Population: A Minireview. Hindawi Publishing Corporation. 2012:1-10. [ Links ]
12. Severiche D, Correa A. FRECUENCIAS ALÉLICAS DEL HLA-DQ Y SU POSIBLE ASOCIACIÓN CON LES. 2011:1-8. [ Links ]
13. Shimane K, Kochi Y, Suzuki A, Okada Y, Ishii T, Horita T, et al. An association analysis of HLA-DRB1 with systemic lupus erythematosus and rheumatoid arthritis in a Japanese population: effects of *09:01 allele on disease phenotypes. Rheumatology. 2013:52:1172-82. [ Links ]