Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO  uBio
uBio
Share
Cuadernos Hospital de Clínicas
Print version ISSN 1562-6776
Cuad. - Hosp. Clín. vol.54 no.1 La Paz 2009
ARTÍCULOS ORIGINALES
Detección de mutaciones en los Genes rpoB y katG asociados a resistencia en cepas de Mycobacterium tuberculosis de Bolivia
Autor para correspondencia: Aneth Vásquez Michel, anethvasquez@gmail.com, Germán Melean1, Aneth Vasquez Michel1, Elvin Mollinedo Perez1, Ana Rada Tarifa, Pablo Almaraz Ossio2, Mirtha Camacho3 , Jorge Sainz1
1. Instituto de Genética, Facultad de Medicina, Universidad Mayor de San Andrés, Av. Saavedra 2246, La Paz, Bolivia;
2. Laboratorio de Genética Molecular-Unidad de Genética Médica. Instituto de Genética, Facultad de Medicina,
Universidad Mayor de San Andrés, Av. Saavedra 2246, La Paz, Bolivia; 3. Laboratorio Nacional de Referencia de Tuberculosis, Instituto Nacional de Laboratorios de Salud, (INLASA). Rafel Zubieta Nº1889, Miraflores, La Paz, Bolivia
Autor para correspondencia: Aneth Vásquez Michel, anethvasquez@gmail.com
RESUMEN
Objetivo: Evaluar la sensibilidad y especificidad de la genotipificación, como instrumento de diagnóstico rápido y confiable para la detección de mutaciones en los genes rpoB y katG asociados a resistencia,en cepas de Mycobacterium tuberculosis de Bolivia.
Diseño: Test Diagnóstico
Metodología: Las cepas analizadas fueron aisladas y enviadas por los diferentes Laboratorios de la Red Nacional de Diagnóstico de Tuberculosis de Bolivia entre febrero y diciembre de 2007. La muestra para el presente estudio estuvo constituida por un total de 65 aislamientos previamente caracterizados por métodos fenotípicos de cultivo y pruebas de sensibilidad a la RIF e INH, por el método de las proporciones Canetti-Rist. La genotipificación ha sido realizada utilizando el kit Genotype MTBDR®, basado en la utilización de métodos de amplificación e hibridización, para detectar mutaciones a nivel de los marcadores de resistencia rpoB y katG.
Resultados: Se procedió al cálculo de la sensibilidad y especificidad de la prueba de diagnóstico; además de los valores predictivos positivo y negativo. Dicho análisis muestra los siguientes resultados: sensibilidad 74%, especificidad 92%, valor predictivo positivo 92% y valor predictivo negativo 73%.
Conclusiones: La prueba de genotipificación Genotype MTBDR al cumplir con criterios de validación para un estudio de test diagnóstico en nuestro medio, constituye un método rápido, útil y confiable para ser utilizado en el diagnóstico y determinación rutinaria de sensibilidad y resistencia en cepas MTBC.
PALABRAS CLAVE: Mycobacterium tuberculosis, rpoB, katG, resistencia, rifampicina, isoniacida.
Detection of mutations in rpoB and katG genes
associated to resistance in Mycobacterium tuberculosis
strains from Bolivia
ABSTRACT
Objective: To evaluate the sensitivity and specificity of genotyping as a tool for rapid and reliable detection of mutations in rpoB and katG genes associated with resistance in Mycobacterium tuberculosis strains from Bolivia.
Design: Diagnostic Test
Methodology: The strains analyzed were isolated and submitted by different laboratories of the National Network for Diagnosis of Tuberculosis of Bolivia between February and December 2007. The sample for this study consisted of 65 isolates previously characterized by phenotypic methods of culture and sensitivity testing to RIF and INH by the Canetti-Rist proportion method. Genotyping of these samples has been done using the MTBDR Genotype kit, according to amplification and hybridization methods to detect mutations at the rpoB and katG resistance markers.
Results: The sensitivity and specificity of the diagnostic tests were calculated, as well as the positive and negative predictive
values. This analysis shows the following results: sensitivity 74%, specificity 92%, positive predictive value 92%, and negative predictive value 73%.
Conclusions: The genotyping test using Genotype MTBDR, meeting validation criteria for a diagnostic test study in our country, constitutes a quick, useful and reliable tool for use in diagnosis and routine determination of sensitivity and resistance in MTBC strains.
KEYWORDS: Mycobacterium tuberculosis, rpoB, katG, resistance, rifampin, isoniazid
Cómo citar este artículo: Melean G, Vasquez A, Mollinedo E, Rada A, Almaraz P, Camacho M, Sainz J. Detección de mutaciones en los Genes rpoB y katG asociados a resistencia en cepas de Mycobacterium tuberculosis de Bolivia. Cuad Hosp Clín 2009;54:20-26.
INTRODUCCIÓN
La Tuberculosis (TBC) es una enfermedad infectocontagiosa causada por diversas especies del género Mycobacterium, todas ellas pertenecientes al Complejo Mycobacterium tuberculosis. La especie más importante y representativa es Mycobacterium tuberculosis. La TBC es posiblemente la enfermedad infecciosa más prevalente en el mundo, afecta a un tercio de la población mundial, alrededor de 8 millones de personas se infectan anualmente y 1,8 millones fallecen.1
Según datos de la gestión 2004, la notificación de la incidencia de tuberculosis clasifica a Bolivia entre los países con alta carga de tuberculosis con una incidencia de 100 o más por 100.000 habitantes, compartiendo esta situación con países del continente africano. En la región de las Américas, Bolivia continua siendo un país de atención prioritaria para la OPS, por la carga de tuberculosis que presenta, constituyéndose en el grupo que aporta un 80% a la carga notificada, compartiendo esta situación con Haití, República Dominicana, Perú, Brasil entre otros.2
La situación se agrava por el incremento de cepas de M. tuberculosis multidrogo resistentes (MDR), que se las define operativamente como resistentes por lo menos a la rifampicina (RIF) y a la isoniacida (IHN), fármacos que constituyen el eje de la terapia antituberculosa de primera linea.3
La resistencia a la RIF se debe aproximadamente en un 95% de las cepas, a mutaciones en un fragmento relativamente pequeño (81pb) del gen rpo que codifica para la subunidad B de la RNA-polimerasa bacteriana,4-6 por el contrario las mutaciones que causan resistencia a INH están localizadas en varios genes y regiones. Se han identificado entre 50-95% de cepas que contienen mutaciones en el codón 315 del gen katG,5-7 entre 20-35% mutantes en la región reguladora del gen inhA7,8 y un restante 10-15% en otros genes.5,8
La identificación rápida y confiable de la resistencia a antimicrobianos en M. tuberculosis es un importante reto para asegurar una terapia pronta y adecuada, y así limitar el impacto negativo en Salud Publica por la diseminación de cepas multiresistentes. Para ello se necesitan métodos basados en técnicas moleculares para evaluar la presencia de mutaciones genómicas que confieren resistencia. Puesto que, la detección de resistencia por métodos convencionales (fenotípicos) resulta poco práctica debido al largo tiempo que implica el crecimiento in vitro de M. tuberculosis,9 además, la detección directa de mutaciones conocidas es más confiable en cuanto a la predicción de la respuesta terapéutica.
Con el fin de permitir la detección de las mutaciones más frecuentes que determinan resistencia a la RIF y también a la mayor proporción de mutaciones que causan resistencia a la INH, en aislamientos de M. tuberculosis, ha sido desarrollado un ensayo de tiras comerciales con sondas de ADN (Genotype MTBDR®; Hain Lifescience, Nehren, Germany). Basado en un multiplex PCR en combinación con hibridación reversa de los amplicones rpoB y katG, para identificar ya sea de secuencias de tipo salvaje (sensibles) o mutaciones específicas en estos genes (resistentes).10
El objetivo de este estudio, fue evaluar la sensibilidad y especificidad de la Genotipificación, como instrumento de diagnóstico rápido y confiable para la detección de mutaciones en los genes rpoB y katG asociados a resistencia a la rifampicina y a la isoniacida, en cepas de M. tuberculosis nativas de Bolivia.
MATERIALES Y MÉTODOS
Cepas Analizadas
Las cepas analizadas fueron aisladas entre febrero a diciembre de 2007 en diferentes Departamentos de Bolivia y enviadas por los Laboratorios de la Red Nacional de Diagnóstico de Tuberculosis al Laboratorio Nacional de Referencia en Diagnóstico de la Tuberculosis del Instituto Nacional de Laboratorios de Salud (INLASA), la muestra estuvo constituida por 65 aislamientos de M. tuberculosis previamente caracterizadas por métodos fenotípicos de cultivo y pruebas de sensibilidad a RIF e INH, por el método de proporciones estándar en medio Lowenstein-Jensen (método de Canetti-Rist).
El panel incluyó 15 cepas MDR, 24 cepas monoresistentes y 2 cepas poliresistentes no MDR). También se incluyeron 24 cepas sensibles (Tabla 1).
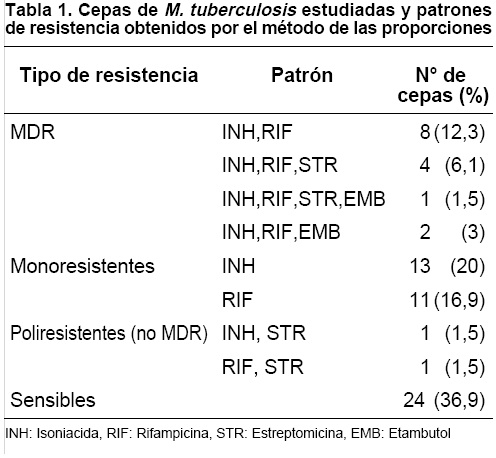
Esta caracterización fenotípica se constituye en el patrón de oro (gold standard).
Caracterización Genotípica
Ensayo Genotype MTBDR. Este ensayo está basado en la hibridización reversa entre amplicones derivados de un multiplex PCR y sondas unidas a nitrocelulosa que cubren secuencias superpuestas wild type (WT), de las regiones rpoB WT1-W5 (Figura 1), que son las mutaciones más frecuentes para rpo y también WT T1-T2, mutaciones a nivel 315 en el gen katG. La presencia de mutación es indicada por la falta de hibridización en las sondas MUT, que se evidencia por ausencia de marca. También está incluido un control universal (UC) que verifica la presencia de amplicones de bacterias gram-positivas con alto contenido genómico de G+C y un control específico para aislamientos del complejo M. tuberculosis (TUB).
Extracción de ADN
Fueron resuspendidas de 3 a 5 colonias de medio sólido en 300 ul de agua destilada estéril, y lisadas por incubación a 95ºc (baño de agua) por 20 minutos y posterior sonicación por 15 min, luego fueron centrifugados los tubos a 13.000 rpm por 5 minutos y 5μl del sobrenadante fueron usados para la amplificación por PCR.
Amplificación Las regiones genómicas implicadas en la resistencia fueron amplificadas de acuerdo a recomendaciones del fabricante.

La mezcla para amplificación estuvo constituida por 35 μl de mezcla del primer-nucleótido (PNM), provisto en el kit, 5 μl de tampón con 2mM de MgCl2, 2U de Taq DNA polimerasa termoestable y 5 μl de solución de DNA cromosómico extraído, todo en un volumen final de 50 μl. La amplificación fue realizada en un termociclador Amplitron I, con los siguientes parámetros de temperatura: 5 min de desnaturalización a 95ºc, seguido por 10 ciclos de 30s a 95ºC y 2 min a 58ºC, seguido por 20 ciclos adicionales de 25s a 95ºC, 40s a 53ºC y 40s a 70ºC, finalizando con un paso de extensión final de 8 min a 70ºC. La presencia de los amplicones fue verificada por electroforesis en gel de agarosa al 1,5 %.
Hibridización
Luego de la amplificación, se realizó un paso de desnaturalización, posteriormente los amplicones de hebra simple marcados con biotina fueron hibridados a membranas unidas a sondas por 30min a 45°C en agitador semiautomático. Las tiras fueron posteriormente lavadas por 15 min a 45°C de acuerdo a instrucciones del fabricante, las señales de hibridización se detectaron por adición de un conjugado de estreptavidina-fosfatasa alcalina, seguida de una reacción de substrato de fosfatasa alcalina.
RESULTADOS
La presencia de secuencias de ADN salvaje (wild type) sensibles o mutadas (resistentes) en la región de 81-pb de rpoB (Figura 1) y en el codon 315 de katG, es evidenciada por señales claras de hibridización en las tiras, que pueden ser analizadas usando los templetes adjuntos al kit. Además la PCR múltiple y las tiras incluyen un control de amplificación específico para aislamientos del complejo M. tuberculosis y un control de amplificación de otra especie bacteriana.
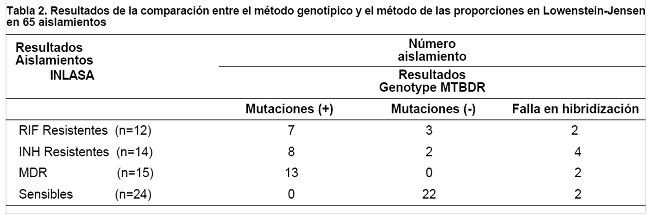
A pesar de que las 41 cepas resistentes amplificaron con éxito, ocho aislamientos, no se tomaron en cuenta, debido a la falta de señales de hibridización positivas con las sondas específicas de control, por lo tanto se tomaron en cuenta para el análisis estadístico solamente 33 cepas resistentes, situación similar ocurrió con las cepas sensibles en las que se descartaron 2 por la misma razón. Los resultados son mostrados en la Tabla 2.
Se obtuvieron los siguientes valores de concordancia entre ambos métodos: resistencia a Rifampicina 64%, a Isoniacida 60% y MDR 87%, cepas sensibles 100%.
En cuanto a la resistencia a la RIF, tanto en las cepas monoresistentes a RIF como MDR, se obtuvieron mutaciones en 19 de las 33 (58 %) resistentes con 3 patrones de hibridización para el gen rpoB, el codón más afectado fue el S531L (53%), seguido de los codones H526Y (23%), D516V (18%) y H526D (6%), en dos cepas mutantes no se pudo determinar el codón. Ninguna de las cepas reportadas como sensibles presentó mutaciones a este nivel.
Del mismo modo tomando en cuenta tanto cepas monoresistentes a INH y MDR, se detectaron mutaciones en el codón 315, en 22 cepas (67%), pero en ninguna de las cepas sensibles se detectaron mutaciones. En la mayoría de las cepas se pudo verificar distintas mutaciones: 13 (S315T1), 8 (S315T2) y 1 inespecífica que solo presentó la ausencia de la señal de hibridización wild type, por otro lado, en discrepancia con el fenotipo, 5 cepas caracterizadas previamente como resistentes, presentaron un patrón de hibridización sensible (wild type).
Análisis de test diagnóstico
Se procedió al cálculo de la sensibilidad y especificidad en base al paquete informático Epi-Info versión 6.0 tomando en cuenta los siguientes datos:
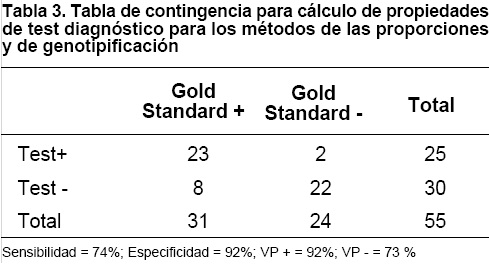
Intervalo de confianza estimado 95%, por tanto se acepta un error tipo 1 del 5%; Potencia estimada de la prueba del 80%; además de los valores predictivos positivos y negativos. Se tomó como patrón de oro (gold standard) la caracterización fenotípica.
Dicho análisis mostró los siguientes resultados. 23 muestras fueron positivas por ambas pruebas, 2 fueron falsos positivos, 8 falsos negativos, 22 muestras fueron negativas, estas últimas corresponden a las cepas sensibles. Por lo tanto la sensibilidad del método es de 74%, la especificidad 92%, valor predictivo positivo 92% y valor predictivo negativo 73%. Lo que demuestra que el método de genotipificación es bastante sensible y altamente específico (Tabla 3).
DISCUSIÓN
Si buscamos controlar la diseminación de las cepas de M. tuberculosis multidrogo resistentes (MDR) y además mejorar la terapia antituberculosa en pacientes infectados por estas cepas, está por demás claro que se debe reducir el tiempo que toma la identificación de estas cepas, ya que el cultivo, la identificación y las pruebas de sensibilidad tradicionales demoran mucho. La mejor manera de lograrlo es introduciendo métodos moleculares modernos que puedan ser aplicados en cepas o directamente en muestras clínicas.
Los resultados del presente estudio indican que los métodos de genotipificación utilizando el kit Genotype MTBDR tienen en general un buen rendimiento (amplificación exitosa en todas las cepas), así como valores de sensibilidad y especificidad bastante elevados tanto para el diagnóstico como para la determinación rápida de resistencia y sensibilidad a fármacos antituberculosos.
La sensibilidad del test hallada en el presente estudio es del 74%, más baja en comparación a otros estudios con el mismo método, sin embargo esto se debe probablemente a la diferencia en el tamaño muestral entre este y los otros estudios en los que supera el 90%, en lo referente a la especificidad (92%), el valor en otros estudios es similar.10-13
También es importante, conocer el tipo y la frecuencia de las regiones mutadas, debido a que muchas publicaciones subrayan la relevancia de este tipo de análisis molecular, ya que mutaciones específicas en rpoB podrían estar relacionadas con resistencia de alto o bajo nivel a la RIF. En el presente estudio las mutaciones más frecuentes se encontraron en las osiciones S531L (53%) y H526Y (23%), resultados que coinciden con otros estudios,4,14,15 que además se relacionan con la expresión clínica de niveles altos de resistencia a la RIF, lo cual tiene connotaciones epidemiológicas que nos estarían indicando que, la mayor parte de cepas circulantes en nuestro país al igual que en otros países tienen alto nivel de resistencia a la RIF, información que desde luego no es detectable utilizando pruebas de sensibilidad convencionales en medio Lowenstein-Jensen con una concentración fija de RIF de 40ug/ml.
Por otro lado, como se puede evidenciar en los resultados de concordancia enfocados a la detección de resistencia a la isoniacida, y tomando en cuenta que el kit, determina solamente la resistencia mediada por el gen katG se conoce que se da en un 50-95% involucrados, 20-35% que contienen mutaciones en inhA,8 y un 10-15% tienen mutaciones en la región intergénica del gen ahpC-oxyR y otros genes menos frecuentes que están en estudio como ndh, kasA.18 Este hecho posiblemente explicaría que, 5 cepas monoresistentes a la Isoniacida por el método convencional resultaron ser sensibles por nuestro método, lo que nos llevaría a inferir que efectivamente estas cepas son resistentes, pero esta resistencia no estaría determinada por mutaciones en katG, sino probablemente por otros genes.
En relación a la distribución de mutaciones asociadas con la resistencia a INH y a RIF se identificó, un aparente desequilibrio en cuanto a concordancia de resultados, entre las cepas monoresistentes y aquellas MDR, el test que utilizamos parece ser mucho más eficaz detectando cepas MDR que detectando cepas monoresistentes, como podemos comprobar en la Tabla 2, el porcentaje de concordancia de detección de este tipo de cepas es mucho más alto (87%) en MDR que en cepas monoresistentes que oscila entre 60-64%. No obstante, nuevamente es importante mencionar que, el número de muestras utilizado en el presente estudio es muy pequeño para un análisis estadístico definitivo en este sentido, sin embargo esta tendencia, es ratificada en otros estudios, que sugieren que la frecuencia en encontrar cepas monoresistentes por este test es un tanto más baja.10,17
En cuanto a las 8 cepas que fueron excluidas del análisis estadístico, por no presentar señales de hibridización que denotaran mutaciones, pero fueron resistentes al menos a un antituberculoso por el método de referencia fenotípico, la explicación hipotética más cercana a la que podemos referirnos es el fenómeno de la "heteroresistencia" que representa una mezcla de subpoblaciones bacterianas wild type (sensibles) y resistentes en el cultivo inicial, constituyéndose este fenómeno en un importante obstáculo tanto para la terapia como para los métodos moleculares de detección de resistencia, ya que el límite de detección de la prueba es de 10% de ADN mutante en el total de la población bacteriana y sí la proporción de bacterias resistentes de un aislamiento es menor a este porcentaje, sería muy difícilmente detectado por este método molecular.4
En el caso de las cepas que presentan solo ausencia de señal de hibridización en la sonda wild type indicativo de sensibilidad, que sin embargo, no se encuentra la mutación específica, como se evidencia en 3 casos en este estudio (2 en RIF y 1 en INH), según bibliografía,10,13 se las considera resistentes debido a que probablemente la región del codón afectada por la mutación se encuentra fuera de las sondas específicas del kit, por lo tanto no podemos concluir que esta cepa es sensible, sino más bien por argumentos moleculares se las considera resistentes.
En aquellas cepas fenotípicamente resistentes cuyo resultado fue sensible por genotipificación se podrían plantear otras alternativas como ser: heteroresistencia, mecanismos de resistencia adicionales y mutaciones poco frecuentes que se encuentren fuera de las regiones investigadas.
En general, en comparación con los métodos convencionales fenotípicos de cultivo que actualmente se realizan, la interpretación y el análisis de los resultados de la prueba de Biología Molecular nos muestra una serie de ventajas, entre las que podemos mencionar:
- Acortamiento del tiempo, ya que utilizando los métodos convencionales por norma de la OPSOMS9 no se debe estudiar la sensibilidad de una cepa que tenga menos de 30 días de cultivo, debido a que las bacterias resistentes a la isoniacida son generalmente más lentos en multiplicarse y se corre el riesgo de que las colonias de estos aún no se encuentren formadas. El no incluir estas colonias en el estudio llevaría a clasificar como sensible una cepa que no lo es.
- El hecho de efectuar pruebas de sensibilidad en cultivos almacenados más de dos meses por métodos convencionales, que podría condicionar falsos negativos no se da con los métodos de genotipificación, debido a que existen estudios que demuestran que en el rendimiento de las pruebas de genotipificación no influye el tiempo de almacenamiento de las cepas, pudiendo utilizar aún cepas congeladas más de 6 años, lo que permitiría realizar incluso estudios retrospectivos.13
- El corto tiempo que toma la realización de este método, permite la optimización de la terapia oportuna, ya que se dispone del diagnóstico de sensibilidad mucho antes de que los resultados confirmatorios del cultivo estén disponibles.
- El ensayo genético no requiere de organismos viables y por lo tanto se reduce el riesgo biológico en el laboratorio.
- El ensayo permite una detección rápida y específica de las mutaciones más frecuentes que condicionan la resistencia a la INH y a la RIF, del mismo modo permite también una fácil interpretación de los resultados, sin requerir de la experticia y alta tecnología que se requiere para la interpretación de datos de secuenciación y PCR en tiempo real (otras técnicas moleculares que se están aplicando en otros países para estudios de diagnóstico y resistencia en M. tuberculosis)
Finalmente, coincidimos con las conclusiones de otros autores que sugieren que la principal limitación de este método es la baja sensibilidad de detección de resistencia a la isoniacida,11,19 debido a que como se ha señalado varias veces en el presente manuscrito, el test está dirigido a la detección de resistencia a la INH, solo en mutaciones en el gen katG S315T.
En conclusión, la prueba de genotipificación Genotype MTBDR al cumplir con criterios de validación para un estudio de test diagnóstico en nuestro medio (Tabla 3), se constituye como un método rápido, útil y confiable para ser utilizado en el diagnóstico y determinación rutinaria de sensibilidad y resistencia en cepas MTBC, de la misma manera puede ser aplicado en circunstancias donde se requieran resultados de sensibilidad rápidos, ya sea para utilizarse en casos de pacientes altamente infectados en los que se sospecha sean portadores de cepas MDR, pacientes en re-tratamiento y para casos de contacto con posibles cepas resistentes.
AGRADECIMIENTOS
Agradecemos al Dr. Milton Magne por las pruebas fenotípicas de las cepas de M. tuberculosis mediante el método Canetti-Rist con sus respectivos resultados, realizadas en el Laboratorio Nacional de Referencia de Tuberculosis del INLASA. Un agradecimiento especial a la Dra. Pilar Navia por el apoyo epidemiológico y estadístico al presente trabajo.
REFERENCIAS
1. Thomas MD. Tuberculosis. En: Fauci A, Braunwald E, Kasper D, Hauser E,Longo D, Jameson L, Loscano J. Harrison: Principios de Medicina Interna. Mexico D.F: Editorial McGraw-Hill; 1989. p. 769-779. [ Links ]
2. Programa Nacional de Tuberculosis (PNT). Tuberculosis, La Paz, Bolivia; 2008 [ Links ]
3. Espinal MA. The global situation of MDR-TB. Tuberculosis 2003, (Edinburgh) 83:44-51. [ Links ]
4. Heep M, Brandstatter U, Rieger N, Lehn E, Richter S, Rusch-Gerdes. Frequency of rpo mutations inside and outside the cluster I region in rifampin - resistant clinical Mycobacterium tuberculosis isolates. J. Clin. Microbiol. 2001; 39: 107-110. [ Links ]
5. Ramaswamy S, and Musser J. Molecular Genetic basis of antimicrobial agent resistance in Mycobacterium tuberculosis isolated in Italy. J.Clin.Microbiol. 1998; 37: 1197-1199. [ Links ]
6. Talenti A, Honore N, Bernasconi C, March J, Ortega A, Heym B, Takiff HE, Cole ST. Genotyping assessment of isoniacid and rifampin resistance in Mycobacterium tuberculosis: a blind study at reference laboratory level. J Clin Microbiol 1997;35:719-723. [ Links ]
7. Slayden R and Barry C. The genetics and biochemestry of isoniazid resistance in Mycobacterium tuberculosis. Microbes infect 2000;2:659-669. [ Links ]
8. Musser J, Kapur D, Williams B, Kreiswirth D, Van Soolingen D, van Embden J. Characterization of the catalaseperoxidase gene (katG) and inhA locus in isoniazid-resistant and susceptible strains of Mycobacterium tuberculosis by automated DNA sequencing: restricted array of mutations associated with drug resistance. J Infect Dis 1996;173:196-202. [ Links ]
9. World Health Organization. Global tuberculosis control: surveillance, planning, financing. WHO Report 2007. WHO/HTM/TB/2007,411.Geneva, Switzerland:WHO, 2007. [ Links ]
10. Hillemann D, Weizenegger M, Kubica T, Richter E, Niemann S. Use of genotype MTBDR assay for rapid detection of rifampin and isoniazid resistance in Mycobacterium tuberculosis complex isolates. J Clin Microbiol 2005; 43(8): 3699-3703. [ Links ]
11. Miotto P, Piana F, Penati V, Canducci F, Battista G, Cirillo M. Use of genotype MTBDR assay for molecular detection of rifampin and isoniazid resistance in Mycobacterium tuberculosis clinical strains isolated in Italy. J Clin Mocrobiol 2006;44(7):2485-2491. [ Links ]
12. Cengiz C, Turhan A, Akinci P and Soyler I. Evaluation of genotype MTBDR assay for rapid detection of rifampin and isoniazid resistance in Mycobacterium tuberculosis isolates. J Clin Microbiol 2006;44;(7):2338-2342. [ Links ]
13. Somoskovi A, Dormandy J , Mitsani D, Rivenburg J, Salfinger M. Use of smear âpositive samples to assess the PCRbased genotype MTBDR assay for rapid, direct detection of the Mycobacterium tuberculosis complex as well as its resistance to isoniazid and rifampin. J Clin Microbiol 2006;44(12):4459-4463. [ Links ]
14. de Oliveira M, da Silva R, Cardoso O, Gomes H, Fonseca L, Werneck A, et al. Rapid detection of resistance against rifampin in isolates of Mycobacterium tuberculosis from Brazilian patients using a reverse-âphase hibridazion assay. J Microbiol Methods 2003;53:335-342. [ Links ]
15. Viader J, Luna J, Reyes R, Valdez L, del Bosque M, Guerrero O. Frequency of mutations in rpo and codon 315 and 463 of katG in rifampin-and/or isoniazid -resistant Mycobacterium tuberculosis isolates from northeast Mexico. Microb Drug Resist 2003;9:33-38. [ Links ]
16. Mokrousov L, Narvskaya T, Otten E, Limeschenko L and Vyshnesky B. High prevalence of katG Ser315Trh substitution among isoniazid resistant Mycobacterium tuberculosis clinical isolates from Northwestern Russia, 1996-2001. Antimicrob Agents Chemother 2002;46:1417- 1424. [ Links ]
17. Hazbon M, Brimacombe M, Bobadilla M, Cavatore M, Gerrero M, Varma H et al. Population genetics study of isoniazid resistance mutations and evolution of multi-drugresistant Mycobacterium tuberculosis. Antimicrob. Agents Chemother 2006;50:2640-2649. [ Links ]
18. Sreevatsan S, Pan X, Zhang Y, Deretic V, Musser M. Analysis of the oxyR-ahpC region in isoniazid-resistant and susceptible Mycobacterium tuberculosis complex organism recovered from diseased humans and animals in diverse localities. Antimicrob Agents Chemother 1997;41:600. [ Links ]
19. Mäkinen J, Marttila M, Marjamaki M, Vijanen K, Soini H. Comparison of two commercially available DNA line probe assays for detection of multidrug-resistant Mycobacterium tuberculosis. J Clin Microbiol 2006;44:350-352