Servicios Personalizados
Revista
Articulo
Indicadores
Links relacionados
Compartir
Cuadernos Hospital de Clínicas
versión impresa ISSN 1562-6776
Cuad. - Hosp. Clín. vol.53 no.1 La Paz 2008
ARTICULO ORIGINAL
UMSAgen, método para la extracción simultánea de RNA y DNA para diagnóstico molecular
Ricardo Amaru*, Rosario Peñaloza*, Hortencia Miguez*, Gina Torres*, Heriberto Cuevas*.
RESUMEN
INTRODUCCIÓN
El RNA y el DNA son materiales genéticos de la célula cuya extracción es útil para la medicina moderna. Actualmente existen varios métodos de extracción de RNA y DNA por separado y a costos altos. En este trabajo se describe el método UMSAgen como alternativa para la extracción simultanea de RNA total y DNA genómico para diagnósticos moleculares.
MATERIAL Y MÉTODOS
Se obtuvieron veinte muestras de médula ósea de pacientes con leucemia mieloide crónica. Los leucocitos fueron separados de los glóbulos rojos con tampón de lisis. Aproximadamente 3 x 106 de células fueron lisadas en guanidio y conservadas a -20 ºC hasta el momento de la extracción del RNA y DNA. Se realizaron extracciones con 3 diferentes métodos: a) El método comercial Quiagen para el RNA total, b) el método clásico fenol-cloroformo para el DNA genómico y c) el método UMSAgen para extracción simultanea de RNA y DNA.
RESULTADOS
La pureza del RNA total y DNA genómico extraídos por el método UMSAgen es similar a la extraída con el Kit Quiagen para el RNA y la técnica clásica para el DNA. La evaluación realizada por electroforesis en agarosa y la amplificación del daño molecular BCR-ABL y JAK-2 V617F, por RT-PCR y PCR respectivamente, fue exitosa.
CONCLUSIÓN
El método UMSAgen es una alternativa fácil y económica para la extracción de RNA total y DNA genómico en diagnóstico e investigación molecular.
PALABRAS CLAVES:
Rev. Cuadernos 2008, Vol. 53 No.1(Pags. 38 - 43). RNA, DNA, extracción, PCR, RT-PCR y electroforesis en agarosa.
ABSTRACT
INTRODUCTION
The RNA and the DNA are genetic materials of the cell. The RNA/DNA extraction is necessary in modern medicine. Several RNA/DNA isolation methods exist but they are very expensive and therefore not applicable in our country. In this work, we describe the UMSAgen method as an alternative for simultaneous total RNA and genomic DNA extraction for molecular diagnosis.
MATERIAL AND METHODS
Twenty bone marrow samples of chronic myeloid leukemia patients were obtained. The leukocytes were separated from the erythrocytes with lyses solution; 3x106 cells were lysed in guanidio solution and conserved at -20ºC until the extraction procedure. The extraction of nucleotide acids was realized by 3 different methods: a) commercial method QuiagenTM for RNA extraction, b) classic method (phenol/chloroform) for DNA extraction and c) UMSAgen method for simultaneous RNA/DNA extraction. The two first methods were used as a control test for UMSAgen.
RESULTS
The quality and quantity of total RNA and genomic DNA obtained by the UMSAgen method were similar to that of the QuiagenTM Kit and the classic method, respectively. The ABL-BCR and JAK-2V617F amplification by RT-PCR and PCR were successful.
CONCLUSIONS
The UMSAgen is an easy and inexpensive altrernative method for total RNA and genomic DNA extraction in diagnostic and biomolecular research.
KEY WORDS :
Rev. Cuadernos 2008, Vol. 53 No.1(Pags. 38 - 43). RNA, DNA, extraction, PCR, RT- PCR and electrophoresis.
INTRODUCCIÓN
La extracción y manipulación del material genético de las células eucarióticas representado por el RNA y el DNA es rutina de la medicina moderna. El RNA es producto de la transcripción de genes1, descubierto por Alexander Rich y David Davies en 19532, su rol en la síntesis de proteínas fue descrito en 19393 y posteriormente el mecanismo de su síntesis fue descrito por Severo Ochoa4. El RNA total esta constituido por el 80% de RNA ribosomico (28S, 18S, 5 S y 5.8S), 5% de RNAmensajero y 15% de RNA de transferencia5, 6. El RNA es muy lábil, se degrada fácilmente por las RNasas presentes en la piel y por temperaturas superiores a 4ºC, lo que constituye el problema principal de la extracción del RNA, por ello se utiliza inhibidores de la RNasa, guantes para su manipulación y temperaturas inferiores a 4ºC 1, 5. La extracción del RNA total o RNA mensajero de células y tejidos es fundamental en la síntesis de cDNA para la amplificación de RNA anormales, como en la leucemia mieloide crónica (LMC), cuyo diagnostico requiere la demostración de la traslocación 9;22 (BCR-ABL) por el método RT-PCR 7,8,9. El RNA total puede ser extraído por electroforesis en gel, ultracentrifugación por gradiente de densidad o cromatografía de intercambio iónico. Para la extracción del RNA las células se lisan en triton X-100 con el inhibidor complejo vanadil ribonucleósido, que deja intacto el núcleo y permite la extracción del RNA citoplasmático, o en guanidio isotiocianato que además tiene actividad inhibidora de RNAsa1,5. La doble hélice de DNA fue descrito por primera vez por Watson JD y Crick FHC10, posteriormente se describieron otros tipos de DNA11,12. El DNA para su extracción requiere ser separado de los componentes de membrana, proteínas residuales y polisacáridos. Para evitar la degradación del DNA es necesario utilizar material libre de DNAsas y manipulación cuidadosa1,5. El método clásico para la proteólisis utiliza proteinasa K y el fenol cloroformo para separación del DNA de las proteínas, lípidos e hidratos de carbono5. Las enfermedades con daños moleculares requieren DNA de buena calidad para la amplificación del DNA por PCR, como la trombocitosis esencial que presenta la mutación de JAK-2 V617F13,14. Los métodos clásicos y los kits comerciales para la extracción del RNA y DNA son costosos y requieren de equipos sofisticados. Por ello, en el presente trabajo presentamos un método estandarizado en nuestro laboratorio, que se caracteriza por ser fácil y económico, que permite obtener simultáneamente RNA y DNA de buena calidad, útil para el diagnóstico molecular. Este método ha sido denominado UMSAgen.
MATERIAL Y MÉTODOS MUESTRAS
Se obtuvieron veinte muestras de médula ósea de pacientes con leucemia mieloide crónica en tubos Venoject con EDTA y procesadas inmediatamente para la separación de leucocitos de los glóbulos rojos con tampón de lisis. Aproximadamente 3 millones de células fueron lisadas en 300 µl de guanidio y conservadas a -20 ºC hasta el momento de la extracción del RNA y DNA.
EXTRACCIÓN DE RNA TOTAL Y DNA GENÓMICO POR EL MÉTODO UMSAGEN. Fundamento
El método que proponemos se basa en la separación de proteínas, RNA y DNA aprovechando las diferentes características de solubilidad en fase orgánica y acuosa.
Reactivos
• Agua DEPC 0.1%
Dietil Pirocarbonato 1 ml Agua c.s.p 1000 ml
• Tiocianato de guanidina 4M
Guanidina Tiocianto 23.64 g N-Lauril Sarcosil 250 mg Citrato de Sodio. 2 H2O 367 mg Beta-Mercapto Etanol 14,3 M 350 µl Agua DEPC c.s.p. 50 ml
• Cloroformo- alcohol Isoamílico
Cloroformo 49 ml Alcohol Isoamilico 1 ml
• Acetato de sodio 3M
Acetato de sodio 13,608 g Agua c.s.p. 50 ml
• Solución de lisis para glóbulos rojos 10X
Cloruro de amonio Bicarbonato de potasio Etilen diamino tetra acético sal disódica
• Fenol ácido
Procedimiento
-
Trabajar a 4ºC.
-
Descongelar la muestra y agitar en vortex y
agregar: 30 µl Acetato de Sodio 330 µl Fenol Acido 100 µl Cloroformo alcohol Isoamilico 49:1
• Mezclar enérgicamente en vortex y dejar 10 min a -20ºC.
-
Centrifugar a 13000 rpm 15 min a 4ºC.
-
Recuperar cuidadosamente la fase acuosa (sobrenadante) en otro tubo eppendorf.
-
Agregar 500 µl de Isopropanol y mezclar
suavemente por inversión.
-
Extracción de DNA
-
Sacar el DNA precipitado con la punta de una micropipeta en un tubo eppendorf que contenga 500 µl de etanol frío al 70%.
-
Centrifugar a 13000 rpm 5 min, desechar el sobrenadante y secar el sedimento a temperatura ambiente.
-
Agregar 200 µl de agua destilada estéril y dejar a temperatura ambiente hasta que se disuelva el DNA y conservar a 4ºC.
-
Extracción de RNA
-
Dejar a - 20 ºC por 60 min el tubo que contienen RNA.
-
Centrifugar a 13000 rpm 15 min a 4ºC y decantar el sobrenadante.
-
Agregar 500 µl de Etanol frío al 70 %, mezclando suavemente por inversión.
-
Centrifugar a 13000 rpm 5 min a 4 oC y decantar totalmente el sobrenadante.
-
Resuspender el sedimento con
-
50 µl de agua DEPC fría
-
5 µl de Acetato de Sodio
• 100 µl de Etanol absoluto frío Mezclar bien por inversión y dejar a - 20 ºC por una noche.
-
Centrifugar a 13000 rpm 15 min a 4 oC y decantar totalmente el sobrenadante.
-
Secar el sedimento a temperatura ambiente hasta eliminación total del sobrenadante.
-
Resuspender el sedimento con 30 µl de agua DEPC y conservar el RNA extraído a -20ºC.
EXTRACCIÓN DEL RNA TOTAL POR EL MÉTODO KIT QIAGEN.
Fundamento
El método QIAGEN representa una nueva tecnología para extraer RNA total, que combina las propiedades selectivas de unión de una membrana de Silicagel colocada en una columna, con la velocidad y la tecnología microspin.
Reactivos
-
Tampón de lisis de eritrocitos ( Tampón El)
-
Tampón de lisis (Tampón RLT)
-
Tampón RPE
-
Tampón de lavado (Tampón RW1)
-
Agua libre de RNasa
Procedimiento
La extracción del RNA total se realizó de acuerdo a las instrucciones del fabricante.
EXTRACCIÓN DEL DNA GENÓMICO CON EL MÉTODO CLÁSICO. Fundamento
Se basa en la digestión de proteínas mediante la proteínasa K y la diferencia de polaridad de los ácidos nucleicos, proteínas, lipoproteínas y polisacáridos; además de la solubilidad de los ácidos nucleicos en solución acuosa.
Reactivos
-
SDS 10%
-
TNE 1X
-
Proteinasa k
-
Fenol – cloroformo/alcohol isoamílico (1:1)
-
Cloroformo/alcohol isoamílico (24:1)
-
Acetato de sodio 3M
-
Etanol Absoluto.
Procedimiento
La extracción del DNA genómico se realizó de acuerdo al método descrito por Maniatis T (5).
Análisis cuantitativo.
La concentración de RNA y DNA se determinó por espectrofotometría (Biophotomether -Eppendorf, Hamburgo - Germany) realizando lecturas a 260 nm.
Análisis cualitativo.
La presencia del RNA ribosómico y DNA genómico se verificó mediante la separación electroforética a 100 V por 45 minutos en gel de agarosa al 1 % con bromuro de etidio y observados en el transiluminador con luz UV. La pureza del RNA y DNA se evaluó mediante el cociente entre absorbancia a 260 y 280 nm obtenidas por espectrofotometría (Biophotomether - Eppendorf, Hamburgo - Germany).
Amplificación de RNA (RT-PCR)
La amplificación del RNA mensajero se realizó mediante la técnica del RT-PCR (reacción en cadena de la polimerasa) utilizando primers oligonucleótidos para la síntesis de cDNA y primers específicos para daño molecular BCR-ABL p210 (6). Los productos amplificados fueron separados por electroforesis en gel de agarosa al 2% con bromuro de etidio y observados en el transiluminador con luz UV.
Amplificación de DNA (DNA-PCR)
La amplificación del DNA se realizó mediante la técnica del PCR utilizando primers para la detección del exon 12 del gen JAK-2 (30,31). Los productos amplificados fueron separados por electroforesis en gel de agarosa al 2% con bromuro de etidio y observados en el transiluminador con luz UV.
RESULTADOS Se analizaron 20 muestras de médula ósea de pacientes con leucemia mieloide crónica. A partir de cada muestra se realizaron tres extracciones: Primero con el método UMSAgen para extraer simultaneamente RNA total y DNA genómico, segundo con el método comercial Kit Quiagen para extraer RNA total y tercero con el método clásico para la extracción de DNA genómico. Los métodos de extracción de RNA total Quiagen y del DNA por el método clásico se consideraron como metodos “Gold Standard” para el presente estudio. La concentración de RNA total encontrada por el método UMSAgen fue de 0.11 µg/µl +/- 0.14 y la pureza
1.8 +/- 0.23; mientras que, con el método Quiagen la concentración fue de 0.075 µg/µl+/- 0.013 y la pureza fue de 1.9 +/-0.1 (Cuadro 1)
Cuadro 1. Extracción de RNA total por el método UMSAgen y KIT QIAGEN
| UMSAgen | KIT QIAGEN | |
|---|---|---|
| No | 20 | 20 |
| µg/µl | 0.11 + 1.14 | 0.08 + 0.01 |
| 260/280 | 1.8 + 0.23 | 1.9 + 0.16 |
La separación electroforética del RNA total se observa en la figura 1.
Fig. 1

Fotografía de corrida electroforética de RNA total. Se observan 3 bandas de ácidos nucleicos, la primera corresponde al DNA remanente, la segunda al RNA ribosómico 28S y la tercera al RNA ribosómico 18S.
Se realizó la amplificación del gen quimérico BCRABL por RT-PCR, a partir de los RNA extraídos por el método UMSAgen, el daño molecular esta presente en todas las muestras analizadas. (Figura 2).
Fig. 2
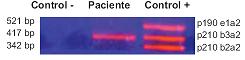
Fotografía de corrida electroforética de la amplifi cación del BCR-ABL. La primera columna corresponde a control negativo; la segunda columna corresponde al paciente con Leucemia mieloide crónica BCR-ABL p210b3a2 positivo; la tercera columna es el control positivo representado por las diferentes combinaciones del gen quimérico BCR-ABL p210 realizado por un mix de RNA total de 3 pacientes. Los RNA fueron extraídos con el método UMSAgen
La concentración y la pureza de DNA genómico obtenida por el método UMSAgen y el método clásico se observan en el cuadro 2.
Cuadro 2. Extracción de DNA genómico por el método UMSAgen y Clásico
| UMSAgen | CLASICO | |
|---|---|---|
| No | 20 | 20 |
| µg/µl | 1.76 + 1.7 | 0.29 + 0.07 |
| 260/280 | 1.7 + 0.2 | 1.8 + 0.1 |
La separación electroforética del DNA genómico obtenido por dos métodos diferentes se observa en la figura 3.
Fig. 3

Fotografía de corrida electroforética de DNA genómico. El DNA genómico extraído tanto con el método clásico y el método UMSAgen presentan peso molecular superior a 23.000bp.
Con los DNA extraídos por el método UMSAgen se realizó la amplificación del exón 12 del gen JAK-2 (Figura 4), gen presente en leucocitos.
Fig. 4

Fotografía de corrida electroforética para amplificación de la mutación JAK-2 V617F a partir de DNA. La primera columna corresponde a un paciente con trombocitosis esencial JAK-2 V617F positivo que presenta dos banda la superior es el alelo no mutado y el inferior el alelo mutado; la segunda columna corresponde a un paciente con Leucemia mieloide crónica BCRABL p210 b3a2 positivo, que presenta ambos alelos no mutado. Ambos DNA fueron extraídos por el método UMSAgen.
En el mercado internacional existen diferentes técnicas para la obtención del DNA y RNA a costos elevados. El kit UMSAgen se constituye en una técnica alternativa para la extracción del RNA y DNA simultáneamente, de manera simple, rápida y a bajo costo. Se ha realizado la comparación de precios con kits de diferentes industrias (cuadro 3)
| Kit (50 test) | Aplicación | Precio Kit $US | Precio kIT Bs* |
| UMSAgen | Obtención DNA y RNA | 141.5 | 1000 |
| DNeasy mini kit ® | Obtención DNA | 316 | 5630 |
| RNeasy mini kit ® | Obtención RNA | 477 | |
| AquaPure Genomic DNA Isolation ® | Obtención DNA | 225 | 3266 |
| AquaPure Genomic RNA Isolation ® | Obtención RNA | 235 |
* Precio aproximado, dependiente de cambio de moneda extranjera y costos de aduana y currier
DISCUSIÓN
Los métodos de extracción de RNA total y del DNA genómico han sido constantemente modificados y se han diseñado diversos métodos. En los últimos años se hicieron populares los Kits patentados que simplifican la extracción de RNA y DNA con costos cada vez mayores. Con el método de extracción UMSAgen se obtiene un RNA total de buena calidad (DO260/280 1.8 +/- 0.23) y concentración aceptable (0.11+/- 0,14 µg/µl), estos resultados son similares con los obtenidos con el método Kit Qiagen, concentración de 0,08 +/- 0.01 µg/µl y pureza de 1.9 +/- 0.16. Además el método UMSAgen permite simultáneamente la extracción del DNA genómico de alta calidad (DO260/280 1.7 +/- 0.2) y concentración de 1.76 µg/µl, cuyos resultados son similares a los DNA extraído con el método clásico concentración 0.29+/- 0,07 µg/µl y pureza de 1.8+/-0,1 µg/µl . La electroforesis del RNA extraído con el método UMSAgen presenta poca cantidad de DNA con respecto al Kit Qiagen, debido a que con el UMSAgen se extrae simultáneamente el RNA y DNA. La intensidad de las bandas que corresponden a los RNA ribosómico 28S y 18S es similar con ambas técnicas. La electroforesis del DNA extraído con el método UMSAgen presenta banda de alto peso molecular (> 23.000 bp), similar al DNA extraído por el método clásico. A partir del RNA extraído con el método UMSA gen, de los pacientes con leucemia mieloide crónica, se ha sintetizado cDNA seguido de amplificación del daño molecular BCR-ABL; todas muestras dieron positivo para este daño molecular. Por otra parte, a partir del DNA extraído con el método UMSAgen se ha amplificado para la mutación del gen JAK-2, como era de esperar, los resultados fueron negativos; porque la mutación JAK-2 esta presente solo en enfermedades mieloproliferativas. En conclusión, el método de extracción simultánea de RNA total y DNA genómico por el método UMSAgen es útil para estudios moleculares, y su ventaja se encuentra en la facilidad de extracción y bajo costo.
REFERENCIAS
1. Giorgio Corte, Paola Briata. Biologia Molecolare. Tecniche di base. 1994. Microart´s S.p.A. Genova Italia.
2. TechTalk.2007, vol 51, número 14. [ Links ]
3. Caspersson T, Schultz J (1939). “Pentose nucleotides in the cytoplasm of growing tissues”. Nature 143: 602–3
4. Ochoa S (1959). Enzymatic synthesis of ribonucleic acid. Nobel Lecture.
5. Maniatis T, Fritsch EF, Sambrock J. Molecular cloning: A laboratory Manual. Ed. 1982. Cold Spring Harbor. New York.
6. Szymanski M, Barciszewska M, Erdmann V, Barciszewski J. REVIEW, 5 S rRNA: structure and interactions. Biochem. J. (2003) 371, 641–65.
7. Van Dongen JJ, Macintyre EA, Gabert JA, Delabesse E, Rossi V, Saglio G, Gottardi E, Rambaldi A, AW, San Miguel JF, Biodi A. Standardized RT-PCR analysis of fusion gene transcripts from chromosome aberrations in acute leukemia for detection of minimal residual disease. Report of the BIOMED-1 Concerted Action. Leukemia. 1999; 13(12):1901-28. [ Links ]
8. Rambaldi, Attuati V. Amaru R, Biodi A, Barbui T. Molecular diagnosis and clinical relevance of t(9;22), t(4;11) and t(1 ;19) chromosome abnormalities in a consecutive group of 141 adult patients with acute lymphoblastic leukemia. Leuk Lymphoma. 1996 May;21(5-6):457-66. [ Links ]
9. Müller MC, Saglio G, Lin F, Pfeifer H, Press RD, Tubbs RR, Paschka P, Gottardi E, O’Brien SG, Ottmann OG, Stockinger H, Wieczorek L, Merx K, König H, Schwindel U, Hehlmann R, Hochhaus A. An international study to standardize the detection and quantitation of BCR-ABL transcripts from stabilized peripheral blood preparations by quantitative RT-PCR. Haematologica.
2007 Jul;92(7):970-3.
10. Watson JD, Crick FHC. Molecular structure of nucleic acids: A structure for deoxyribose nucleic acid. Nature 1953;171:737. [ Links ]
11. Miller SA, Dykes DD, Polesky HF. A simple salting out procedure for extraction DNA from human nucleated cells. Nucleic Acid Res. 1988; 16:1215. [ Links ]
12. Amaru R, Miguez H, Penaloza R, Torres G, Silvestre J, Cuevas H. DNA-UMSAgen, extracción de DNA genómico para diagnostico molecular. Método rápido y económico. Rev. Cuaderno 2006, 51 (2): 11-14. [ Links ]
13. Baxter EJ, Scott LM, Campbell PJ, East C, Fourouclas N, Swanton S, Vassiliou GS, Bench AJ, Boyd EM, Curtin N, Scott MA, Erber WN, Green AR. Acquired mutation of the tyrosine kinase JAK2 in human myeloproliferative disorders. Lancet. 2005 Mar 19-25;365(9464):1054-61. [ Links ]
14. Mata R, Subirá D, García-Raso A, Llamas P. JAK2 as a molecular marker in myeloproliferative diseases. Cardiovasc Hematol Agents Med Chem. 2007 Jul;5(3):198-203. [ Links ]












 uBio
uBio 

