Cuadernos Hospital de Clínicas
versión impresa ISSN 1562-6776
Cuad. - Hosp. Clín. vol.50 no.2 La Paz 2005
ARTICULO ORIGINAL
Multiresistencia antimicrobiana asociada a integrones en enteropatógenos de la diarrea infantil y Escherichia coli de la flora normal en niños menores de 5 años en la ciudad de La Paz
Claudia Rodas*, Kjetil Halvorsen*, Volga Iñiguez***
* Líc. Bioquímica
** Ph. D Estadística
*** Ph. D Biología
RESUMEN
Con el objeto de evaluar la resistencia antimicrobiana a 11 antibióticos y su relación con la presencia de integrones (clase 1-3), se analizo un total de 138 muestras que incluyen enteropatógenos (Shigella spp, EPEC, ETEC y EHEC) aislados de heces de niños con diarrea, así como E. coli no diarreogénica, aislada de heces de niños sanos de la ciudad de La Paz. Entre los marcadores de resistencia más frecuentemente encontrados, se destacan Ampicilina (95%) y Estreptomicina (90%) seguido por Cotrimoxazol (60%). Se encontró un elevado porcentaje (96%) de aislados resistentes al menos a 1 antibiótico, donde la multiresistencia de 3 hasta 6 antibióticos representó el 82%. Los aislados de Shigella spp y E. coli no diarreogénica presentaron perfiles de resistencia similares, predominando la multiresistencia ( a 4 antibióticos, en contraste a ETEC y EPEC, con menores porcentajes de resistencia. Esto sugiere que E. coli no diarreogénica puede servir de reservorio de genes de multiresistencia a otros patógenos y viceversa y que la expansión de la multiresistencia estaría relacionada a la prevalencia de enteropatógenos circulantes. Se observó una asociación significativa entre la presencia del integrón de clase 2 con la multiresistencia ( a 4 antibióticos en todos los aislados analizados y con el integrón de clase 1 (Shigella spp y E. coli no diarreogénica). La presencia de integrones estuvo asociada en mayor frecuencia a aislados resistentes a 7 antibióticos analizados. Este estudio constituye el primer reporte en nuestro medio de la multiresistencia antimicrobiana asociada a integrones detectados por métodos moleculares, y refleja la amplia distribución de los mismos, tanto en la flora normal como patogénica de la población infantil de la ciudad de La Paz. En conjunto, los datos obtenidos tienen relevancia para el tratamiento antimicrobiano de enfermedades diarreicas y control de la diseminación de la resistencia.
Palabras clave
Rev. Cuadernos 2005;50(2):38-48 Multiresistencia a antibióticos, integrón, enteropatógeno, E coli ND (E. coli no diarreogénica) EPEC (E. coli enteropatogénica), ETEC (E. coli enterotoxigénica); EHEC (E. coli enterohemorrágica), Shigella spp.
ABSTRACT
A total of 138 samples collected in the city of La Paz, including enteropathogens isolated from stools of children with diarrhea as well as fecal E. coli from healthy children, were analyzed to evalúate integron-associated antimicrobial multi-resistance distribution. The highest rate of resistance was against Ampicillin (95%), Streptomycin (90%) followed by Cotrimoxazole (60%). A high percentage (96%) of antimicrobial resistance isolates to at least one antibiotic and multi-resistance to between 3-6 antibiotics (82%) was found. Resistance patterns were similar among Shigella spp and non-diarrheogenic E coli, showing high multi-resistance prevalence to ( 4 antibiotics, in contrast to ETEC and EPEC. These data suggest, that non-diarrheogenic E coli can serve as multi-resistance genes reservoir to other pathogens and vice versa, and that multi-resistance expansion may be associated with the circulating enteropathogens prevalence. In all isolates, a significant association among class 2 integran and (4 antibiotic multi-resistance was found, as well as between class 1 integron and isolates from Shigella and non-diarrheogenic E coli. Integran presence was associated with the resistance to 7 antibiotics. This is the first report of molecular detection of integrons among pathogenic and normal flora of child population of city of La Paz demonstrating a wide integron-associated antimicrobial multi-resistance. These results are relevant to antimicrobial diarrhea treatment and resistance dissemination control.
Key words
Antibiotic multiresistance, Int (integron), enteropathogen, E. coli ND (E. coli non diarrheogenic), EPEC (E. coli enteropathogenic), ETEC (E. coli enterotoxigen); EHEC (E. coli enterohemorragic).
INTRODUCCIÓN
El aumento progresivo de resistencia antimicrobiana de los patógenos entéricos, particularmente Shigella, Escherichia coli enterotoxigénica, Salmonella typhi y Vibrio cholerae, es un problema crítico para la población que vive en países en vías de desarrollo donde existen altos porcentajes de enfermedades diarreicas agudas (EDA) que están generalmente asociadas a la mortalidad infantil(1,2,3). Se estima que en países subdesarrollados, cerca de 4 millones de niños menores de 5 años mueren por año por gastroenteritis aguda (4) donde la desnutrición es un factor adicional de alto riesgo tanto en la diarrea aguda como en la persistente donde en muchos casos, se requiere tratamiento antimicrobiano (5,6).
A pesar de que una proporción importante de las EDA son de origen infeccioso que incluye en parte a enteropatógenos bacterianos, la terapia antimicrobiana tiene un rol limitado, dependiendo del patógeno, estado del paciente, curso de la enfermedad y otros factores. En Bolivia, para tratar la shigellosis se administra como norma Sulfametoxazol-Trimetoprim (Cotrimoxazol) (7). Así también, en Chile, El Salvador, Brasil y México, el tratamiento con antimicrobianos sigue siendo la herramienta de elección para disminuir la severidad y/o muerte por shigellosis, diarrea del infante y otras enfermedades entéricas como colitis (8,9,10,11,12).
La creciente resistencia a antimicrobianos en aislados patógenos es de gran preocupación especialmente en países en vías de desarrollo, donde los antibióticos alternativos no están muy comercializados o son muy caros (13). La auto administración de antibióticos, la facilidad al acceso sin prescripción médica y su uso inapropiado y excesivo son los principales condicionantes de la resistencia bacteriana que sumadas a las condiciones sanitarias deficientes y hacinamiento permiten su rápida diseminación en regiones pobres y en desarrollo (14). Así, el proceso de selección de la resistencia es un fenómeno natural que se incrementa con el abuso y el uso indiscriminado de antibióticos en el tratamiento de las enfermedades infecciosas (15,16).
Este fenómeno afecta no solo a los patógenos bacterianos sino también a las bacterias de la flora normal, sobre todo a las provenientes del tracto intestinal que podrían adquirir multiresistencia a diferentes antibióticos debido a la frecuente exposición por la administración vía oral de los mismos. De esta manera la flora comensal puede constituirse en un reservorio de genes de resistencia (17,18).
La resistencia a diferentes antimicrobianos se encuentra en genes localizados en cromosomas, plásmidos o transposones. La transferencia horizontal de genes de multiresistencia favorece su distribución entre diferentes bacterias de una y de diferente especie (19).
En 1990, se descubrió además, un elemento genético asociado a la resistencia que involucra la integración, por el mecanismo de recombinación específica, de uno o varios genes de resistencia bajo el control de un solo promotor. Este elemento se conoce como integrón y se encuentra como parte de los transposones de la familia Tn21 o independientemente, en diferentes grupos de plásmidos y cromosomas (20).
Los integrones son sistemas de expresión genética que incorporan varios marcos de lectura abierta (ORF's) y los convierten en genes funcionales. Los integrones promueven la captura de uno o más cassettes genéticos dentro del mismo sitio de unión, por lo tanto, forman grupos o clusters de genes de resistencia antimicrobiana (21). Se conocen hasta el presente 5 diferentes clases de integrones, que se distinguen por su respectivo gen de la integrasa y que comprenden alrededor de 60 genes de resistencia organizados en casettes de resistencia (22,23,24).
La asociación de integrones con elementos móviles promueve la transmisión horizontal entre plásmidos y cromosomas así como entre diferentes replicones, contribuyendo de esta manera a la diseminación de genes de resistencia (25,26). Los integrones han sido encontrados en bacterias patógenas resistentes a antibióticos Gram negativas y Gram positivas que circulan en hospitales y en la comunidad (27,28).
En el presente trabajo, se pretende contribuir al conocimiento de los mecanismos de resistencia antimicrobiana en enteropatógenos aislados de heces de niños con procesos diarreicos y en Escherichia coli de la flora comensal aislada de muestras control (niños sanos), mediante el análisis de la resistencia a 11 diferentes antimicrobianos, asociada a la presencia y distribución de las diferentes clases de integrones.
MATERIAL Y MÉTODOS
Muestras de estudio. Se analizaron un total de 138 aislados bacterianos, de las cuales 100 muestras provenían de la colección de cepas de enterobacterias patógenas del Instituto de Biología Molecular y Biotecnología (I.B.M.B.) aisladas entre los años 1994 y 2002 a partir de heces de niños menores a 5 años con procesos diarreicos infecciosos; y las restantes 38 muestras se aislaron de niños sanos considerados como muestras control aisladas entre el 2002 y 2005.
Aislamiento, cultivo e identificación de los aislados bacterianos. Las muestras de heces fecales fueron sembradas en agar MacConkey, agar XLD y agar SS donde las colonias que crecieron fueron identificadas por las respectivas pruebas bioquímicas: SIM, TSI, LIA, Citrato de Simmons, Ureasa, Rojo Congo (29) para la identificación de aislados de Escherichia coli y Shigella spp.
Identificación de genes patógenos por medio de PCR. A los aislados que provenían de niños sanos como de niños con procesos diarreicos, se verificó la presencia de genes de patogenicidad de las siguientes categorías de E. coli diarreogénica: EPEC (eae), ETEC (lt y st) y EHEC (stx1 y stx2) por medio de PCR con los cebadores característicos para cada uno de estos genes (30). Las amplificaciones se realizaron en un termociclador Biorad Serie 5615 - B. Los productos de PCR se analizaron por medio de electroforesis en geles de agarosa al 1% a 100 Voltios en 0.5X de buffer TBE y se visualizaron en un transluminador de luz ultravioleta en presencia de bromuro de etidio (5mg/ml).
Resistencia a antibióticos. Se determinó los patrones de resistencia de los aislados bacterianos por el método de difusión en agar según las normas del NCCLS (31). Como control de calidad se emplearon las cepas Escherichia coli ATCC 25922, E. coli productora de beta-lactamasa ATCC 35218, Staphylococcus aureus ATCC 25923, Enterococcus faecalis ATCC 29212 y Pseudomonas aeruginosa ATCC 27853, con los cuales se comparó las dimensiones de las zonas de inhibición. Los agentes antimicrobianos en disco utilizados fueron; Ampicilina (Amp): 10 (g/ml, Cloranfenicol (Cm): 30 (g/ml, Estreptomicina (Sm): 10 (g/ml, Cotrimoxazol (SXT): 25 (g/ml, Tetraciclina (Tet): 30 ( g/ml, Gentamicina (Gm): 10 ( g/ml, Ciprofloxacina (Cip): 5 (g/ml, Cefoxitina (Fox): 30 (g/ml, Cefotaxima (CTX): 30 (g/ml, Ampicilina -Sulbactam (AmpSul): 10 - 10 (g/ml y Ácido Nalidíxico (AcN): 30 ( g/ml.
Detección de integrones de clase 1, 2 y 3. A todos los aislados bacterianos se realizó la detección de las diferentes clases de integrones (clase 1, clase 2 y clase 3) mediante PCR utilizando cebadores específicos (32) La mezcla de reacción contenía: 0,4 (I de dNTPs (1,25 mM de cada desoxiribonucleótido), 1 (I de MgCI2 (50mM), 2 (l de tampón PCR 1Ox, 0.12 (I de taq DNA polimerasa (Promega), 0.5 (I de cada cebador (20 (M) (Promega) y agua destilada (5,48 (I) hasta completar un volumen total de 10 (I. La amplificación se realizó en el termociclador BioRad Serie 5615 - B con las siguientes condiciones de ciclaje: un ciclo a 94°C por 5 minutos, 35 ciclos a 94°C por 1 minuto, 55°C por 1 minuto 30 segundos y 72°C por dos minutos y finalmente una extensión final de 72°C por 10 minutos. Los productos de PCR se analizaron por los mismos anteriormente descritos. Los tamaños esperados de los productos de PCR fueron de 892 pb (intI1), 467 pb (intI2) y 760 pb (intI3).
Análisis de restricción de los productos génicos amplificados de integrones de clase 1, 2 y 3 (PCR - RFLP). Los productos de PCR fueron confirmados por medio de restricción enzimática del producto de amplificación de intll con la enzima SphI. De igual manera se verificaron los productos amplificados de los genes intl2 e intl3 por restricción enzimática con HaeIII y RsaI, respectivamente.
Métodos estadísticos. Los datos obtenidos se analizaron mediante el "Programa R" con un valor de significancia de p<0.05. Se utilizó el "Test de Chisq" (33) para comparar los valores de significancia de las muestras patógenas y las muestras control. Asimismo, se utilizó el "Test de Wilcox" (33) para evaluar los valores p de multiresistencia antimicrobiana asociada con la presencia de integrones.
RESULTADOS
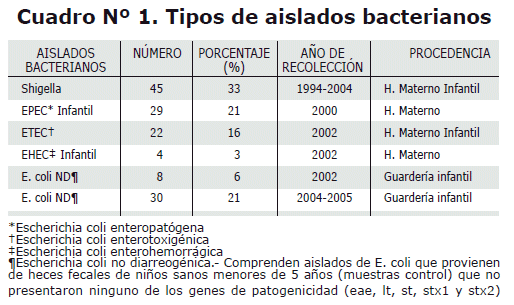
Análisis de las muestras. Del total de muestras analizadas, un 72% provinieron de niños con procesos diarreicos y un 28% de niños sanos; en estos últimos, los aislados de E. coli fueron definidos como E. coli ND (E. coli no diarreogénica), los cuales no presentaron los genes de patogenicidad analizados por PCR. El porcentaje de cada tipo de aislado bacteriano entérico se muestra en el Cuadro N° 1 con un promedio de 29 muestras.
Análisis bacteriano y perfiles de resistencia.
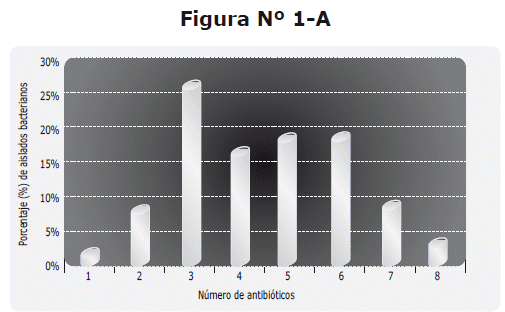
EL 96% (132) de los 138 aislados analizados, presentaron resistencia a por lo menos 1 antibiótico. En la Figura 1A se observa el porcentaje de resistencia a entre 1 y 8 antibióticos en el total de los aislados bacterianos estudiados, donde predominaron los aislados resistentes de 3 hasta 6 antibióticos que, en conjunto, representaron el 82% (n = 110).
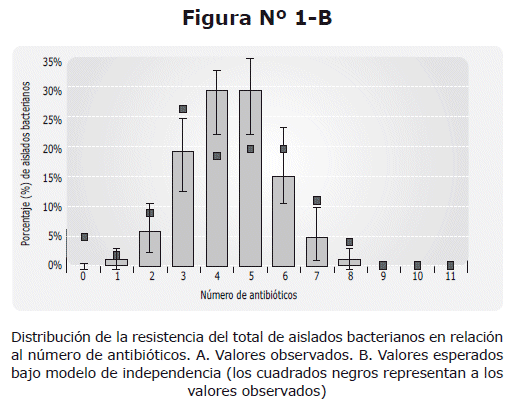
El análisis estadístico de la distribución del porcentaje de aislados resistentes al número de antibióticos analizados, mostró que la proporción de resistencia observada fue significativamente diferente a los valores esperados (p<0.05) (Figura N° 1B) bajo un modelo de independencia. Esto es evidente sobre todo para los valores de resistencia a 3, 4, 5, 7 y 8 antibióticos así como para la sensibilidad a todos los antibióticos.
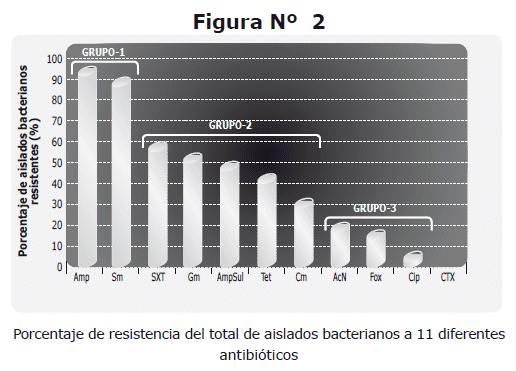
En la Figura N° 2 se observa el porcentaje de resistencia que presentaron los aislados bacterianos a cada uno de los 11 antimicrobianos por separado, siendo la resistencia a Amp la más frecuentemente encontrada, seguida por la resistencia a Sm. Estos dos marcadores de resistencia se encontraron en porcentajes mayores al 90% en los aislados bacterianos estudiados por lo que se los clasificó convencionalmente como pertenecientes al Grupo 1 (alta frecuencia). En el Grupo 2 (frecuencia intermedia) se encontraron a los marcadores de resistencia a SXT (60%), Gm (54%), AmpSul (48%), Tet (42%) y Cm (30%). Finalmente, el Grupo 3 (baja frecuencia) que comprende al AcN (15%), Fox (13%) y Cip (2%) hacia los cuales los porcentajes de resistencia en los aislados bacterianos fueron menores al 20%.
Resistencia a antibióticos en los aislados de Shigella y en las diferentes categorías de E. coli. Se analizó el porcentaje de aislados resistentes en relación al número de antibióticos analizados en cada enteropatógeno por separado así como en E. coli no diarreogénica (Figura 3). En los aislados de Shigella, se observó la resistencia desde 2 hasta 8 antibióticos con predominio de la multiresistencia a 5 (29%) y 6 antibióticos (20%). Los aislados de E. coli no diarreogénica mostraron resistencia desde 2 hasta 8 antibióticos con porcentajes máximos de multiresistencia a 4 y 6 antibióticos (26 y 24%, respectivamente) donde los aislados resistentes a 2, 3, 5, 7 y 8 antibióticos no fueron mayores al 16%. En EPEC, se encontraron aislados resistentes desde 3 hasta 8 antibióticos, siendo la multiresistencia a 3 antibióticos la más común y la que alcanzó el 41% en contraste a la multiresistencia a 4 y 5 antibióticos (10% y 13%, respectivamente). Los aislados de ETEC, mostraron resistencia a un solo antibiótico en un 8%, siendo la única categoría que presentó este patrón. El rango de aislados resistentes comprendió desde 1 hasta 6 antibióticos en contraste a Shigella, EPEC y E. coli no diarreogénica. Por una parte, no se observó multiresistencia a 7 y 8 antibióticos, por otra, predominó la resistencia a 2 antibióticos (32%). Finalmente, se observaron 3 aislados de ETEC y 2 aislados de E. coli no diarreogénica que fueron sensibles a los 11 antibióticos analizados (datos no mostrados). Los datos de EHEC, no fueron incluidos en los porcentajes de aislados resistentes presentados por no ser significativamente equivalentes con los otros aislados bacterianos. Sin embargo, en estos aislados la proporción de multiresistencia a 3 y 4 antibióticos fue de 2/4 (datos no mostrados).
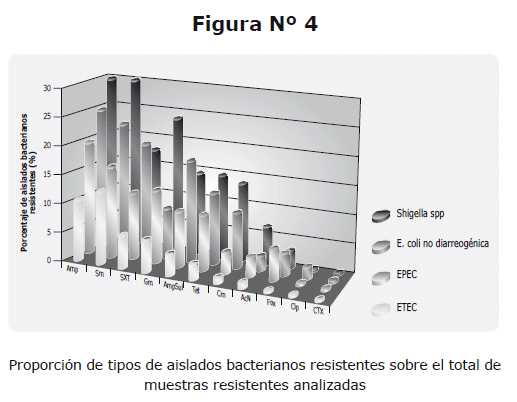
A su vez, el análisis de la resistencia a cada antibiótico por separado mostró que los aislados de Shigella spp, E. coli no diarreogénica y EPEC fueron los que presentaron patrones similares de resistencia a Amp, Sm, SXT, Tet, AcN y Fox. Se observaron diferencias en relación al mayor porcentaje de aislados resistentes a Gm en Shigella spp (78%) en contraste a E. coli no diarreogénica; y a AmpSul en E. coli no diarreogénica (70%) en contraste a Shigella spp. Por su parte, ETEC presentó en todos los aislados analizados menores porcentajes de resistencia a todos los antibióticos analizados a excepción de Sm.
Estos datos fueron corroborados en el análisis de la resistencia de cada tipo de aislado bacteriano sobre el total de aislados resistentes, observándose que Shigella spp y E. coli no diarreogénica representan la mayor proporción, seguidos de EPEC y finalmente ETEC (Figura N° 4).

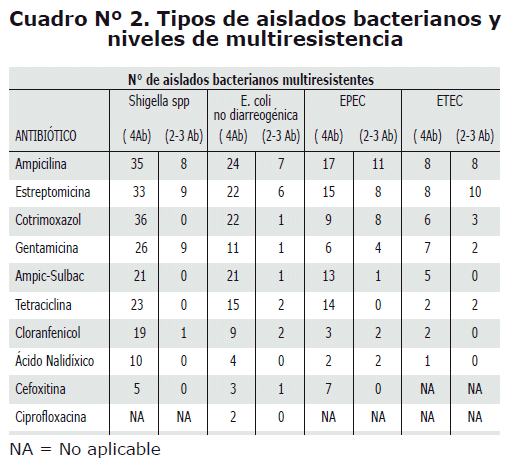
Nivel de multiresistencia a antibióticos en aislados bacterianos entéricos. Se consideraron 2 criterios de multiresistencia: la asociada a entre 2 y 3 antibióticos y la multiresistencia (a 4 antibióticos. En Shigella como en E. coli no diarreogénica, el total de aislados resistentes estuvieron asociados significativamente (p<0.05) a la multiresistencia ( a 4 antibióticos, en contraste tanto a EPEC donde la multiresistencia a 5 antibióticos (Amp, Sm, AmpSul, Gm y Cm), como a ETEC donde la resistencia a solo 2 antibióticos (SXT y Gm) estuvieron asociados a la multiresistencia) a 4 antibióticos (Cuadro N° 2).
Integrones y resistencia antimicrobiana. La presencia de integrones en los aislados bacterianos fue analizada por la amplificación de los fragmentos de los genes correspondientes a las clases 1, 2 y 3. En la Figura 5 se observa las tres clases de integrones, con su respectivo marcador de peso molecular. El 74% del total de aislados bacterianos analizados presentaron integrones (clase 1, 2 o 3); siendo el integrón de clase 2 el de mayor frecuencia (62%), en comparación con los integrones de clase 1 (37%) y clase 3 (5%) (p<0.05).
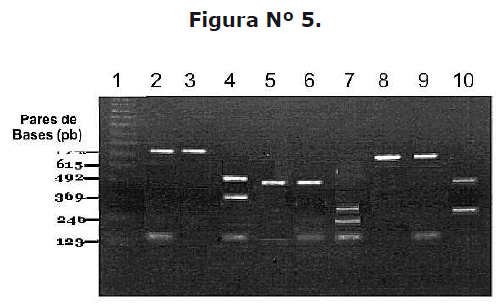
Carriles: 1, marcador de peso molecular (2693, 1603, 1198, 794, 615, 492, 369, 246, 123); 2 y 3, integrón de clase 1 (892 pb); 4, restricción enzimática con Sphl (493 y 399); 5 y 6, integren de clase 2 (467 pb); 7, restricción enzimática con Hae III (270 y 197 pb); 8 y 9, integrón de clase 3 (760 pb); 10, restricción enzimática con Rsal (500 y 260 pb). Identificación molecular y restricción enzimática de los integrones de clase 1, 2 y 3
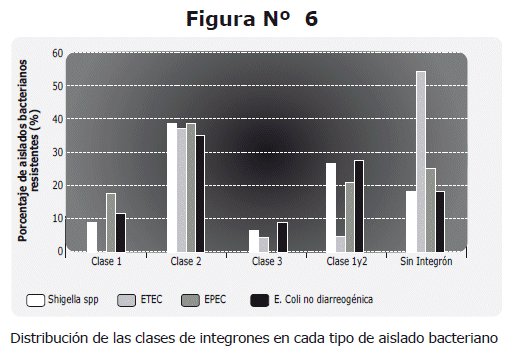
El integrón de clase 2 se observó en proporciones similares en todos los aislados analizados en un rango de 35% a 38%. Resalta la ausencia de integrones en los aislados de ETEC (55%) en comparación con Shigella y E. coli no diarreogénica (18%) y EPEC (22%). Tanto en Shigella como en E. coli no diarreogénica se observaron proporciones similares del integrón de clase 1 y en combinaciones conjuntas de la clase 1 y clase 2 que contrastan con los aislados en ETEC. Se observó también que EPEC fue la categoría bacteriana que relativamente presentó la mayor proporción del integrón de clase 1 (Figura 6). El análisis de los datos estadísticos mostró que la asociación con el integrón de clase 2 es la más significativa (x2gL =45.46).
Nivel de multiresistencia y presencia de integrones. La presencia de integrones (clase 1 y 2) fue analizada entre los aislados bacterianos con distintos niveles de multiresistencia. En el Cuadro 3 se observa que el integrón de clase 2 fue el que presentó asociación significativa a la multiresistencia (a 4 antibióticos en todos los tipos de aislado bacteriano, en contraste al integrón de clase 1 que solo mostró este patrón en Shigella y E. coli no diarreogénica) (p<0.05).
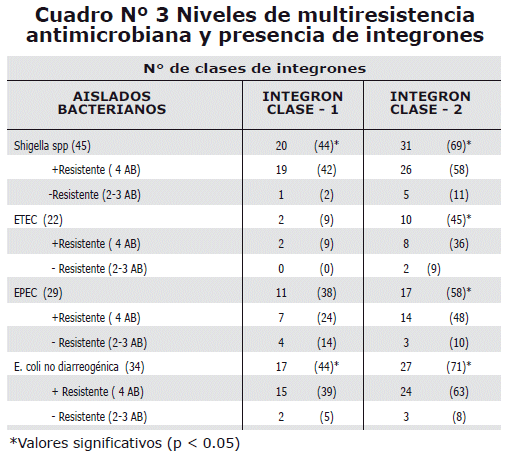
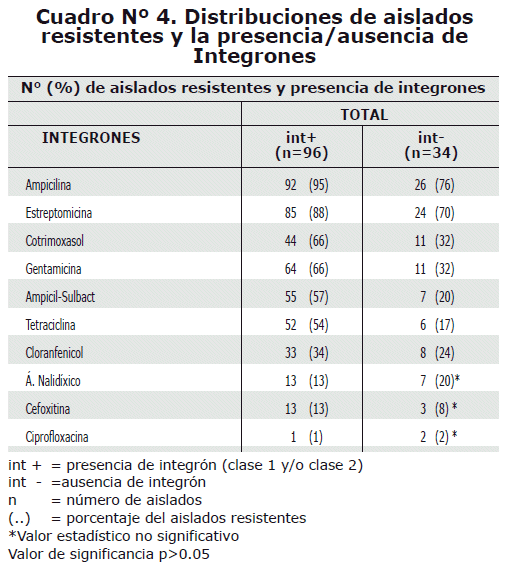
Resistencia antimicrobiana y asociación con integrones. El análisis entre la presencia de integrones y resistencia a los antibióticos analizados mostró que en el total de aislados que presentaron integrones (Cuadro 4) se observaron mayores frecuencias de resistencia (p<0.05) para los siguientes antibióticos Tet, SXT, AmpSul Amp, Gm (excepto en Shigella spp) y Sm con porcentajes mayores en un 37%, 34%, 37%, 19%, 34% y 18%, respectivamente, en comparación a los no portadores de integrones.
DISCUSIÓN
Población estudiada y resistencia a antibióticos
En el presente estudio, en la muestra analizada representativa de 4 diferentes enteropatógenos aislados a partir de procesos diarreicos infecciosos y de E coli de la flora comensal de niños sanos, se observa que la multiresistencia a antibióticos esta ampliamente diseminada. Estos datos reflejan claramente la presión de selección existente, por el extensivo e inadecuado uso de antibióticos en nuestro medio, donde pese al costo biológico de la resistencia conjunta a diferentes antibióticos, las bacterias multiresistentes a 3 y 6 antibióticos fueron las mas frecuentemente encontradas (82%) en significativo contraste a una reducida proporción (4%) de bacterias sensibles a los 11 antibióticos testados.
Por una parte, la flora normal comensal del tracto intestinal al estar sujeta a una exposición frecuente a los antibióticos, sobre todo de aquellos que se administran por vía oral, seria un reservorio importante de genes de multiresistencia (34,17,18) con capacidad potencial de transmisión a organismos patógenos y podría además considerarse una bacteria indicadora de la multiresistencia circulante en otros patógenos. Se esperaría entonces, mayor similitud entre los patrones de multiresistencia de los enteropatógenos más prevalentes con los de las bacterias de la flora normal, como lo observado en este trabajo entre Shigella y E. coli no diarreogénica, en contraste a EHEC y ETEC. Esto implicaría, que existen diferencias en cuanto a la exposición a los antimicrobianos, que estarían también dependiendo de factores como el grado de circulación en la población humana, reservorios animales, etc. Al respecto, es interesante observar que aunque EPEC y ETEC tienen porcentajes de prevalencia similares, esta última categoría contrasta por su sensibilidad a varios de los antibióticos testados, por lo que futuros estudios deberán confirmar estos resultados, así como validar los datos de EHEC en un número mayor de muestras.
Es importante considerar además, que en el trabajo se consideraron antibióticos utilizados en el tratamiento de gastroenteritis, así como de otras enfermedades infecciosas que permite evaluar la magnitud y extensión de los marcadores de resistencia circulantes, observándose tanto en E. coli no diarreogénica como en los patógenos entéricos, resistencia a ambos tipos de antibióticos.
En este contexto, pese a que los antimicrobianos suministrados a niños son mas restringidos que los de adultos, la resistencia observada al marcador Tet, indicaría que la colonización de la flora intestinal a través de la transmisión de bacterias multiresistentes presentes en la comunidad, es muy común, por lo que los niños portarían bacterias multiresistentes a antibióticos a los cuales no habrían estado necesariamente expuestos.
En cuanto a la distribución de los diversos marcadores de resistencia en la población analizada se puede inferir que dado que las muestras provienen de diversos periodos, la multiresistencia no es un fenómeno exclusivo a los últimos años por lo que periódicamente deberán evaluarse las tendencias de incremento/decremento de la resistencia a los antibióticos mas utilizados.
Los marcadores de resistencia a Amp y Sm resaltan por su elevada frecuencia en la población analizada, que en caso de confirmarse éstos valores en estudios mas extensivos, deberá considerarse la eliminación de su uso en los tratamientos de enfermedades infecciosas, puesto que además de inefectivos, contribuirían a limitar aun mas las opciones de la terapia antimicrobiana, co-seleccionando a otros marcadores de resistencia asociados.
Tradicionalmente, la Ampicilina, Trimetoprima y Cotrimoxazol, se han usado mundialmente en el tratamiento de la diarrea, siendo este ultimo considerado la droga de elección en el tratamiento de shigellosis en nuestro país (7). La resistencia al Cotrimoxazol, que constituye el tercer marcador de mayor frecuencia, es significativa por la alta proporción de aislados resistentes (60%), por presentar porcentajes similares de resistencia en E. coli no diarreogénica, Shigella y EPEC, así como por su asociación a la multiresistencia (a 4 antibióticos, estando presente en 5 de las 9 combinaciones significativas encontradas. Estos datos apuntan en conjunto, a una reconsideración del uso extensivo del Cotrimoxazol y a un control estricto en base a la información del patrón de sensibilidad de las cepas circulantes, para prevenir la expansión de bacterias resistentes, la cual tendría un efecto negativo en el control de la diarrea sanguinolenta causada por Shigella. En otros estudios a nivel mundial, se ha reportado de forma similar la resistencia a antimicrobianos de patógenos entéricos (1,10,11,15,35,36,37) y de E. coli proveniente de heces fecales de niños sanos (34) a los antimicrobianos mas comunmente usados como el Cotrimoxazol.
Por otra parte, los marcadores de resistencia que se encuentran en baja frecuencia (Grupo 3), en particular AcN, Fox y Cip podrían servir como alternativa, en caso de ser necesario, en el tratamiento de diarreas bacterianas (38) y se diferencian por su distribución en aislados de otras regiones (39-41) donde se observan porcentajes de resistencia mayores.
Integrones y resistencia a antibióticos
Dada la creciente relevancia de los integrones como factores principales de acumulación y diseminación de la multiresistencia a antibióticos, los estudios de asociación de la multiresistencia con la distribución y prevalencia de las clases de integrones son importantes para comprender a nivel molecular los mecanismos de resistencia.
Del conjunto de tipos de integrones, los de clase 1 y 2 son generalmente los más frecuentes en bacterias resistentes. En este trabajo, la alta prevalencia de integrones encontrada en el total de aislados mayoritariamente resistentes (Figura 6, Cuadro N° 4), refleja que estos elementos son comunes en los genomas de la población estudiada, en particular el integrón de clase 2 encontrado en todos los tipos de aislados analizados, seguido del de clase 1 y en contraste al integrón de clase 3. Estos datos, difieren de otros estudios (8,42-47) donde el integrón de clase 1 es el mas encontrado en aislados Gram negativos, particularmente en Enterobacterias, aunque coincide con lo reportado recientemente por Peirano et al. 2005 (48) en aislados de Shigella.
La asociación entre los integrones de clase 1 y/o 2 con la multiresistencia a antibióticos se puede inferir a partir de las siguientes observaciones:
1. La presencia de integrones (clase 1 y 2) esta significativamente asociada a la resistencia a gran parte de los antibióticos analizados (Cuadro N° 3 y 4).
2. La ausencia de integrones es mas común en los aislados de ETEC (55%) y EHEC (datos no mostrados) que son las categorías patogénicas de E. coli que presentaron menores proporciones de resistencia a los antibióticos analizados, en comparación a Shigella y E. coli no diarreogénica (mayoritariamente resistentes), donde la prevalencia de los integrones de clase 1 y 2 fue la mayor (Figura N° 6).
3. En todos los aislados (Cuadro N° 3), a excepción de EHEC (datos no mostrados), la multiresistencia (a 4 antibióticos está significativamente asociada a la presencia de al menos uno de los integrones analizados).
La adquisición de integrones es considerada como una de las principales causas de multiresistencia antimicrobiana en aislados Gram negativos, especialmente en aislados entericos. Se ha reportado que aislados que portan integrones, presentan mayor resistencia a los antibióticos, que aquellos que no los portan, así como la presencia de integrones en la flora normal de adultos (45,49-50). Se podría deducir que la transferencia de integrones entre bacterias de una misma especie y entre especies diferentes se lleva a cabo a partir de una estructura básica y no mediante genes individuales que facilitaría la diseminación de los integrones y por consiguiente, de las resistencias relacionado a ellos (27)
La alta prevalencia de la multiresistencia antimicrobiana asociada a integrones en la flora comensal y en patógenos que afectan a la población infantil que es mas vulnerable a las enfermedades infecciosas, significa un riesgo a la salud pública, en el contexto global donde éstas, provocan anualmente la muerte de alrededor de 11 millones de niños, mas del 90% de los cuales se producen en países en desarrollo. Si bien muchas diarreas de origen bacteriano son auto-limitantes y no justifican el tratamiento antimicrobiano, las complicaciones asociadas a la desnutrición o a otras enfermedades intercurrentes pueden requerir su uso. Así, en el manejo de la shigellosis, el uso apropiado de antibióticos juega un papel importante en el tiempo de recuperación, y disminución de la excreción del patógeno. El incremento de la multiresistencia disminuye el espectro de antibióticos efectivos disponibles, que desde ya es limitado en niños, sin considerar las limitaciones económicas del costo de las nuevas generaciones de antibióticos.
Al ser éste, el primer reporte de la asociación entre multiresistencia e integrones, futuros estudios deberán analizar el contenido genético de los casettes genéticos presentes en los integrones encontrados así como su relación con los fenotipos de resistencia.
AGRADECIMIENTOS
Este trabajo fue realizado en el Instituto de Biología Molecular y Biotecnología (I.B.M.B.) con el apoyo parcial del Programa de colaboración sueca ASDI - SAREC: Proyecto Enterobacterias y constituyo parte del trabajo de tesis de grado en Bioquímica de la Lic. Claudia Rodas. Los autores agradecen al personal del Hospital del Niño Ovidio Aliaga y Hospital Materno Infantil CNS.
REFERENCIAS
1. Guerrero I. Changing resistance in enteric pathogens. Jacksonville Med 2001; 2: 1 -3 [ Links ]
2. Tarreja N, Mohan B. Antimicrobial resistance in selected bacterial enteropathogens in north India. J. Indian Med Res 2004; 120: 39 - 43 [ Links ]
3. Bennish M. Treatment of infectious diarrhea: the role of antimicrobial therapy. APUA Newsletter 1995; 13 (4): 1 - 6 [ Links ]
4. Torres M, Pírez M. Etiology of children's diarrhea in Montevideo, Uruguay: associated pathogens and unusual isolates. J. Clin Microbiol 2001; 39 (6): 2134 - 2139. [ Links ]
5. Lawler J, Wallace M. Diagnosis and treatment of bacterial diarrhea. Curr Gastroenterol Rep 2003; 5: 287 - 294 [ Links ]
6. Okeke I, Fayinka S. Antibiotic resistance in Escherichia coli from Nigerian students, 1986 - 1998. Emerg Infect Dis 2000; 6 (4): 393 -395 [ Links ]
7. Zamora A, Cordero G. Evaluación de la Estrategia "Atención Integrada a las Enfermedades Prevalentes de la Infancia" (AIEPI) en Servicios de Salud, Bolivia. Ministerio de Salud y Previsión Social 1999 [ Links ]
8. Reyes A, Bello H, Domínguez M. Prevalence and types of class 1 integrons in aminoglycoside - resistant Enterobacteriaceae from several Chilean hospitals. J. Antimicrob Chemother 2003; 51: 317 - 321 [ Links ]
9. Nolasco C. AIEPI: Argumentos técnicos para la estrategia de atención integral al niño. Rev Pediatr El Salvador 2005; 1 (8):1 - 15 [ Links ]
10. Lima A, Pinho C. High frequeney of strains multiply resistant to ampicillin, trimethoprim sulfametoxazole, streptomycin, chloramfenicol and tetracyeline isolated from patient with shigellosis in Brazil during the period 1988 to 1993. Antimicrob Agents Chemother 1995; 39(1): 256 - 259 [ Links ]
11. Solórzano - Santos F. Resistencia de bacterias respiratorias y entéricas a antibióticos. Salud Pública Mex 2003; 40 (6): 510 - 516 [ Links ]
12. Tolia V. Acute infectious Diarrhea in Children. Curr Treat Opt Infect Dis 2002; 4: 183 - 194 [ Links ]
13. Vila J, Vargas M. Antimicrobial Resistance of Diarrheagenic Escherichia coli isolated from children under the age of 5 years from Ifakara, Tanzania. Antimicrob Agents Chemother 1999. 43 (12): 3022 - 3024 [ Links ]
14. Ashkenzi S. Antibiotic treatment of bacterial gastroenteritis. J. Pediatr Infect Dis 1991; 10: 140 - 148. [ Links ]
15. Sack R. Antimicrobial resistance in organism causing diarrheal diseases. Clin Infect Dis 1997; 24: 102 -105. [ Links ]
16. Merino L, Hreñuk G. Resistencia a antibióticos y epidemiología molecular de Shigella spp en el nordeste argentino. Rev Panam Salud Pública 2004; 15 (4): 219 - 122 [ Links ]
17. Millar M, Walsh T. Carriage of antibiotic resistant bacteria by healthy children. J. Antimicrob Chemother 2001; 47: 605 - 610 [ Links ]
18. Barreta J. Evaluación de la resistencia a antibióticos de cepas de E. coli de muestras fecales provenientes de niños sanos en edad escolar de la ciudad de La Paz. Tesis de Licenciatura. Universidad Mayor de San Andrés. La Paz, Bolivia; 2002 [ Links ]
19. De la Cruz I, Davies I. Horizontal gene transfer and the origin of species: lessons from bacteria. Trends Microbiol 2000; 2: 128 - 133 [ Links ]
20. Levesque C, Piche L, Larose C, Roy P. PCR mapping of integrons reveals several novel combinations of resistance genes. Antimicrob Agents Chemother 199; 39 (1): 185-191.
21. Carattoli A. Importance of integrones in the diffusion of resistance. INRA Sciences 2001; 32: 243 - 259 [ Links ]
22. Collis C, Kim MJ, Partridge SR, Stokes HW, Hall RM. Characterization of the class 3 integran and the site-specific recombination system it determines. J. Bacteriol 2002; 184: 3017 - 3026. [ Links ]
23. White P, Rawlinson D. Current status of the aadA and dfr gene cassette families. J. Antimicrob Chemother 2001; 47: 495 - 496 [ Links ]
24. Collis C, Hall R.M. Site specific deletion and rearrangement of integran insert genes catalyzed by the integran DNA integrase. J. Bacteriol 1992; 174: 1574 - 1585. [ Links ]
25. Melver C, White P, Jones L. Integrons and gene cassettes in Enterobacteriaceae. Antimicrob Agents Chemother 2001; 45: 2658 - 2661 [ Links ]
26. Roy P. Integrons: novel mobile genetic elements mediating antibiotic resistance in Enterobacteria and Psuedomonas. APUA Newsletter 1995; 13(3): 1, 4-6 [ Links ]
27. Gallardo F, Ruiz J, Soto SM. Distintos mecanismos de resistencia asociados a integrones en aislamientos clínicos de Salmonella typhimurium. Rev Esp Quimioter 2003; 16(4): 398 - 402 [ Links ]
28. Correia M. Molecular characterization of a new class 3 integran in Klebsiella pneumoniae. Antimicrob Agents Chemother 2003; 47 (9): 2838 - 2843 [ Links ]
29. Flores A, Suarez J. Four biochemical tests for identification of probable enteroinvasive Escherichia coli and Shigella strains. Rev Latinoam Microbiol 1999; 41: 259 - 261 [ Links ]
30. Reitan M, Betting K. Identification of virulent genes in different isolates of Escherichia coli from healthy children. J. Bacteriol 1999; 13: 13 - 21 [ Links ]
31. Cona E. Condiciones para un buen estudio de susceptibilidad mediante test de difusión en agar. Rev Chil Infect 2002; 19 (2): 77 - 81 [ Links ]
32. Muñoz J, Domínguez M, Zambrana R. Integrones y cassettes genéticos de resistencia a antimicrobianos en cepas de Shigella flexneri. Rev Med Chil 2003; 34: 1-10 [ Links ]
33. Efrón B. Large - Scale simultaneous Hypothesis testing: The choise of a nuil hipótesis. J. Am Stat Assoc 2004; 99 (465): 96 - 110 [ Links ]
34. Infante B, Grape M, Larsson M, Kristiansson C, Pallecchi L, Rossolini GM, Kronvall G. Acquired sulphonamide resistance genes in fecal Escherichia coli from healthy children in Bolivia and Peru. J. Antimicrob Agents 2005; 25 (4): 308 - 312. [ Links ]
35. Prats G, Mirelis B, Llovet C, Muñoz E and Navarro F. Antibiotic resistance trends in enteropathogenic bacteria isolated in 1985- 1987 and 1995-1998 in Barcelona. Antimicrob Agents Chemother 2000; 44: 1140 - 1145.
36. Vila J, Vargas T, Ruiz J, Corachan M, De Anta M, and Gascon J. Quinolone resistance in enterotoxigenic Esherichia coli causing diarrhea in travelers to India in comparison with other geographical areas. Antimicrob. Agents Chemother 2000; 44: 1731 - 1733.
37. Hoge C, Gambel W, Srijan A, Pitarangsi C. Trends in antibiotic resistance among diarrheal pathogens isolated in Thailand over 15 years. Clin. Infect. Dis 1998; 26: 341 - 345. [ Links ]
38. Navia M, Capitano L, Ruiz J, Vargas M. Typing and characterization of mechanism of resistance of Shigella spp. Isolated form feces of children under 5 years of age from Ifakara, Tanzania. J. Clin Microbiol 1999; 37 (10): 3113 - 3117. [ Links ]
39. Nordmann P, Poirel L. Emergence of plasmid-mediated resistance to quinolones in Enterobacteriaceae. J Antimicrob Chemother 2005; 56 (3): 463 - 469. [ Links ]
40. Murray A, Coia JE, Mather H, Brown DJ. Ciprofloxacin resistance in non-typhoidal Salmonella serotypes in Scotland, 1993-2003. J Antimicrob Chemother 2005; 56 (1): 110 -114. [ Links ]
41. Fernandez-Cuenca F, Amblar G, Pascual A, Martinez-Martinez L. Relationships between resistance to cefoxitin, ciprofloxacin and amoxicilin-clavulanic acid in Escherichia coli clinical isolates. Enferm Infecc Microbiol Clin. 2005; 23 (8): 510 - 511 [ Links ]
42. Kang SG, Lee DY, Shin SJ, Ahn JM, Yoo HS. Changes in patterns of antimicrobial susceptibility and class 1 integron carriage among Escherichia coli isolates. J Vet Sci. 2005; 6 (3): 201 - 205. [ Links ]
43. Nogrady N, Gado I, Toth A, Paszti J. Antibiotic resistance and class 1 integron patterns of non-typhoidal human Salmonella serotypes isolated in Hungary in 2002 and 2003. Int J Antimicrob Agents 2005; 26 (2): 126 - 132. [ Links ]
44. Fonseca EL. Vieira W, Cipriano R, Vicente AC. Class 1 integrons in Pseudomonas aeruginosa isolates from clinical settings in Amazon región, Brazil. FEMS Immunol Med Microbiol 2005; 44 (3): 303 - 309 [ Links ]
45. Kang HY, Jeong YS, Tae SH, Choi CH, Moon DC, Lee WK, Lee YC, Seol SY, Cho DT, Lee JC. Characterization of antimicrobial resistance and class 1 integrons found in Escherichia coli isolates from humans and animals in Korea. J Antimicrob Chemother 2005; 55 (5): 639 - 644. [ Links ]
46. Zhang H, Zhang X, Yamasaki S, Miyoshi S, Shinoda S. Identification and characterization of class 1 integran resistance gene cassettes among Salmonella strains isolated from healthy humans in China. Microbiol Immunol 2004; 48 (9): 639 - 645. [ Links ]
47. Tosini F, Visca P, Luzzi I, Dionisi AM, Pezzella C, Petrucca A, Carattoli A. Class 1 integran-borne multiple-antibiotic resistance carried by IncFI and IncL/M plasmids in Salmonella entérica serotype typhimurium. Antimicrob Agents Chemother 1998; 3: 3053 - 3058. [ Links ]
48. Peirano G, Agerso Y, Aarestrup FM, Prazeres Rodrigues D. Occurrence of integrons and resistance genes among sulphonamide-resistant Shigella spp. from Brazil. Antimicrob Chemother. 2005; 55 (3): 301 -305. [ Links ]
49. Mazel D, Dychinco B, Webb VA, Davies J: Antibiotic resistance in the ECOR collection: integrons and identification of a novel aad gene. Antimicrob Agents Chemother 2000; 44: 1568 - 1574. [ Links ]
50. Skurnik D, Le Menac'h A, Zurakowski D, Mazel D, Courvalin P, Denamur E, Andremont A, Ruimy R. Integron-associated antibiotic resistance and phylogenetic grouping of Escherichia coli isolates from healthy subjects free of recent antibiotic exposure. Antimicrob Agents Chemother 2005; 49 (7): 3062 - 3065. [ Links ]












 uBio
uBio 

