Ecología en Bolivia
versión impresa ISSN 1605-2528versión On-line ISSN 2075-5023
Ecología en Bolivia vol.51 no.2 La Paz set. 2016
Artículo
Una introducción a la estructura filogenética de comunidades: Un caso de estudio en palmas de Bolivia
An introduction to phylogenetic community structure: a case study in Bolivian palms
Francisco J. Velásquez-Puentes1,2 & Christine D. Bacon1
1Department of Biological and Environmental Sciences, University of Gothenburg, Box 461, SE-405 30 Göteborg, Suecia.
2Departamento de Química y Biología, Universidad del Norte, Km.5 vía Puerto Colombia, Barranquilla, Colombia.
Autores de correspondencia: velasquez.puentes.francisco@gmail.com, christinedbacon@gmail.com
Resumen
La estructura filogenética de comunidades estudia los patrones de dispersión filogenética y de diversidad de los linajes evolutivos a lo largo del espacio geográfico, además es una herramienta valiosa para inferir procesos ecológicos y macroevolutivos que determinan la distribución y coexistencia de especies. En este trabajo estudiamos la diversidad y estructura filogenética de cuatro comunidades regionales de palmas en Bolivia definidas por las siguientes unidades fitogeográficas: Amazonía, Andes, Cerrado y Gran Chaco. Nuestros resultados indican: 1) una alta diversidad filogenética en las comunidades de los Andes y de la Amazonía, 2) agrupamiento filogenético para las especies de los Andes y del Cerrado, y 3) diferenciación filogenética entre tres de las cuatro comunidades regionales (Amazonía, Andes y Cerrado). Sugerimos que los patrones de estructura y diversidad filogenética observados fueron producidos principalmente por la diversificación in situ y por los efectos de filtros ambientales que parecen ser importantes en las regiones de los Andes y el Cerrado. Además, sugerimos que el alto grado de recambio filogenético encontrado puede haber sido mediado por las condiciones climáticas regionales y por barreras biogeográficas como el levantamiento Andino. Estos resultados resaltan la importancia de la información filogenética en los estudios de ensamblaje de comunidades.
Palabras clave: Arecaceae, Bolivia, Estructura filogenética, Macroevolución.
Abstract
The field of community phylogenetics is focused on the study of phylogenetic structure and diversity of evolutionary lineages across geographic space and represents a valuable tool to infer ecological and macroevolutionary processes determining species distributions and co-existence. In this study we analyzed patterns of phylogenetic diversity and structure from four regional palm communities of Bolivia defined by main phytogeographical units: Amazon, Andes, Cerrado, and Gran Chaco. Our results indicate: 1) high phylogenetic diversity for the Andean and Amazonian regional communities, 2) phylogenetic clustering for the species from the Andes and the Cerrado, and 3) phylogenetic differentiation between three of the four studied regional communities (Amazon, Andes, and Cerrado). We suggest the obtained patterns of phylogenetic diversity and structure were produced mainly by in situ diversification and by the effects of environmental filters that seem to be important in the Andes and the Cerrado. In addition, the high degree of phylogenetic turnover found, could have been mediated by regional climatic conditions and by biogeographic barriers such as the Andean uplift. These results highlight the relevance of phylogenetic information on community assembly studies.
Keywords: Areacaceae, Bolivia, Phylogenetic structure, Macroevolution
Introducción
La biodiversidad es un concepto complejo que abarca entidades biológicas, evolutivas, y fenotípicas en escalas de tiempo y espacio diferentes (Pavoine & Bonsall 2011). Esta diversidad se distribuye heterogéneamente en el espacio geográfico, patrón que puede ser explicado, entre otros, por diversos factores ambientales, biológicos, e históricos que han ido moldeando la distribución de especies en la tierra (Gaston 2000).
El estudio del ensamblaje de comunidades se encarga de entender los patrones que determinan la distribución de especies dentro de una comunidad y su dinámica, lo que ha sido uno de los objetivos más desafiantes y más viejos de la ecología (Pavoine & Bonsall 2011, Cavalheri et al. 2015). En los últimos años la inclusión de información filogenética en el estudio de comunidades ha demostrado ser de gran utilidad para esclarecer los procesos que determinan el ensamblaje de comunidades en escalas espaciales y temporales distintas (Webb et al. 2002, Cavender-Bares et al. 2009).
Webb et al. (2002) propusieron que el patrón de las relaciones filogenéticas entre especies puede reflejar los mecanismos que hacen posible su coexistencia dentro de una comunidad. Asumiendo conservatismo de nicho, en donde especies cercanamente emparentadas son más similares en cuanto a sus requerimientos ecológicos que especies lejanamente emparentadas (Peterson 1999), un patrón filogenético en donde las especies de una comunidad están lejanamente relacionadas (sobredispersión filogenética) resaltaría el papel de la competencia entre especies como mecanismo principal del ensamblaje, ya que sólo especies con requerimientos ecológicos distintos podrían coexistir. Por otro lado, si las especies dentro de la comunidad se encuentran cercanamente emparentadas (agrupamiento filogenético) el mecanismo de ensamblaje estaría mediado por filtros ambientales en donde las condiciones bióticas favorecen la coexistencia de especies con requerimientos y características similares, las cuales les permiten sobrevivir en determinados tipos de hábitats. Alternativamente un patrón aleatorio de las relaciones filogenéticas entre las especies de la comunidad apuntaría a mecanismos de ensamblaje neutros como la deriva ecológica, o a un balance en los efectos de las interacciones inter-específicas y los filtros ambientales (Purves & Pacala 2005).
La interpretación de los mecanismos de ensamblaje de las comunidades depende de la escala espacial siendo considerada. A escalas espaciales grandes por ejemplo a nivel continental, el agrupamiento filogenético de una muestra regional es el resultado de procesos biogeográficos e históricos que operan también a grandes escalas temporales (Webb et al. 2002, Ricklefs 2004). A nivel local los filtros ambientales son más importantes, mientras que las interacciones denso-dependientes predominarían en escalas más pequeñas (Cavender-Bares et al. 2009). Las características fenotípicas involucradas en la especialización ambiental y su grado de homoplasia (i.e. qué tan lábil es su evolución) también son relevantes a la hora de interpretar los mecanismos de ensamblaje. Por ejemplo, cuando especies cercanas se han especializado en nichos diferentes (i.e. no hay conservatismo de nicho), las especies más distantemente relacionadas pueden coexistir en una comunidad como resultado de filtros ambientales operando en rasgos convergentes (Losos et al. 2003, Losos 2008), generando de esta manera un patrón de sobredispersión filogenética que generalmente se obtendría como resultado de mecanismos de competencia inter-específica. Entretanto, el desplazamiento de caracteres generado por la competencia inter-específica puede permitir la coexistencia de especies cercanas y generar patrones filogenéticos agrupados que comúnmente se producen por la acción de los filtros ambientales (Cavender-Bares et al. 2009).
Además de los mecanismos tradicionales (filtros ambientales e interacciones inter-específicas), existen otros procesos que pueden moldear la estructura filogenética de las comunidades, como por ejemplo las interacciones planta-polinizador, la especificidad entre herbívoros y patógenos, la dinámica de nichos temporales, la hibridación y los mecanismos de especiación (Cavender-Bares et al. 2009).
Tradicionalmente los índices usados para medir la estructura o grado de dispersión filogenética de las comunidades son el índice de relación neta y el del taxón más cercano, denominados NRI (Net Relatedness Index) y NTI (Nearest Taxon Index) respectivamente por sus siglas en inglés. Ambos índices se calculan de forma similar de acuerdo a la siguiente ecuación (Webb et al. 2002, 2008):
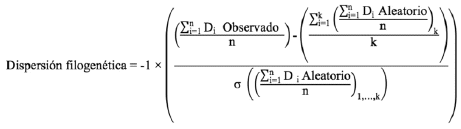
Di representa la distancia filogenética entre un par de especies i en la comunidad o en otras palabras la longitud de las ramas que conectan a un determinado par de especies a lo largo de una filogenia, n representa el número de pares de especies implementados y k el número de iteraciones o veces que se re-organizan aleatoriamente las especies dentro de la filogenia. Para el caso del índice NRI la distancia Di se calcula para cada combinación posible de pares de especies dentro de la comunidad, mientras que para el NTI esta distancia se calcula para un set limitado de especies conformado por cada especie dentro de la comunidad y su especie más cercana dentro del mismo grupo. Podemos ver entonces que el NRI captura la información filogenética global, a diferencia del NTI que captura la información filogenética más reciente (nodos y ramas más cercanos a las puntas terminales de la filogenia). Para cada índice se obtiene el promedio de las distancias filogenéticas observadas entre los pares de especies y se mide qué tan diferente es éste del obtenido al promediar las distancias filogenéticas promedio resultantes de cada aleatorización. La diferencia es estandarizada por la desviación estándar de los promedios obtenidos en cada aleatorización del 'pool' de especies. Para ambos índices, valores positivos indican agrupamiento filogenético, valores negativos denotan sobredispersión filogenética y valores cercanos a cero indican un patrón de ensamblaje aleatorio (Webb et al. 2002).
Además de estos dos índices, existen otras medidas que también se usan para caracterizar la diversidady dinámicafilogenéticade comunidades. Una de ellas es el índice de diversidad filogenética PD (Phylogenetic Diversity) propuesto por Faith (1992), que es equivalente a la suma de las longitudes de rama que conectan a todos los pares de especies dentro de una comunidad a lo largo de una filogenia. Por otro lado, análogo a la diversidad beta (definida como el recambio de especies a lo largo de las comunidades y el espacio geográfico; Whittaker 1972, Graham & Fine 2008), existe también la diversidad filogenética beta o recambio filogenético que mide el cambio en las relaciones filogenéticas entre las especies a lo largo de las comunidades. Existen diferentes medidas de recambio filogenético que comparan las relaciones evolutivas entre pares conformados por especies dentro y entre comunidades, como por ejemplo la medida de recambio filogenético πst (Hardy & Senterre 2007) y los análogos al NRI y NTI: β NRI y β NTI (Webb et al. 2008). Las medidas de recambio filogenético son de gran utilidad cuando se estudian en función de la heterogeneidad ambientaly topográfica para determinar la dinámica de los patrones filogenéticos de comunidades a lo largo de gradientes ambientales y espaciales, complementando así los estudios de ensamblaje y estructura filogenética de comunidades (Graham & Fine 2008).
La familia de las palmas (Arecaceae) es considerada un grupo modelo (Couvreur & Baker 2013, Bacon 2013) que ha sido extensamente estudiado y para el cual existe amplia información morfológica y filogenética. Las palmas de Bolivia comprenden alrededor de 100 especies clasificadas en 28 géneros (Moraes 2015) y distribuidas en distintos tipos de hábitat, incluyendo bosques húmedos tropicales, andinos, secos y xerofíticos (Moraes 2007). En este estudio usamos las comunidades regionales de palmas de cuatro unidades fitogeográficas representativas de Bolivia (Amazonía, Andes, Gran Chaco y Cerrado) con el propósito de determinar su estructura e identidad filogenética.
Métodos
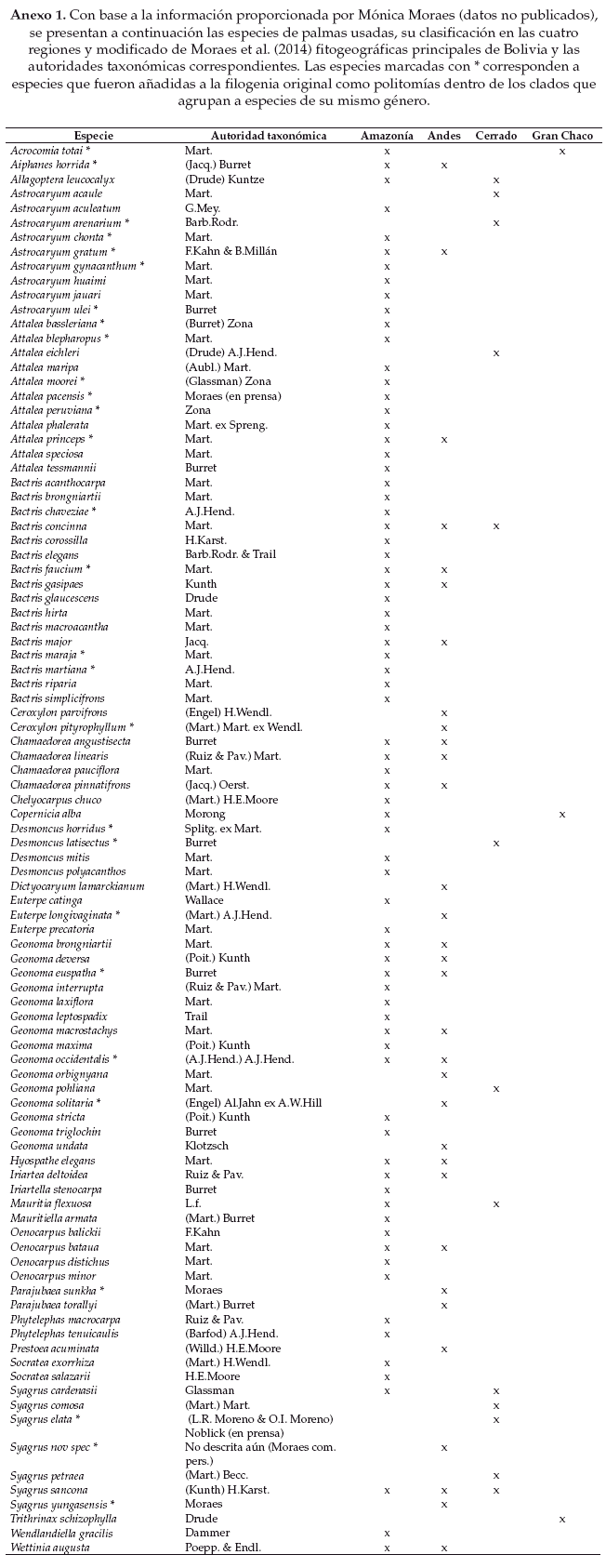
Para nuestro estudio utilizamos 96 especies de las 100 especies de palmas reportadas actualmente para Bolivia (Moraes 2015). Las especies fueron clasificadas en cuatro grupos de comunidades regionales de acuerdo a su presencia dentro de las principales unidades fitogeográficas del país (Moraes 2007): Amazonía, Andes, Gran Chaco y Cerrado.
Para este estudio, la información filogenética de las especies de palmas de Bolivia fue extraída de una filogenia global para las especies de palmas de las Américas obtenida a partir de secuencias de ADN de un gen nuclear (RPB2) y de un gen cloroplástico (matK) (Bacon et al. en preparación). Veintiocho especies de palmas de Bolivia no están presentes en la filogenia, así que estas especies fueron añadidas como politomías dentro del clado que agrupa a las especies del mismo género (Anexo 1).
Los índices de dispersión filogenética fueron estimados usando el paquete Picante v. 1.6-2 (Kembel et al. 2010) implementado en la plataforma estadística R v. 3.2.1 (R Core Team 2015). Picante implementa los índices SESMPD (Standardized Effect Size of MeanPairwise Distance) y SESMNTD (Standardized Effect Size of Mean Nearest Taxon Distance) que son equivalentes a los índices NRI y NTI de acuerdo a la siguiente relación (Webb et al. 2008):
![]()
Adicionalmente estimamos el grado de diversidad filogenética dentro de cada comunidad utilizando el índice de diversidad filogenética PD (Phylogenetic Diversity) empleando también el paquete Picante. Finalmente el índice πst de recambio filogenético (Hardy & Senterre 2007) fue estimado usando el paquete SpacodiR v. 0.13 (Eastman et al. 2011).
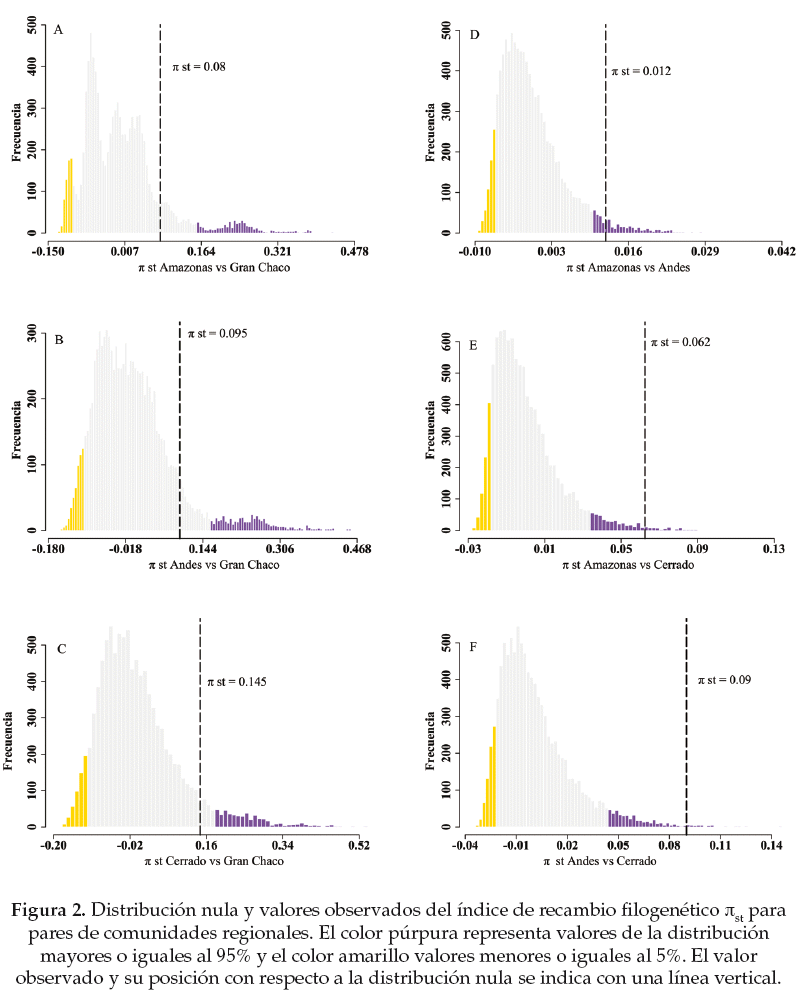
Para establecer la significancia estadística de los resultados observados para el SESMPD, SESMNTD, y πst se implementaron 10000 aleatorizaciones de las especies presentes a lo largo de la filogenia usando la función "taxa.labels" en Picante y la función "resamp.1a" en SpacodiR. Elegimos un nivel de significancia estadística del 5%, en donde las comunidades pueden diferir significativamente de un ensamblaje al azar y encontrarse filogenéticamente sobredispersas (p > 0.95), filogenéticamente agrupadas (p < 0.05), o no diferir significativamente de un ensamblaje aleatorio (p > 0.05 y p < 0.95). En el caso del recambio filogenético, valores observados mayores al 95% de la distribución nula que se obtiene a partir de la aleatorización, indican que las especies dentro de una misma comunidad se encuentran en promedio más cercanamente emparentadas que especies de diferentes comunidades, mientras que valores menores al 5% indican que las especies de diferentes comunidades se encuentran en promedio más cercanamente emparentadas que especies de una misma comunidad (Hardy & Senterre 2007).
Resultados
El árbol filogenético usado y la clasificación de las especies en las unidades fitogeográficas se muestran en la figura 1. Nuestros resultados de diversidad filogenética (medida en millones de años) indican una mayor diversidad para la comunidad regional de palmas de la Amazonía (PD = 2015.84), seguida por las especies de palmas de los Andes (PD = 1074.11), Cerrado (PD = 523.87) y por la comunidad regional del Gran Chaco que muestra la menor diversidad (PD = 272.82). Este patrón también se ve reflejado en el número de especies de palmas presentes en cada región: 75 especies para la Amazonía, 32 para los Andes, 13 para el Cerrado, y 3 para el Gran Chaco.
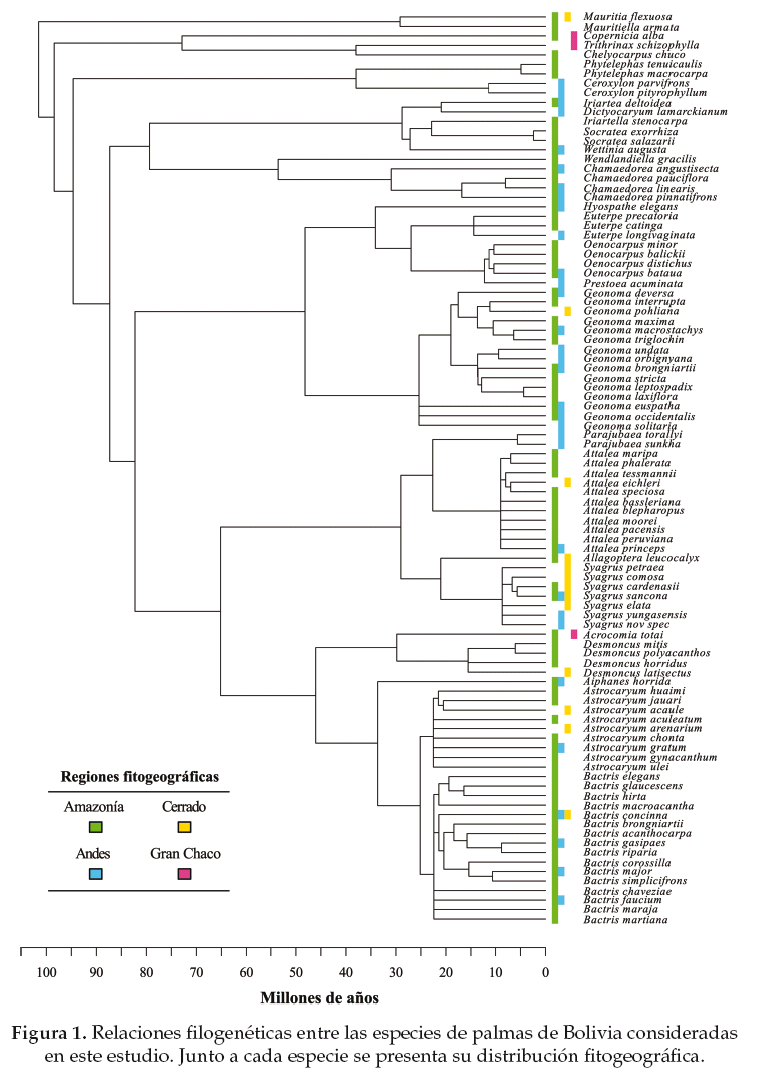
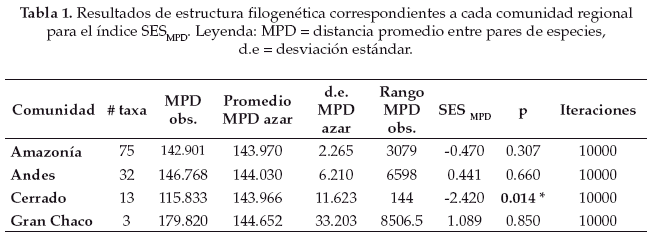
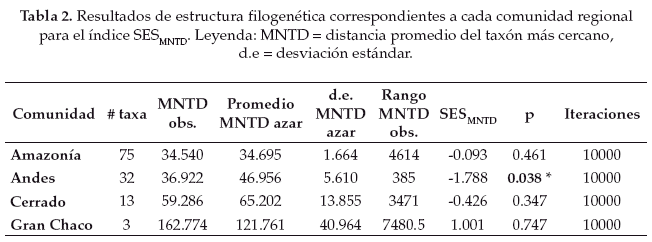
De acuerdo al grado de estructura filogenética obtenido para cada comunidad regional (Tablas 1-2) podemos ver que tanto el SESMPD como el SESMNTD indican un patrón de estructura filogenética aleatorio para las comunidades regionales de la Amazonía y del Gran Chaco, aunque los índices difieren en los patrones obtenidos para las comunidades regionales de los Andes y del Cerrado. Para la comunidad regional del Cerrado el SESMPD indica que existe agrupamiento filogenético entre las especies (p < 0.05), mientras que el SESMNTD muestra un patrón que no difiere significativamente de un ensamblaje aleatorio. En el caso de la comunidad regional de los Andes el SES indica un patrón de estructura filogenética aleatorio, mientras que el SESMNTD indica agrupamiento filogenético (p < 0.05).
La medida de recambio filogenético implementada (π st) indica que las especies de palmas de las regiones de los Andes, Cerrado y de la Amazonía se encuentran diferenciadas filogenéticamente (p > 0.95, Fig. 2 d-f) es decir que en promedio las especies dentro de cada una de estas regiones se encuentran más cercanamente emparentadas entre si que entre especies de regiones distintas, o en otras palabras que las especies de palmas de estas regiones se encuentran agrupadas filogenéticamente con respecto a las demás regiones. Para el caso de la región del Gran Chaco no detectamos una diferenciación filogenética significativa de las especies con respecto a las otras tres regiones (Fig. 2 a-c).
Discusión
Las regiones de los Andes y de la Amazonía se caracterizan por ser extraordinariamente biodiversas y por presentar altos niveles de endemismos (Myers et al. 2000, Antonelli & Sanmartín 2011). A diferencia de la Amazonía oriental, la región occidental que incluye parte del territorio de Bolivia, Colombia, Ecuador, Perú y el occidente de Brasil, se caracteriza por su alta diversidad tanto a nivel de especies (Ter Steege et al. 2003, Hoorn et al. 2010) como en diversidad filogenética (Honorio-Coronado et al. 2015). Para Bolivia, la fracción correspondiente a la región fitogeográfica de la Amazonía también abarca la mayor diversidad de especies de palmas del país seguida por los Andes (Moraes 2007, Moraes et al. 2014). De forma análoga, nuestros resultados resaltan la mayor diversidad filogenética de palmas para la región de la Amazonía de Bolivia seguida por la región andina. Esta alta diversidad filogenética puede ser explicada por diversos factores. Según Honorio-Coronado et al. (2015), la alta diversidad filogenética y riqueza de especies de plantas presentes en la región occidental de la Amazonía podría haberse derivado del intercambio de linajes entre los Andes y la región fronteriza de la Amazonía occidental; adicionalmente los autores plantean que las condiciones fértiles y la baja estacionalidad de la región favorecerían la colonización de linajes con historias evolutivas diferentes. Alternativamente, la alta diversidad filogenética de la Amazonía puede ser producto de complejos procesos de diversificación in situ relacionados con la adaptación y la especialización edáfica (Fine et al. 2005). Para el caso de la comunidad regional de los Andes, el levantamiento Andino que ha promovido el aislamiento geográfico y la diversificación en la región (Luebert & Weigend 2014) puede explicar la alta diversidad filogenética encontrada. Por su parte, las regiones fitogeográficas del Cerrado y del Gran Chaco presentan la menor diversidad filogenética del conjunto de palmas de Bolivia. Estas regiones se caracterizan por tener ambientes con prolongadas estaciones secas (Gottsberger & Silberbauer-Gottsberger 2006) que pueden actuar como barreras ecofisiológicas y limitar la diversidad de linajes evolutivos (Honorio-Coronado et al. 2015). Por otro lado, los patrones de diversidad filogenética encontrados se ven reflejados en el número de especies de palmas en cada región. Se ha demostrado que existe una correlación entre la diversidad filogenética y la riqueza de especies (Rodrigues & Gaston 2002), aunque esta relación puede variar a nivel biogeográfico (Forest et al. 2007). A diferencia de la riqueza de especies, la diversidad filogenética añade información sobre las relaciones entre especies que son producto de procesos evolutivos, por lo que en conjunto con la diversidad taxonómica y funcional es un índice útil para establecer estrategias de conservación e identificar áreas clave para la biodiversidad (Davies & Buckley 2011, Brooks et al. 2015).
De acuerdo al grado de estructura filogenética, vemos que la comunidad regional de los Andes y del Cerrado de Bolivia se encuentran filogenéticamente agrupadas (Tabla 2). Es importante notar que este agrupamiento filogenético es significativo para el SESMNTD en la comunidad regional de los Andes y para el SESMTD en la comunidad regional del Cerrado. Dado que el SESMNTD es más sensible a la información filogenética cerca a las puntas terminales de la filogenia, los mecanismos que han contribuido a la coexistencia de especies cercanas dentro de estas dos regiones pueden haber operado en tiempos diferentes, siendo estos más recientes para la región Andina. Adicionalmente, valores significativos para el NTI o SESMNTD pueden surgir como resultado de procesos de diversificación in situ (Vamosi et al. 2009, Hennequin et al. 2014). En particular, el levantamiento de los Andes juega un papel importante como barrera biogeográfica que ha promovido la diversificación in situ enriqueciendo la biodiversidad de la región (Hughes & Eastwood 2006, Madriñán et al. 2013, Luebert & Weigend 2014). Sumado a esto, el levantamiento de los Andes regula el desplazamiento de especies animales dispersoras de semillas (Eiserhardt et al. 2011) y presenta un amplio rango de condiciones climáticas que imponen fuertes demandas fisiológicas para los organismos (Nakazato etal. 2008, Projecto-Garcia et al. 2013, DuBay & Witt 2014), que actúan como filtros ambientales favoreciendo los patrones de agrupamiento filogenético (Graham et al. 2009). De igual forma en la región del Cerrado, las fuertes sequías, incendios naturales recurrentes, y suelos pobres en nutrientes característicos de la región (Gottsberger & Silberbauer-Gottsberger 2006) pueden actuar como filtros ambientales que promueven el agrupamiento filogenético. Otros estudios en palmas reportan también este patrón, por ejemplo, Loiola et al. (2012) estudiaron comunidades locales de especies leñosas del Cerrado y detectaron patrones de agrupamiento filogenético; Kissling et al. (2012) atribuyen un incremento en el agrupamiento filogenético de las comunidades de palmas neo-tropicales de regiones secas y estacionales a las oscilaciones climáticas durante el periodo geológico Cuaternario; y Weigelt et al. (2015) encontraron que el agrupamiento filogenético atribuido a la baja capacidad de dispersión y al aislamiento geográfico en comunidades de palmas de islas es el patrón predominante.
A diferencia de las comunidades regionales del Cerrado y de los Andes, se encontró una dispersión filogenética aleatoria para las comunidades regionales de la Amazonía y del Gran Chaco de Bolivia. Una compleja variedad de interacciones y de procesos ecológicos y evolutivos pueden generar efectos opuestos de agrupamiento y sobredispersión que resultan en una señal filogenética difusa y una dispersión aleatoria (Purves & Pacala 2005, Cavender-Bares et al. 2009), lo que puede ser el caso de la comunidad de palmas amazónicas. Para la región del Gran Chaco, que contribuye con una fracción muy pequeña (2.4 %) del total de especies de palmas presentes en Bolivia, el poder estadístico puede no ser lo suficientemente grande para detectar un patrón de estructura filogenética diferente al azar. Según Kraft et al. (2007) el poder estadístico para detectar estructura filogenética se reduce en comunidades donde hay muy pocas especies o una cantidad de especies similar al 'pool' de especies regional, favoreciendo la hipótesis nula de ensamblaje aleatorio cuando ésta es falsa (error tipo II).
Nuestros resultados de recambio filogenético apuntan a que las especies de palmas dentro de cada región fitogeográfica están más cercanamente emparentadas entre si que con especies de otras regiones, excepto para la región del Gran Chaco. Este es un resultado muy importante dado que implica que en las tres regiones estructuradas filogenéticamente (Amazonía, Andes y Cerrado) existen procesos evolutivos que operan independientemente y que promueven la diversificación de especies dentro de las regiones, generando la diversidad y los patrones de estructura filogenética observados. Según Pennington et al. (2006), sonpocos los estudios que han determinado si los eventos de diversificación ocurren dentro de los biomas o entre los biomas (i.e. a lo largo de las fronteras que los separan), nuestro estudio sugiere que en el caso de las palmas de la Amazonía, del Cerrado y de los Andes, la diversificación in-situ es el mecanismo principal que ha contribuido con el ensamblaje de las comunidades a nivel regional. Por su parte, en la comunidad regional del Gran Chaco la falta de diferenciación filogenética puede atribuirse a que son pocas las especies de palmas presentes en esta región de Bolivia para detectar diferencias significativas, o a que las especies de palmas allí presentes se originaron en diferentes regiones fitogeográficas. Adicionalmente se han reportado factores que están fuertemente relacionados con el recambio filogenético de palmas en el Neotrópico, como barreras biogeográficas fuertes (i.e. los Andes) o sutiles (i.e. sabanas) que favorecen la evolución de nichos, el aislamiento geográfico de las especies y el recambio filogenético (Eiserhardt et al. 2013, Kissling et al. 2012). También se ha reportado para las especies de palmas neotropicales que la diferenciación filogenética se incrementa con la diferenciación de los nichos climáticos (Eiserhardt et al. 2013). Por lo tanto la adaptación de las especies de palmas a las condiciones climáticas presentes en cada región y el aislamiento causado por las barreras biogeográficas como los Andes, pueden ser factores que probablemente han promovido el recambio o diferenciación filogenética a lo largo de las regiones fitogeográficas estudiadas.
Conclusiones
Los patrones de diversidad, recambio y estructura filogenética obtenidos para las comunidades regionales de palmas de Bolivia, reflejan mecanismos ecológicos como los filtros ambientales y procesos macroevolutivos como la diversificación in situ, que probablemente contribuyeron con el ensamblaje de las comunidades de palmas regionales. Nuestros resultados indican que la diversificación in situ ha operado independientemente en las regiones de la Amazonía, los Andes y el Cerrado, promoviendo la diversidad de palmas de las comunidades a nivel regional. Además de esto, para la comunidad regional de los Andes y del Cerrado, los filtros ambientales relacionados con condiciones climáticas severas, han contribuido posiblemente en la configuración de los ensamblajes limitando la coexistencia de linajes filogenéticamente distantes, mientras que para la comunidad regional de la Amazonía la ausencia de un patrón (i.e. agrupado o sobredisperso) representa posiblemente un equilibrio entre los efectos de los filtros ambientales y la variedad de interacciones ecológicas de una región que por su alta diversidad filogenética refleja también una larga y rica historia evolutiva. Dada la baja cantidad de especies de palmas para el Gran Chaco en Bolivia, sería adecuado extender el muestreo a otras localidades de esta región fitogeográfica fuera del territorio boliviano y de esta forma obtener un muestreo representativo. Finalmente la diferenciación filogenética entre las comunidades regionales de palmas puede haber sido mediada por barreras biogeográficas y por las condiciones climáticas regionales. Vemos entonces que existe un gran potencial en el estudio de la estructura y la diversidad filogenética de comunidades, que además de tener grandes implicaciones y usos en conservación, aportan elementos y nuevas dimensiones al entendimiento de los factores ecológicos y evolutivos que determinan la distribución y coexistencia de las especies a lo largo del espacio geográfico.
Agradecimientos
Agradecemos la invitación de Mónica Moraes para someter este manuscrito a la revista Ecología en Bolivia. FJV-P fue apoyado por una beca de la Royal Swedish Society of Arts and Sciences Kungl. Vetenskaps akademien otorgada a C.D. Bacon y CDB fue apoyada por una beca del Consejo Europeo de Investigación bajo el Séptimo Programa Marco de Investigación y Desarrollo Tecnológico (PM/2007-2013, ERC Grant Agreement n. 331024) otorgada a A. Antonelli.
Referencias
Antonelli, A. & I. Sanmartín. 2011. Why are there so many plant species in the Neotropics? Taxon 60(2): 403-414.
Bacon, C.D. 2013. Biome evolution and biogeographical change through time. Frontiers of Biogeography 5(4): 227-231. [ Links ]
Brooks, T.M., A. Cuttelod, D.P. Faith, J. Garcia-Moreno, P. Langhammer & S. Perez-Espona. 2015. Why and how might genetic and phylogenetic diversity be reflected in the identification of key biodiversity areas? Philosophical Transactions of the Royal Society B. 370(1662): 20140019.
Cavalheri, H., C. Both & M. Martins. 2015. The interplay between environmental filtering and spatial processes in structuring communities: the case of neotropical snake communities. PLOS ONE 10(6): e0 127959.
Cavender-Bares, J., K.H. Kozak, P V A. Fine & S. W. Kembel. 2009. The merging of community ecology and phylogenetic biology. Ecology Letters 12(7): 693-715.
Couvreur, T.L. & W.J. Baker. 2013. Tropical rain forest evolution: palms as a model group. BMC Biology 11(1): 48.
Davies, T.J. & L.B. Buckley. 2011. Phylogenetic diversity as a window into the evolutionary and biogeographic histories of present-day richness gradients for mammals. Philosophical Transactions of the Royal Society B. 366(1576): 2414-2425.
DuBay, S.G. & C.C. Witt. 2014. Differential high-altitude adaptation and restricted gene flow across a mid-elevation hybrid zone in Andean tit-tyrant flycatchers. Molecular Ecology 23(14): 3551-3565.
Eastman, J.M., C.E.T. Paine & O.J. Hardy. 2011. spacodiR: structuring of phylogenetic diversity in ecological communities. Bioinformatics 27(17): 2437-2438.
Eiserhardt, W.L., J.-C. Svenning, W.J. Baker, T.L.P. Couvreur, and H. Balslev. 2013. Dispersal and niche evolution jointly shape the geographic turn over of phylogenetic clades across continents. Scientific Reports 3:1164.
Eiserhardt, W.L., J.-C. Svenning, W.D. Kissling & H. Balslev. 2011. Geographical ecology of the palms (Arecaceae): determinants of diversity and distributions across spatialscales. Annals of Botany 108(8): 1391-1416.
Faith, D.P. 1992. Conservation evaluation and phylogenetic diversity. Biological Conservation 61(1): 1-10. [ Links ]
Fine, P.V.A., D.C. Daly, G. Villa Muñoz, I. Mesones & K.M. Cameron. 2005. The contribution of edaphic heterogeneity to the evolutionand diversity of Burseraceae trees in the western Amazon. Evolution 59(7): 1464-1478.
Forest, F., R. Grenyer, M. Rouget, T.J. Davies, R.M. Cowling, D.P. Faith, A. Balmford, J.C. Manning, S. Procheş, M. van der Bank, G. Reeves, T.A. Hedderson & V. Savolainen. 2007. Preserving the evolutionary potential of floras in biodiversity hotspots. Nature 445(7129): 757-760.
Gaston, K.J. 2000. Global patterns in biodiversity. Nature 405(6783): 220-227. [ Links ]
Gottsberger, G. & I. Silberbauer-Gottsberger. 2006. Origin, structure, dynamics andplant use. Life in the Cerrado a South American tropical seasonal ecosystem.1. Reta Verlag, Ulm. 280 p.
Graham, C.H. & P V. A. Fine. 2008. Phylogenetic beta diversity: linking ecological and evolutionary processes across space in time. Ecology Letters 11(12): 1265-1277.
Graham, C.H., J.L. Parra, C. Rahbek & J. A. McGuire. 2009. Phylogenetic structure in tropical hummingbird communities. Proceedings of the National Academy of Sciences 106: 19673-19678.
Hardy, O.J. & B. Senterre. 2007. Characterizing the phylogenetic structure of communities by an additive partitioning of phylogenetic diversity. Journal of Ecology 95(3): 493-506.
Hennequin, S., M. Kessler, S. Lindsay & H. Schneider. 2014. Evolutionary patterns in the assembly of fern diversity onthe oceanic Mascarene Islands. Journal of Biogeography 41(9): 1651-1663.
Honorio Coronado, E.N., K.G. Dexter, R.T. Pennington, J. Chave, S.L. Lewis, M.N. Alexiades, E. Alvarez, A. Alves de Oliveira, I. L. Amaral, A. Araujo-Murakami, E. J.M.M. Arets, G. A. Aymard, C. Baraloto, D. Bonal, R. Brienen, C. Cerón, F. Cornejo Valverde, A. Di Fiore, W. Farfan-Rios, T.R. Feldpausch, N. Higuchi, I-. Huamantupa-Chuquimaco, S.G. Laurance, W.F. Laurance, G. López-Gonzalez, B.S. Marimon, B. HurMarimon-Junior, A. Monteagudo Mendoza, D. Neill, W. Palacios Cuenca, M.C. Peñuela Mora, N.C. A. Pitman, A. Prieto, C. A. Quesada, H. Ramirez Angulo, A. Rudas, A.R. Ruschel, N. Salinas Revilla, R.P. Salomão,A. Segalin de Andrade, M.R. Silman, W. Spironello, H. ter Steege, J. Terborgh, M. Toledo, L. Valenzuela Gamarra, I.C.G. Vieira, E. Vilanova Torre, V. Vos& O.L. Phillips. 2015. Phylogenetic diversity of Amazonian tree communities. Diversity and Distributions 21(11): 1295-1307.
Hoorn, C., F.P. Wesselingh, H. ter Steege, M.A. Bermudez, A. Mora, J. Sevink, I. Sanmartin, A. Sanchez-Meseguer, C.L. Anderson, J.P. Figueiredo, C. Jaramillo, D. Riff, F.R. Negri, H. Hooghiemstra, J. Lundberg, T. Stadler, T. Särkinen & A. Antonelli. 2010. Amazonia throughtime: Andean uplift, climate change, landscape evolution, and biodiversity. Science 330(6006): 927-931.
Hughes, C. & R. Eastwood. 2006. Island radiation on a continental scale: Exceptional rates of plant diversification after up lift of the Andes. Proceedings of the National Academy of Sciences 103(27): 10334-10339.
Kembel, S.W., P.D. Cowan, M.R. Helmus, W.K. Cornwell, H. Morlon, D.D. Ackerly, S.P. Blomberg & C.O. Webb. 2010. Picante: R tools for integrating phylogenies and ecology. Bioinformatics 26(11): 1463-1464.
Kissling, W.D., W.L. Eiserhardt, W.J. Baker, F. Borchsenius, T.L.P. Couvreur, H. Balslev & J.-C. Svenning. 2012. Cenozoic imprints on the phylogenetic structure of palm species assemblages worldwide. Proceedings of the National Academy of Sciences 109(19): 7379-7384.
Kraft, N.J.B., W.K. Cornwell, C.O. Webb & D.D. Ackerly. 2007. Trait evolution, community assembly, and the phylogenetic structure of ecological communities. The American Naturalist 170(2): 271-283.
Loiola, P.P., I.A. Silva, D.M. Silva & M.A. Batalha. 2012. Underdispersion of anti-herbivore defence traits and phylogenetic structure of cerrado tree species atfine spatial scale. Journal of Vegetation Science 23(6): 1095-1104.
Losos, J.B. 2008. Phylogenetic niche conservatism, phylogenetic signal and the relationship between phylogenetic relatedness and ecological similarity among species. Ecology Letters 11(10): 995-1003. [ Links ]
Losos, J.B., M. Leal, R.E. Glor, K. de Queiroz, P.E. Hertz, L. Rodríguez Schettino, A. Chamizo Lara, T.R. Jackman & A. Larson. 2003. Niche lability inthe evolution of a Caribbean lizard community. Nature 424(6948): 542-545.
Luebert, F. & M. Weigend. 2014. Phylogenetic insights into Andean plant diversification. Frontiers in Ecology and Evolution 2: 27.
Madriñán, S., A.J. Cortés & J.E. Richardson. 2013. Páramo is the world's fastest evolving and coolest biodiversity hotspot. Frontiers in Genetics 4: 192.
Moraes R., M. 2007. Phytogeographical patterns of Bolivian palms. Palms 51(4): 177-186. [ Links ]
Moraes R., M. 2015. Actualización de la lista de especies nativas de Arecaceae para Bolivia. Revista de la Sociedad Boliviana de Botánica 8(1): 17-26. [ Links ]
Moraes R., M., B. Rios-Uzeda, L.R. Moreno, G. Huanca-Huarachi & D. Larrea-Alcázar. 2014. Using potential distribution models for patterns of species richness, endemism, and phytogeography of palm species in Bolivia. Tropical Conservation Science 7(1): 45-60.
Myers, N., R.A. Mittermeier, C.G. Mittermeier, G.A.B. da Fonseca & J. Kent. 2000. Biodiversity hotspots for conservation priorities. Nature 403(6772): 853-858.
Nakazato, T., M. Bogonovich & L.C. Moyle. 2008. Environmental factors predict adaptive phenotypic differentiation within and between two wild Andean tomatoes. Evolution 62(4): 774-792.
Pavoine, S. & M.B. Bonsall. 2011. Measuring biodiversity to explain community assembly: a unified approach. Biological Reviews 86(4): 792-812.
Pennington, R.T., J.E. Richardson & M. Lavin. 2006. Insights into the historical construction of species-rich biomes from dated plant phylogenies, neutral ecological theory and phylogenetic community structure. New Phytologist 172(4): 605-616.
Peterson, A.T. 1999. Conservatism of ecological niches in evolutionary time. Science 285(5431): 1265-1267. [ Links ]
Projecto-Garcia, J., C. Natarajan, H. Moriyama, R.E. Weber, A. Fago, Z.A. Cheviron, R. Dudley, J.A. McGuire, C.C. Witt & J.F. Storz. 2013. Repeated elevational transitions in hemoglobin function during the evolution of Andean hummingbirds. Proceedings of the National Academy of Sciences 110(51): 20669-20674.
Purves, D.W. & S.W. Pacala. 2005. Ecological drift in niche-structured communities: neutral pattern does not imply neutral process. Pp. 107-138. En: Burslem, D.F.R.P., M.A. Pinard & S.E. Hartley (eds.). Biotic Interactions in the Tropics. Cambridge University Press, Cambridge.
R Core Team. 2015. R: A language and environment for statistical computing. R Foundation for Statistical Computing, Vienna, Austria. URL http://www.R-project.org/. [ Links ]
Ricklef s, R.E. 2004. A comprehensive frame work for global patterns in biodiversity. Ecology Letters 7(1): 1-15. [ Links ]
Rodrigues, A.S. & K.J. Gaston. 2002. Maximising phylogenetic diversity in the selection of networks of conservation areas. BiologicalConservation 105(1): 103-111.
Ter Steege, H., N. Pitman, D. Sabatier, H. Castellanos, P. Van Der Hout, D.C. Daly, M. Silveira, O. Phillips, R. Vasquez, T. Van Andel, J. Duivenvoorden, A. Adalardo De Oliveira, R. Ek, R. Lilwah, R. Thomas, J. Van Essen, C. Baider, P. Maas, S. Mori, J. Terborgh, P. Núñez Vargas, H. Mogollón & W. Morawetz.. 2003. A spatial model of tree a-diversity and tree density for the Amazon. Biodiversity & Conservation 12(11): 2255-2277.
Vamosi, S.M., S.B. Heard, J.C. Vamosi & C.O. Webb. 2009. Emerging patterns in the comparative analysis of phylogenetic communitystructure. Molecular Ecology 18(4): 572-592.
Webb, C.O., D.D. Ackerly & S.W. Kembel. 2008. Phylocom: software for the analysis of phylogenetic community structure and trait evolution. Bioinformatics 24(18): 2098-2100.
Webb, C.O., D.D. Ackerly, M.A. McPeek & M.J. Donoghue. 2002. Phylogenies and community ecology. Annual Review of Ecology and Systematics 33(1): 475-505.
Weigelt, P., W. Daniel Kissling, Y. Kisel, S.A. Fritz, D.N. Karger, M. Kessler, S. Lehtonen, J.-C. Svenning & H. Kreft. 2015. Global patterns and drivers of phylogenetic structure in island floras. Scientific Reports 5:12213.
Whittaker, R.H. 1972. Evolution and measurement of species diversity. Taxon 21: 213-251. [ Links ]
Artículo recibido en: 31 de marzo de 2016
Manejado por: Mónica Moraes R.
Aceptado en: 28 de abril de 2016












 uBio
uBio 

