Servicios Personalizados
Revista
Articulo
Indicadores
Links relacionados
Compartir
Ecología en Bolivia
versión impresa ISSN 1605-2528versión On-line ISSN 2075-5023
Ecología en Bolivia v.41 n.1 La Paz jul. 2006
Artículo
Comparación de modelos de distribución de especies para predecir la distribución potencial de vida silvestre en Bolivia
A comparison of species distribution models to predict wildlife's potential distribution in Bolivia
Kazuya Naoki1*, M. Isabel Gómez2, 3, Ramiro P. López4, Rosa I. Meneses2,4 & Julieta Vargas2
1Instituto de Ecología, Universidad Mayor de San Andrés, Casilla 6394, Correo Central, La Paz, Bolivia. e-mail: knaoki@entelnet.bo, autor de correspondencia
2Museo Nacional de Historia Natural, Casilla 8706, La Paz, Bolivia.
3Asociación Armonía, Casilla 3566, Santa Cruz de la Sierra, Bolivia. 4Herbario Nacional de Bolivia, Casilla 10077, La Paz, Bolivia.
Resumen
Los modelos de distribución de especies basados en un sistema de información geográfica son herramientas útiles para predecir la distribución potencial de las especies y ayudar a tomar decisiones tentativas en la conservación. Actualmente existen varios modelos que tienden a predecir distribuciones significativamente distintas. El objetivo de este estudio es comparar la eficiencia y exactitud de cuatro modelos comúnmente utilizados por biólogos (BIOCLIM, DOMAIN, GARP y regresión logística) en la predicción de la distribución potencial de tres especies de vida silvestre en Bolivia. El modelo DOMAIN, que utiliza el índice de similitud, mostró el mejor rendimiento. La predicción tiende a ser mejor para las especies especialistas de hábitat que para las generalistas.
Palabras clave: Modelos de distribución de especies, sistema de información geográfica, BIOCLIM - DOMAIN - GARP - regresión logística, Bolivia
Abstract
Species distribution models based on geographical information system are powerful tools to predict the potential distribution of species and to help in taking tentative decisions in conservation. Currently several models are available for biologists; however, these models tend to predict significantly different distributions. The objective of this study is to compare the efficiency and accuracy of four commonly used species distribution models (BIOCLIM, DOMAIN, GARP, and logistic regression) to predict the distribution of three wildlife species in Bolivia. The model DOMAIN that uses a similarity index showed the best performance. The prediction tends to be better for habitat specialist species than to generalists.
Key words: Species distribution models, geographical information system, BIOCLIM - DOMAIN - GARP - logistic regression, Bolivia
Introducción
La búsqueda de los factores que determinan la distribución de los organismos ha sido una de las metas centrales de la ecología. Se conoce que varios factores abióticos y bióticos, tales como precipitación, temperatura, evapotranspiración, competencia y depredación interactúan y limitan la distribución de cada especie (Krebs 2001, Molles 2002). En las últimas tres décadas, el estudio de la distribución ha diversificado su enfoque desde de preguntas netamente científicas a las más prácticas para la conservación. Este cambio es una de las respuestas de la comunidad científica a la acelerada degradación de los hábitats naturales y a la extinción de las especies (Groom et al. 2006). Actualmente se considera que la mayor parte de la biodiversidad en el mundo se encuentra en los países tropicales y es en esta zona, donde la tasa de pérdida de biodiversidad ha aumentado a un nivel preocupante en los últimos cincuenta años (Primack et al. 2001). Por tanto, una de las prioridades en estos países es el establecimiento y manejo adecuado de las zonas protegidas para la conservación de la megabiodiversidad (Olson & Dinerstein 1998, Myers et al. 2000, Sechrest et al. 2002). Sin embargo, se conoce poco sobre la distribución de la mayoría de las especies en los trópicos y esta falta de información es un dilema para quienes tienen que tomar decisiones sobre la conservación y el manejo de la vida silvestre en estos países (da Fonseca et al. 2000). Uno de los métodos para resolver este dilema es utilizar modelos de distribución de especies (SDMs en inglés) y los sistemas de información geográfica. Un modelo de distribución de especies establece una distribución potencial de la especie, la cual ayuda a tomar una decisión de conservación tentativa mientras se realice un estudio más intensivo y de largo plazo; también ayuda a dirigir el esfuerzo de la investigación a un área definida. En los últimos 15 años, se han desarrollado numerosos SDMs y su uso en el campo de la biología ha aumentado drásticamente por la mejor disponibilidad de computadoras más eficaces y el más fácil acceso a informaciones espaciales (Scott et al. 2002).
Estos modelos de distribución de especies utilizan dos tipos de información (datos primarios y secundarios) para predecir la distribución potencial de la especie o el tipo de vegetación. Los datos primarios son los sitios de colecta u observación de la especie de interés, los cuales se podrían obtener de las bases de datos de colecciones científicas o publicaciones. Los datos secundarios son la información de clima, topografía o medio ambiente obtenidos de sensores remotos, tales como NDVI (índice de vegetación de diferencia estandarizada) del área donde se quiere predecir la distribución de la especie de interés. Existen varios SDMs que utilizan diferentes algoritmos, tipo de datos primarios (localidades de presencia y/o ausencia) y tipo de datos secundarios. Estos modelos pueden agruparse en cuatro grupos principales, según el algoritmo que se adopte. El algoritmo de “Sobre” utiliza un juego de datos secundarios de cada sitio para derivar un perfil de hábitat adecuado en base a los límites observados para cada condición secundaria (Busby 1991). Los modelos, como BIOCLIM (Busby 1991) y Biomapper (Hirzel 2004), utilizan el algoritmo de “Sobre”. Este algoritmo tiende a sobrepredecir la distribución, asignando localidades donde la combinación de las condiciones son demasiado severas para la especie, aunque cada condición en sí está dentro del rango de distribución (Carpenter et al. 1993). Para evitar este problema, el segundo grupo de modelos - tales como DOMAIN (Carpenter et al. 1993) y BIOM (Nowicki et al. 2004) - utilizan el índice de similitud de las condiciones entre localidades donde la presencia de la especie es conocida y sitios cercanos. El tercer grupo utiliza un modelo estadístico, uno linear que puede ser la regresión logística (LOGIT) o sus derivados (p.e., Cumming 2000, Fleishman et al. 2003). El cuarto grupo utiliza más de un algoritmo; en este grupo se encuentra GARP que utiliza la combinación de cuatro algoritmos: atómico, rango, rango negativo y regresión logística (Stockwell & Noble 1991).
Uno de los problemas es que estos SDMs tienden a predecir distribuciones significativamente diferentes, tanto del área total como de la forma de la distribución de la misma especie (Carpenter et al. 1993, Loiselle et al. 2003). Esta diferencia podría tener una consecuencia grave en la interpretación de la ecología de cada especie y en el plan de conservación (Loiselle et al. 2003). Desafortunadamente, no existe un consenso de cuál SDM tiene el mejor desempeño (B. Loiselle, com. pers. 2004). En Bolivia, varios algoritmos han sido utilizados para predecir la distribución de la vida silvestre o el tipo de vegetación (Mueller et al. 2002, 2003, Sommer et al. 2003, Fernández 2004, Müller et al. 2004, Zambrana 2004). Sin embargo, ningún estudio utilizó ni comparó más de un SDM. Por lo tanto, es una necesidad urgente establecer un procedimiento adecuado para predecir la distribución de especies mediante los SDMs y mejorar los planes y estrategias de la conservación de la vida silvestre de Bolivia. El objetivo de este estudio es comparar la eficiencia y exactitud de los cuatro SDMs más utilizados para predecir la distribución de la vida silvestre en Bolivia. Para eso, se utilizó un SDM para cada algoritmo: BIOCLIM, DOMAIN, regresión logística (LOGIT) y GARP. Para tener la generalidad del estudio y minimizar el sesgo de los resultados, se escogieron tres especies que pertenecen a diferentes grupos taxonómicos y presentan una historia natural y patrón de distribución muy distintos.
Métodos
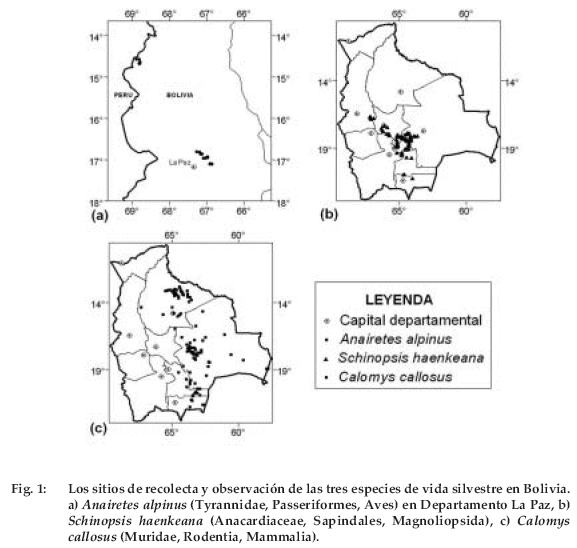
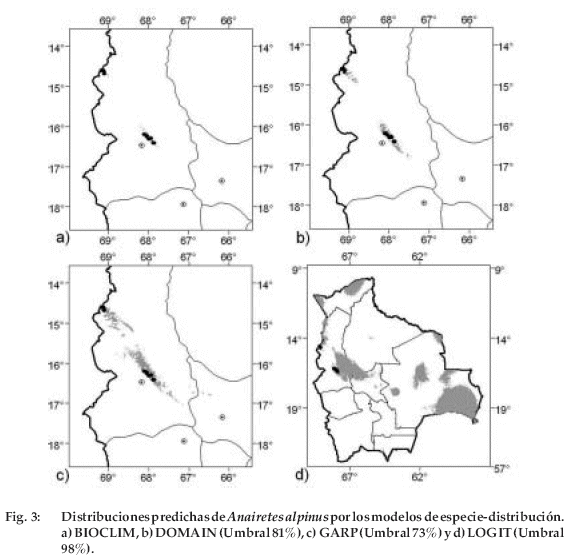
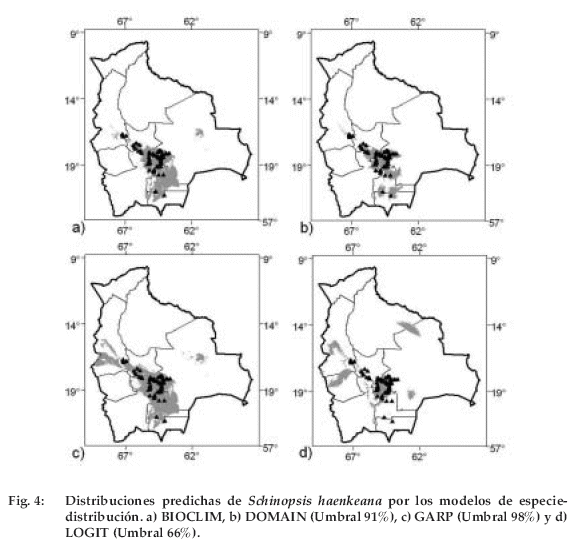
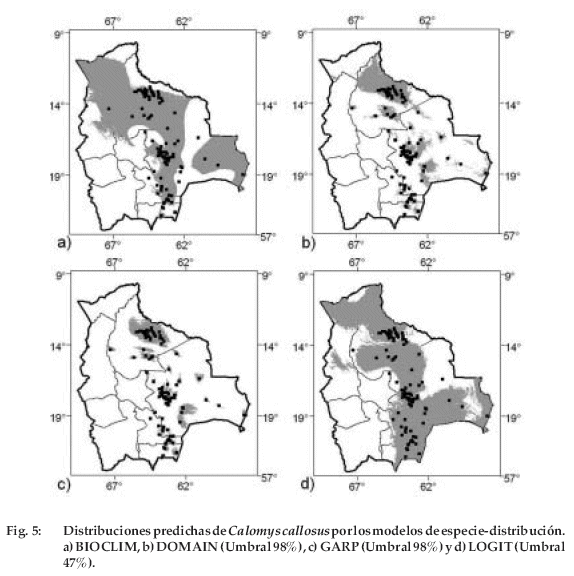
Según la disponibilidad de datos y diversidad del patrón de distribución fueron escogidas tres especies para el estudio. Anairetes alpinus (Tyrannidae, Passeriformes, Aves) es una especie pequeña que habita en los bosques semihúmedos de Polylepis en el sur de Perú y noroeste de Bolivia (Fjeldsa & Krabbe 1990, Fitzpatrick 2004). En Bolivia, su distribución está limitada a fragmentos de bosque de Polylepis pepei en la Cordillera Real en el Depto. La Paz (Figura 1a, M. I. Gómez in prep.). Siendo una especie estrechamente asociada a Polylepis, en un hábitat que está disminuyendo, actualmente se encuentra en la categoría amenazada de la UICN (2004). Schinopsis haenkeana (Anacardiaceae, Sapindales, Magnoliopsida) es un árbol común en regiones secas de los Andes de Bolivia y Argentina entre 1.000 y 2.300 m y frecuentemente se usa como un indicador de la vegetación de los bosques secos interandinos (Ibisch & Mérida 2003) o del Chaco serrano seco (Cabrera 1976, Novara 1985, López 2003a, b). Su distribución se extiende del sur de departamento de La Paz hasta Tarija (Figura 1b). Es un especialista de hábitat con distribución moderadamente limitada y crece en lugares con precipitaciones al menos de 400 mm (Morello 1958) y que no exceden normalmente los 700 mm anuales. Calomys callosus (Muridae, Rodentia, Mammalia) es un roedor común con una distribución regional desde el este y sudoeste del Brasil, Bolivia, Paraguay y norte de Argentina, siendo común en lugares abiertos. En Bolivia se distribuye ampliamente desde tierras bajas hasta 2.050 m en Bolivia (Figura 1c, Anderson 1997). Existe reducida información sobre su hábitat, hábito, comportamiento o alimentación (Anderson 1997); sin embargo, su amplia distribución indica que es una especie generalista de hábitat. Los datos primarios, localidades y coordenadas de A. alpinus fueron obtenidos mediante observaciones de campo y dos publicaciones (Fjeldsa & Kessler 1996, Vogel & Hennessey 2002). Los datos de S. haenkeana fueron obtenidos de las bases de datos del Herbario Nacional de Bolivia (LPB), Tropicos del Missouri Botanical Garden (MO) y observaciones de campo. Los datos de C. callosus fueron obtenidos de Anderson (1997).
Diecinueve tipos de datos bioclimáticos fueron obtenidos de la base de datos de WORLDCLIM (http://biogeo.berkeley.edu/worldclim/worldclim.html). El análisis de correlación de Pearson fue realizado para encontrar los datos climáticos altamente correlacionados (r > 0.9). Para reducir la redundancia de datos y acortar el tiempo de análisis, se eliminaron los datos climáticos redundantes. Ocho datos bioclimáticos poco correlacionados fueron utilizados como entre los secundarios para la predicción de la distribución potencial: temperatura media anual, rango de temperatura diurna, isotermalidad, estacionalidad de temperatura, precipitación anual, precipitación del mes más seco, estacionalidad de la precipitación y la precipitación de los cuatro meses más cálidos,.
Para evaluar el desempeño de cada SDM, se utilizaron cuatro índices: sensitividad, especificidad, poder predictivo positivo (PPP) y Kappa. Estos cuatro índices de prueba fueron calculados usando las siguientes ecuaciones:
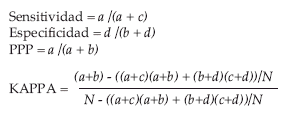
Donde, a es el número de registros presentes correctamente predichos como presentes, b es el número de registros ausentes incorrectamente predichos como presentes, c es el número de registros presentes incorrectamente predichos como ausentes, d es el número de registros ausentes correctamente predichos como ausentes y N es el número total de observaciones = a + b + c + d.
La sensitividad es la proporción de presencias correctamente predichas y su valor alto indica un bajo error de omisión (error de tipo I). La especificidad es la proporción de ausencias correctamente predichas y su valor alto indica un bajo error de comisión (error de tipo II). El PPP es la proporción de presencias correctamente predichas con relación a todas las localidades, donde la presencia de la especie fue predicha (Parra et al. 2004). Kappa es un índice de desempeño global del modelo y está definido como la precisión de la predicción en relación a la predicción al azar. El valor alto de Kappa indica que la predicción tiene ambos errores bajos de omisión y comisión (Fielding & Bell 1997). Estos cuatro índices varían entre 0 y 1; cuanto más cerca de 1 esté el valor, significa un mejor desempeño del modelo.
Para calcular estos índices, se realizó un remuestreo de datos. Para eso, se escogieron aleatoriamente aproximadamente el 70% de las localidades de colecta u observación de cada especie. Este 70% de localidades fue utilizado para generar una distribución de prueba. El 30% restante de los registros fue usado para comprobar la precisión de la predicción de la presencia. Para comprobar la precisión de la predicción de la ausencia, se utilizaron 300 puntos de pseudoausencia generados aleatoriamente dentro de Bolivia. Este proceso de remuestreo fue repetido 10 veces para cada modelo y cada especie.
Los tres modelos de distribución, DOMAIN, GARP y LOGIT son modelos probabilísticos y la extensión de la distribución predicha y los valores de índices dependen del umbral (corte de la probabilidad) que se aplica. Para determinar el mejor umbral para cada predicción, se escogió la probabilidad en que Kappa alcanza el máximo valor (máx-Kappa).
Los valores de los cuatro índices varían entre 0 y 1; por tanto, tienden a seguir la distribución binomial (Sokal & Rohlf 1995). Para normalizar los datos, todos los valores de los índices fueron transformados a arcoseno de raíz cuadrada antes de realizar los análisis estadísticos. Para comparar los valores de índices, se aplicó una ANOVA bifactorial considerando los modelos de distribución y especies como los factores. Cuando se encontró una diferencia significativa en uno de estos factores, se aplicó una prueba a posteriorí de Tukey para realizar las comparaciones pareadas.
Resultados
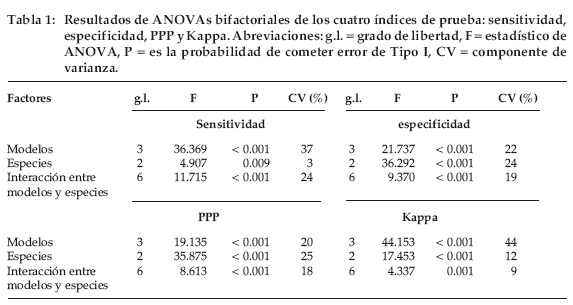
Se observaron diferencias significativas en los cuatro índices de prueba entre los cuatro modelos de distribución (P < 0.001 para todos los índices, Tabla 1) y entre las tres especies (P < 0.01 para todos, Tabla 1). Se encontraron interacciones significativas entre los modelos y las especies en los cuatro índices de prueba (P < 0.01 para todos, Tabla 1).
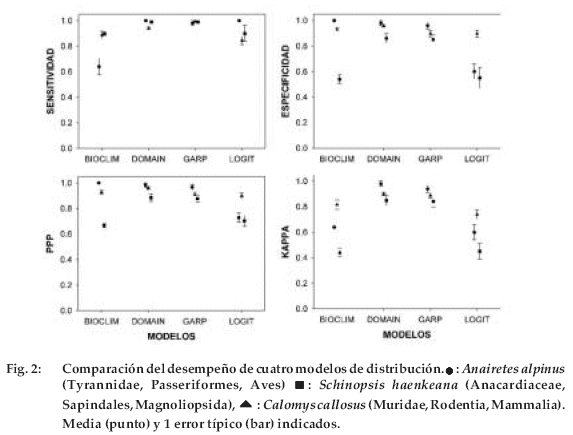
En relación con la sensitividad, GARP mostró significativamente el mejor rendimiento que LOGIT y BIOCLIM (P < 0.001). No se observó diferencia significativa entre GARP y DOMAIN (P = 0.6). BIOCLIM mostró el peor rendimiento que los otros tres modelos (P < 0.001) (Figura 2). El rendimiento de C. callosus es significativamente mayor que S. haenkeana (P < 0.01), pero no mayor que A. alpinus (P = 0.6). En la especificidad, DOMAIN mostró significativamente el mejor rendimiento que LOGIT y BIOCLIM (P < 0.05). No se observó una diferencia significativa entre DOMAIN y GARP (P = 0.6). LOGIT mostró el peor rendimiento que los otros tres modelos (P < 0.001) (Fig. 2).
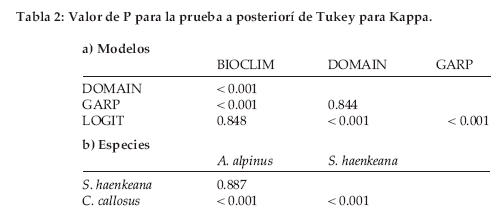
El rendimiento de S. haenkeana y A. alpinus es significativamente mayor que C. callosus (P < 0.001). En PPP, DOMAIN mostró significativamente el mejor rendimiento que LOGIT y BIOCLIM (P < 0.05). No se observó una diferencia significativa entre DOMAIN y GARP (P = 0.5). LOGIT mostró el peor rendimiento que los otros tres modelos (P < 0.001) (Figura 2). El rendimiento de S. haenkeana y A. alpinus es significativamente mayor que C. callosus (P < 0.001). En Kappa, los modelos DOMAIN y GARP fueron mejores que BIOCLIM y LOGIT (P < 0.001) (Figura 2, Tabla 2a). El rendimiento de S. haenkeana y A. alpinus es significativamente mayor que C. callosus (P < 0.001, Tabla 2b).
DOMAIN, GARP y BIOCLIM predijeron una distribución parecida en tamaño y forma para A. alpinus y S. haenkeana (Figuras 3 y 4). En cambio, LOGIT predijo una distribución más fragmentada que contiene varias zonas donde no hay ningún registro de estas especies y las condiciones no son las apropiadas para su presencia (Figuras 3 y 4).
Para C. callosus, DOMAIN y GARP predijeron una distribución más limitada, en general alrededor de los sitios de registro, al contrario que BIOCLIM y LOGIT que predijeron un área más extensa (Figura 5).
Discusión
Los análisis de este estudio mostraron que los cuatro SDMs predicen distribuciones significativamente diferentes como se ha encontrado en otros estudios (Carpenter et al. 1993, Loiselle et al. 2003). Los máximos valores de Kappa fueron observados en DOMAIN en todas las especies, seguido por GARP, lo cual indica que ambos modelos predicen la distribución de las especies mejor que BIOCLIM y LOGIT, manteniendo los errores de tipo I y II relativamente bajos. Aunque en general existe una relación negativa entre sensitividad y especificidad (p.e. Parra et al. 2004), DOMAIN y GARP mostraron el mejor rendimiento en ambos índices, comparado con BIOCLIM y LOGIT. La sobrepredicción de la distribución en BIOCLIM versus con DOMAIN fue observada en este estudio como en el de Carpenter (1993), indicando este problema general de BIOCLIM . La regresión logística (LOGIT) muestra el peor rendimiento entre los cuatro modelos probados; además con frecuencia predice áreas muy alejadas de las zonas conocidas de cada especie con condiciones climáticas muy distintas. Podría haber varias razones por las cuales este modelo genera errores tan grandes. Sin embargo, nos parece que uno de los problemas más graves es la suposición de LOGIT en que los organismos reaccionan linealmente a las condiciones medioambientales, prefiriendo las condiciones extremas. En general, la mayoría de los organismos se adaptan a las condiciones intermedias y evitan las condiciones ambientales extremas (Krebs 2001) y probablemente pocas especies cumplen con la suposición del modelo de regresión logística.
Aunque el número de especies evaluadas es reducido en este estudio, la predicción de las especies especialistas de hábitat fue mejor que la de la especie generalista en todos los índices, excepto en la sensitividad, que fue mejor para la especie generalista. La distribución del hábitat y vegetación está principalmente determinada por las condiciones abióticas en el nivel regional (p.e. Holdridge 1967, temperatura promedia anual, precipitación anual y evapotranspiración). Por tanto, la distribución de las especies especialistas de cierta vegetación y/o hábitat también puede predecirse en base a las mismas condiciones abióticas. Al contrario, las especies generalistas de hábitat, de hábitats con menor influencia climática y/o de amplia distribución podrían cruzar varias condiciones climáticas sin recibir una influencia fuerte por parte de éstas. Por ejemplo, las especies de amplia distribución, como Zonotrichia capensis (Aves, Emberizidae) pueden tolerar condiciones climáticas distintas, mientras exista un hábitat adecuado (zona perturbada antrópicamente) (Ridgely & Greenfield 2001). Otras especies ' como los flamencos (Aves, Phoenicopteridae, Phoenicopterus sp., Phoenicoparrus spp.) - requieren hábitats muy específicos (lagunas bajas de gran extensión en el Altiplano), que no están estrechamente asociadas a la condición climática (del Hoyo et al. 1992). Para predecir la distribución de los flamencos sería necesario considerar factores adicionales, como NDVI, distancia al cuerpo de agua o parámetros topográficos, además de los factores climáticos utilizados en este estudio.
El bajo desempeño de la predicción de la distribución de las especies con amplia distribución también podría ser un artefacto de los índices de prueba utilizados en este estudio. El valor más bajo de Kappa en una especie de amplia distribución como C. callosus podría provenir del bajo valor de especificidad. Como los puntos de ausencia usados en este estudio son de pseudoausencia (puntos escogidos aleatoriamente), la especificidad está inversamente correlacionada con el área predicha (Anderson et al. 2003). Bajo esta metodología, la especie que posee una distribución amplia tiende a tener un valor bajo de especificidad.
Actualmente la mayoría de los modelos utilizan información principalmente de condiciones ambientales y algunos utilizan también información adicional, como uso de tierra y cobertura vegetal (Peterson & Robins 2003, Parra et al. 2004). Estos modelos ignoran la importancia de los procesos biológicos tales como competencia, depredación y mutualismo. En otras palabras, la distribución de otras especies puede afectar la distribución de la especie de interés. Desafortunadamente, la influencia de tales interacciones en la distribución de la mayoría de las especies en Bolivia es desconocida. Sin embargo, mientras aumenten los estudios sobre los procesos biológicos, será posible incorporar a las distribuciones o densidad de estos procesos biológicos en el modelo.
Este estudio utilizó la resolución de distribución de 30 segundos (ca. 1 x 1 km). Aunque esta resolución es más fina que la usada en otros estudios realizados anteriormente en Bolivia (Mueller et al. 2002, Sommer et al. 2003), la mayoría de los organismos probablemente utiliza solo una parte de esta área de 1 x 1 km. Esto se debe a la heterogeneidad del medio ambiente a escala más fina. Las especies utilizan solo ciertos hábitats o microhábitats que existen a manera de mosaico dentro de la grilla de 1 x 1 km que se considera homogénea climáticamente. Es posible generar otro tipo de datos a escala más fina, como datos topográficos (altitud, pendiente, exposición, distancia a cuerpos de agua), utilizando los modelos digitales de elevación (DEM), índices de vegetación y uso de tierra en base a imágenes satelitales. La incorporación de estos datos puede producir la predicción de distribución más realista y a mayor escala (M. I. Gómez et al. datos no publicados).
La predicción de la distribución de las especies en Bolivia está en una etapa inicial y se requieren más estudios para establecer un procedimiento adecuado. En el futuro, también se deberían considerar otros factores, tales como tipos de datos (número de datos , datos adicionales como los topográficos y los de sensores remotos), el valor de los parámetros (en GARP, el número de corridas, valor de convergencia y número de iteraciones), los índices del desempeño del modelo (número de remuestreo, porcentaje de datos para cada remuestreo), escala de análisis (1 km2, mayor o menor) e inclusión de los procesos bióticos (competencia, depredación, mutualismo).
En resumen, para predecir la distribución de las especies en Bolivia, DOMAIN y GARP poseen los mejores rendimientos globales. La predicción de la distribución de una especie generalista de hábitat con una distribución amplia tiende a ser menos precisa, posiblemente por la falta de correlación de su distribución con las condiciones climáticas. Al contrario, la distribución de una especie especialista de hábitat o vegetación tiende a ser más precisa por su requerimiento de condiciones abióticas especificas. La incorporación de otros tipos de datos, tales como los topográficos y los de imágenes satelitales podría aumentar la precisión de su predicción y mejorar la resolución de la distribución.
Agradecimientos
Queremos agradecer al Herbario Nacional de Bolivia en acceder a su base de datos. El manuscrito fue mejorado por los comentarios de dos revisores anónimos. El trabajo del campo de Ramiro P. López y Rosa I. Meneses fue financiado por Conservation International y el de M. Isabel Gómez fue financiado por British Petroleum Conservation Programme, Blake Foundation y Royal Geographical Society.
Referencias
Anderson, R. P., D. Lew & A. T. Peterson. 2003. Using error distributions to select best subsets of predictive models of species´ distributions. Ecological Modelling 162: 211-232.
Anderson, S. 1997. Mammals of Bolivia, taxonomy and distribution. Bulletin of the American Museum of Natural History 231: 1-652.
[ Links ]Busby, J. R. 1991. BIOCLIM - A bioclimate analysis and prediction system. Pp. 64-68. En: C. R. Margules y M. P. Austin (eds.). Nature Conservation: Cost Effective Biological Surveys and Data Analysis. CSIRO, Melbourne.
[ Links ]Cabrera, A. L. 1976. Regiones fitogeográficas argentinas. Pp. 1-85. En: W. F. Kugler (ed.). Enciclopedia Argentina de agricultura y jardinería. ACME, Buenos Aires.
[ Links ]Carpenter, G., A. N. Gillison & J. Winter. 1993. DOMAIN: a flexible modeling procedure for mapping potential distributions of plants and animals. Biodiversity and Conservation 2: 667-680.
Cumming, G. S. 2000. Using between-model comparisons to fine-tune linear models of species ranges. Journal of Biogeography 27: 441-455.
[ Links ]da Fonseca, G. A. B., A. Balmford, C. Bibby, L. Boitani, F. Corsi, T. M. Brooks, C. Gascon, S. Olivieri, R. A. Mittermeier, N. Burgess, E. Dinerstein, D. M. Olson, L. Hannah, J. Lovett, D. Moyer, C. Rahbek, S. N. Stuart & P. Williams. 2000. Following Africa's lead in setting priorities. Nature 405: 393-394.
del Hoyo, J., A. Elliott & J. Sargatal (eds.). 1992. Handbook of the birds of the world. Vol. 1: Ostrich to Ducks. Lynx Edicions, Barcelona.
[ Links ]Fernández, T. M. 2004. Verificando un modelo predictivo de distribución de anfibios para Bolivia. Tesis de Licenciatura en Biología, Universidad Mayor de San Andrés, La Paz.
[ Links ]Fielding, A. H. & J. F. Bell. 1997. A review of methods for the assessment of prediction errors in conservation presence/absence models. Environmental Conservation 24: 38-49.
Fitzpatrick, J. W. 2004. Family Tyrannidae (Tyrant-Flycatchers). Pp. 170-463. En: J. del Hoyo, A. Elliott & D. A. Christie (eds.). Handbook of the Birds of the World. Lynx Edicions, Barcelona.
[ Links ]Fjeldsa, J. & M. Kessler. 1996. Conserving the biological diversity of Polylepis woodlands of the highland of Peru and Bolivia. NORDECO, Copenhagen.
[ Links ]Fjeldsa, J. & N. Krabbe. 1990. Birds of the high Andes. Apollo Books, Svendborg.
[ Links ]Fleishman, E., R. MacNally & J. P. Fay. 2003. Validation tests of predictive models of butterfly occurrence based on environmental variables. Conservation Biology 17: 806-817.
Groom, M. J., G. K. Meffe & C. R. Carroll. 2006. Principles of conservation biology, 3rd ed. Sinauer Associates, Inc., Sunderland, Massachusetts.
[ Links ]Hirzel, A. 2004. BioMapper 3, User's manual (http://www.unil.ch/biomapper).
[ Links ]Holdridge, L. R. 1967. Life zone ecology. Tropical Science Center, San José.
[ Links ]Ibisch, P. L. & G. Mérida (eds.). 2003. Biodiversidad: la riqueza de Bolivia: estado de conocimiento y conservación. Editorial FAN, Santa Cruz de la Sierra.
[ Links ]IUCN. 2004. IUCN red list of threatended species (http://www.redlist.org).
[ Links ]Krebs, C. J. 2001. Ecology: the experimental analysis of distribution and abundance, 5th ed. Benjamin/Cummings, San Francisco.
[ Links ]Loiselle, B. A., C. A. Howell, C. H. Graham, J. M. Goerck, T. Brooks, K. G. Smith & P.H. Williams. 2003. Avoiding pitfalls of using species distribution models in conservation planning. Conservation Biology 17: 1591-1600.
López, R. P. 2003a. Diversidad florística y endemismo de los valles secos bolivianos. Ecología en Bolivia 38: 27-60.
[ Links ]López, R. P. 2003b. Phytogeographical relations of the Andean dry valleys of Bolivia. Journal of Biogeography 30: 1659-1668.
[ Links ]Molles, M. C., Jr. 2002. Ecology: concepts and applications, 2nd ed. McGraw-Hill Companies, Inc., Boston.
[ Links ]Morello, J. 1958. La provincia fitogeográfica del Monte. Opera Lilloana 2: 1-155.
[ Links ]Mueller, R., S. G. Beck & R. Lara. 2002. Vegetación potencial de los bosques de Yungas en Bolivia, basado en datos climáticos. Ecología en Bolivia 37: 5-14.
Müller, R., A. Briancon, I. Hinojosa & P. Ergueta. 2004. Prioridades de conservación en los Yungas Bolivianos. Trópico, La Paz.
[ Links ]Müller, R., C. Nowicki, W. Barthlott & P. L. Ibisch. 2003. Biodiversity and endemism mapping as a tool for regional conservation planning - case study of the Pleurothallidinae (Orchidaceae) of the Andean rain forests in Bolivia. Biodiversity and Conservation 12: 20052024.
Myers, N., R. A. Mittermeier, C. G. Mittermeier, G. A. B. da Fonseca & J. Kent. 2000. Biodiversity hotspots for conservation priorities. Nature 403: 853-858.
Novara, L. J. 1985. Esquema florístico de la ciudad de Salta. Pp. 11-29. En: J. L. Novara (ed.). Guía de viaje de las XX Jornadas Argentinas de Botánica. Sociedad Argentina de Botánica, Salta.
[ Links ]Nowicki, C., A. Ley, R. Caballero, J. H. Sommer, W. Barthlott & P. L. Ibisch. 2004. Extrapolating distribution ranges BIOM 1.1., a computerized bio-climatic model for the extrapolation of species ranges and diversity patterns. Pp. 36-68. En: C. Vásquez & P. L. Ibisch (eds.). Orchids of Bolivia: Diversity and Conservation Status. Vol. 2. Laeliinae, Polystachinae, Sobraliinae with update and complementation of the Pleurothallidinae. Editorial F.A.N., Santa Cruz de la Sierra.
[ Links ]Olson, D. M. & E. Dinerstein. 1998. The global 200: a representation approach to conserving the earth's most biologically valuable ecoregions. Conservation Biology 12: 502-515.
Parra, J. L., C. C. Graham & J. F. Freile. 2004. Evaluating alternative data sets for ecological niche models of birds in the Andes. Ecography 27: 350-360.
Peterson, A. T. & C. R. Robins. 2003. Using ecological-niche modeling to predict Barred Owl invasions with implications for Spotted Owl conservation. Conservation Biology 17: 1161-1165.
Primack, R., R. Rozzi, P. Feinsinger, R. Dirzo & F. Massardo. 2001. Fundamentos de conservación biológica: perspectivas latinoamericanas. Fondo de Cultura Económica, México, D.F.
[ Links ]Ridgely, R. S. & P. J. Greenfield. 2001. The birds of Ecuador. Cornell University Press, Ithaca, Nueva York.
[ Links ]Scott, J. M., P. J. Heglund, M. L. Morrison, J. B. Haufler, M. G. Raphael, W. A. Wall & F. B. Samson (eds.). 2002. Predicting species occurrences: issues of accuracy and scale. Island Press, Washington, D.C.
[ Links ]Sechrest, W., T. M. Brooks, G. A. B. da Fonseca, W. R. Konstant, C. G. Mittermeier, A. Purvis, A. B. Rylands & J. L. Gittleman. 2002. Hotspots and the conservation of evolutionary history. Proceedings of the National Academy of Sciences of the United States of America 99: 2067-2071.
[ Links ]Sokal, R. R. & F. J. Rohlf. 1995. Biometry. 3rd ed. W. H. Freeman and Company, Nueva York.
[ Links ]Sommer, J. H., C. Nowicki, L. Rios, W. Barthlott & P. L. Ibisch. 2003. Extrapolating species ranges and biodiversity in data-poor countries: The computerized model BIOM. Revista de la Sociedad Boliviana de Botánica 4: 171-190.
Stockwell, D. R. B. & I. R. Noble. 1991. Induction of sets of rules from animal distribution data: a robust and informative method of data analysis. Mathematics and Computers in Simulation 32: 249-254.
Vogel, C. & A. B. Hennessey. 2002. Discovery of a new site for Ash-breasted Tit-tyrant Anairetes alpinus in Bolivia. Cotinga 17: 80.
Zambrana, C. M. 2004. Distribución y diversidad potencial de la avifauna en un complejo de áreas protegidas del noroeste de La Paz: implicaciones para la conservación. Tesis de Licenciatura en Biología. Universidad Mayor de San Andrés, La Paz.
[ Links ]
Artículo recibido en: Mayo de 2005.
Manejado por: Mónica Moraes
Aceptado en: Junio de 2006.












 uBio
uBio