Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista de la Sociedad Boliviana de Pediatría
On-line version ISSN 1024-0675
Rev. bol. ped. vol.43 no.3 La Paz Aug. 2004
ARTICULO ORIGINAL
Caracterización geno-fenotípica de aislados de Escherichia coli AEEC de pacientes pediátricos con procesos diarreicos infecciosos en la ciudad de La Paz: implicancias para el diagnóstico y epidemiología de las enfermedades diarreicas agudas
Geno-Phenotypic characterization of aeec Escherichia coli isolated from children with infectious diarrheal diseases in La Paz: relevancy for the diagnosis and epidemiology of acute diarrheal diseases
Dr: Samanta Sánchez*, Paola Ropmecin**, Luis Miguel Guachalla*** y Volga Iñiguez****
Instituto de Biología Molecular y Biotecnología, Unidad de Biología Molecular de Enteropatógenos, Carrera de Biología. Facultad de Ciencias Puras y Naturales. Universidad Mayor de San Andrés
* Lic. Bioquímica
** Lic. Biología
*** Lic. Bioquímica
**** Ph D. Biología
* Autor correspondiente. Dirección: Instituto de Biología Molecular y Biotecnología, Campus Universitario de Cota-Cota. La Paz, Bolivia. E. mail: volgavir@hotmail.com
Artículo recibido 1/10/2004 y fue aprobado para publicación 8/12/2004.
Resumen En el presente estudio se realizó la caracterización de Escherichia coli Enteropatogénica (EPEC) y Escherichia coli Enterohemorrágica (EHEC), dos categorías patogénicas de E. Coli, causantes de la lesión de adherencia y esfacelación (EAEE), en muestras de heces diarreicas de niños menores a 5 años. El perfil patogénico de EAEE se realizo mediante el análisis por PCR, de los genes intimina (eae), bundlina (bfpA) y toxinas siga (stx1 y stx2). Estas pruebas, se complementaron con ensayos fenotípicos de la resistencia a antibióticos, fermentación de sorbitol y producción de b-D-glucoronidasa. La prevalencia de EAEE fue del 7% con preponderancia de las cepas EPEC (95%) sobre EHEC. Se encontró una mayor proporción (83%) de cepas EPEC atípicas que típicas. Un alto porcentaje de los aislados de EPEC es resistente a más de 5 antibióticos analizados. La frecuencia de multiresistencia a bloques de 5 y 2 antibióticos sugiere que la resistencia es transmisible por vía horizontal. La correlación entre la pertenencia a un serogrupo particular de EPEC y las características genotípicas, mostró heterogeneidad en el perfil de patogenicidad tanto entre un mismo como entre diferentes serogrupos, demostrando que el diagnostico de DEC mediante serotipificación no es útil en nuestro medio. Los aislados de EHEC, se caracterizan por presentar una marcada susceptibilidad a los antibióticos. Se reporta la presencia de los serogrupos O157 y O6. Este estudio, constituye el primer reporte en nuestro medio sobre la determinación y caracterización geno-fenotípica de EPEC y EHEC por métodos moleculares. En conjunto, los datos obtenidos tienen relevancia para el diagnostico, tratamiento y estudio de la epidemiología de AEEC en las EDA en Bolivia.
Palabras Claves:
Rev Soc Bol Ped 2004; 43 (3): 132-43: Adherencia y Esfacelación (EA), Bundlina (BFP), Intimina (EAE), Toxina Shiga (STX).
Abstract
In this study, enteropathogenic (EPEC) and enterohemorrhagic (EHEC) E. Coli, two E. Coli categories causing attaching and effacing lesions, were isolated and characterized from children with diarrhea less than 5 years of age. The AEEC pathogenic profile was analyzed by PCR for the presence of the intimin (eae), bundle-forming pilus (bfpA) and Shiga toxin (stx, stx2) genes. Phenotypic analysis for the presence of antibiotic multi-resistance, sorbitol fermentation and B-D glucoronidase were also performed. AEEC prevalence was 7%. EPEC accounted for 95% of the isolates of which 83% were atypical. A high percentage of EPEC isolates is resistant to more than 5 antibiotics. The multi-resistance frequency to 5 and 2 antibiotics suggest antibiotic resistance transmission by lateral transfer. The lack of correlations between EPEC serogroups and genotypic strain profile demonstrates that serological DEC diagnosis is not useful for local isolates. EHEC isolates were remarkably susceptible to most of the antibiotics tested. The isolation of 0157 and 06 serogroups is reported. This is the first report of EPEC and EHEC molecular strain characterization. The results described are relevant for EAEE diagnosis, treatment and epidemiology of diarrheal diseases in Bolivia.
Key words:
Rev Soc Bol Ped 2004; 43 (3): 132-43: Attaching and Effacing (EA), Bundlina (BFP), Intimin (EAE), Shiga toxin (STX).
Introducción
A nivel mundial, las Enfermedades Diarreicas Agudas (EDAs), son un problema importante de salud de la población infantil, principalmente en los países en desarrollo donde se producen anualmente entre 4,6 a 6 millones de muertes constituyendo la segunda causa global de mortalidad infantil. Estas estadísticas se reflejan en que se produce un promedio de 3 episodios de diarrea por año en niños menores a 5 años y una tasa global de mortalidad promedio de más de 10.000 niños por día1-3.
En Bolivia, las EDA constituyen una de las principales causas de mortalidad entre niños menores de 5 años de edad, produciéndose aproximadamente quince mil muertes por año4.
La diversidad de enteropatógenos asociada a las EDA y la falta de acceso a tecnologías sensibles de diagnostico, determinan que, similar a otros países en desarrollo, se desconozca la etiología de una gran parte de los episodios diarreicos infecciosos, los cuales se tratan, por lo general sin conocimiento de la patología subyacente.
Es así que la epidemiología de los principales patógenos asociados a la gastroenteritis infantil y los mecanismos de virulencia, son poco conocidos en nuestro medio.
En los últimos años, un número creciente de patógenos bacterianos y virales han sido asociados a las EDA gracias a la progresiva incorporación de nuevas tecnologías de detección, que han permitido establecer la relación causal entre los microorganismos y las EDA, mediante la identificación y caracterización de genes de patogenicidad y virulencia.
Entre las bacterias asociadas a las EDA, Escherichia coli Diarreogénica (DEC) esta comúnmente asociada a formas endémicas e incluye al menos 6 categorías patogénicas bien definidas, las cuales comprenden a E. Coli enteropatogénica (EPEC), E. Coli enterohemorrágica (EHEC), E. Coli enterotoxigénica (ETEC), E. Coli enteroagregativa (EAEC), E. Coli enteroinvasiva (EIEC) y E. Coli adherente difusa (DAEC). Estos "patotipos" se distinguen de los simbiontes de la flora normal, por la presencia de factores de virulencia adquiridos principalmente por transferencia horizontal a partir de plásmidos, fagos y genomas de otras bacterias"5.
EPEC y EHEC son dos categorías patogénicas de E. Coli que causan la lesión del tipo de "adherencia y esfacelación" (A/E) en las células intestinales. Esta lesión se caracteriza por la destrucción de las microvellosidades de la membrana, la adherencia intima de la bacteria al epitelio intestinal, el reordenamiento del citoesqueleto de la célula huésped, la formación de un pedestal y la iniciación de varías señales de transducción generadoras de la secreción intestinal.
EPEC es uno de los patógenos bacterianos más importantes asociado a la gastroenteritis infecciosa y diarrea esporádica en niños menores a dos años6-12. Además de producir la lesión A/E, EPEC presenta generalmente un plásmido denominado EAF (factor de adherencia de EPEC) relacionado tanto a la adherencia entre bacterias, así como a las células epiteliales del intestino13.
EHEC, por su parte es un patógeno emergente de las últimas décadas, que se caracteriza por producir las citotoxinas Shiga causando diarrea no sanguinolenta, colitis hemorrágica y síndrome hemolítico urémico (SHU)14-15.
Con el propósito de contribuir al conocimiento de la etiología bacteriana de las EDA, en el presente trabajo, se utilizo un conjunto de ensayos genotípicos y fenotípicos para identificar, caracterizar y comparar a EPEC y EHEC, en muestras de heces fecales de pacientes pediátricos con Procesos Diarreicos Infecciosos (PDI). Entre las características analizadas se incluyen a genes de patogenicidad, patrones de resistencia a antibióticos y marcadores bioquímicos. Asimismo, se analizo la validez de la asignación de EPEC en base a la pertenencia a sus serogrupos característicos.
Estos datos, constituyen el primer reporte en Bolivia sobre el aislamiento y análisis de las categorías de DEC causantes de la lesión A/E (AEEC) a partir de heces diarreicas de niños menores a 5 años. Los resultados obtenidos remarcan una mayor prevalencía de EPEC atípica sobre EHEC, heterogeneidad geno-fenotípica de cepas EPEC y EHEC, alta proporción de cepas multiresistentes a 7 antibióticos entre aislados de EPEC, en fuerte contraste a EHEC y la discordancia en la clasificación de EPEC mediante serogrupos con relación al perfil patogénico.
La diversidad genotípica encontrada entre las cepas AEEC, circulantes en los PDI tiene relevancia clínica y epidemiológica para el estudio del rol de EPEC y EHEC en las EDA en nuestro medio.
Material y métodos
Cepas control y cepas clínicas. Se utilizaron cepas de referencia de E. Coli Diarreogénicas (DEC) provenientes del Instituto de Genética Evolutiva y Molecular, de la Universidad Estatal de Pennsylvania, Estados Unidos de Norte América.
Asimismo, se analizaron 31 cepas clínicas de E. Coli, que presentan serogrupo definido, las cuales fueron aisladas en La Paz, Bolivia en 1993 a partir de muestras fecales de niños con PDI.
Población de estudio. En el periodo de Febrero del 2001 a Junio del 2002, se recolectaron 882 muestras de heces fecales provenientes de niños menores a 5 años de edad, con PDI en tres centros de salud de la ciudad de La Paz (Hospital Materno Infantil, Hospital Militar y Hospital del Niño Ovidio Aliaga).
Aislamiento y caracterización fenotípica de E. Coli. Las muestras fecales fueron sembradas e incubadas en Agar MacConkey, durante 18 horas a 37° C. Las colonias fermentadoras de lactosa y con morfología característica de E. Coli, fueron luego analizadas por su capacidad de fermentación de sorbitol (FS) y producción de b-D-glucoronidasa (MUG).
Resistencia a antibióticos. Se consideraron para el análisis a los siguientes antibióticos: ampicilina, eritromicina, estrepto-micina, sulfatrimetoprim, ácido nalidixico, cloranfenicol, tetra-ciclina y gentamicina. La sensibilidad a los ocho antibióticos se evaluó mediante el método de difusión de disco acorde a los lineamientos del Comité de Control de Laboratorios Clínicos16.
Caracterización genotípica
Reacción en Cadena de la Polimerasa (PCR). La presencia de los genes eae (región variable y constante), bfp, stx1, stx2 y mdh correspondientes a las proteínas Intimina, Bundlina, Shiga toxina 1, Shiga toxina 2 y Malato-deshidrogenasa respectivamente, se realizo en los aislados de E. Coli, mediante el uso de cebadores específicos (Cuadro # 1).
Los productos de PCR se visualizaron por electroforesis en geles de agarosa al 1% con bromuro de etidio, en un transiluminador de luz UV.
Análisis por PCR-RFLP. Los fragmentos amplificados del gen eae y bfp se incubaron con las enzimas de restricción Dra I y Msp I respectivamente, por dos horas a 37°C. Posteriormente, los productos de digestión fueron separados por electroforesis en geles de agarosa (NuSieve) al 4% con bromuro de etidio y visualizados con un transiluminador de luz UV.
Procesamiento de datos. Los datos fueron analizados por el test exacto de Fisher. La asociación se considero estadísticamente significativa, con un valor de p< 0.05.
Resultados
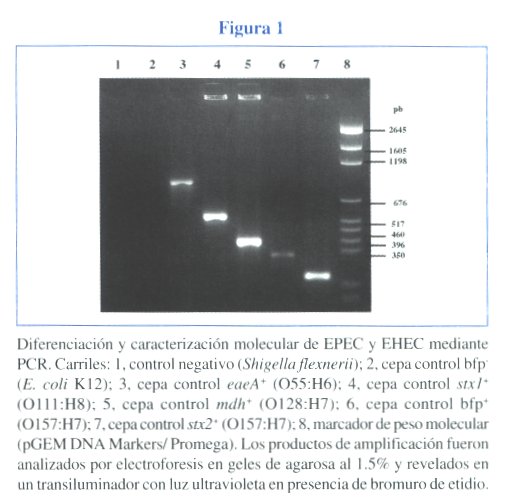
Diferenciación molecular de EPEC y EHEC mediante PCR. En la Figura # 1, se observan los productos amplificados mediante PCR, de regiones de los genes eaeA, stx1, mdh, bfpA y stx2, presentes en cepas de referencia. Los ensayos fueron específicos detectando todos los genes de interés en el 100% de las cepas control de genotipo y fenotipo múltiple (datos no mostrados). El análisis de estos genes en las muestras clínicas permitió la identificación de cepas EPEC y EHEC.
La presencia del gen mdh permitió confirmar que las cepas eran de la especie E. Coli.
EPEC: Identificación y caracterización en muestras clínicas. Del total de 882 muestras clínicas, 58 aislados de E. Coli presentaron genotipo eae+ stx- siendo clasificados como EPEC. Esto represento una prevalencía de infección por EPEC de 6% en el periodo de estudio (Cuadro # 2).
El producto de amplificación del gen eaeA (863pb), fue verificado en el total de cepas eaeA + mediante PCR-RFLP. En el 96% de las mismas, se obtuvo el fragmento esperado de 750pb, correspondiente al extremo 3' de la región amplificada (datos no mostrados).
El gen bfpA, codificante de la bundlina o subunidad estructural del pilus BFP fue detectado en el 17% de las cepas eae+ aisladas en el periodo 2001-2002 representando consiguientemente un total de 10 cepas EPEC típicas y 48 atípicas (Cuadro # 2).
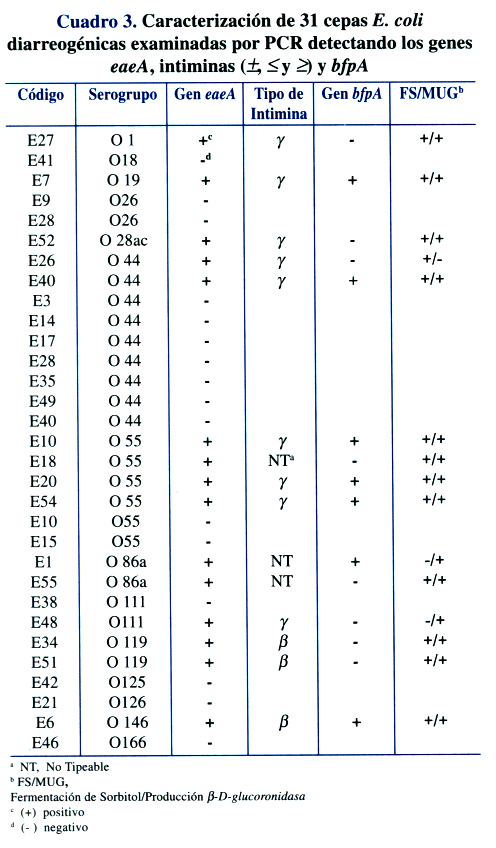
El análisis de las cepas clínicas de E. Coli aisladas en el periodo 1993, mostró que 15 de 31 cepas presentaron el gen eaeA (Cuadro # 3). En este grupo de muestras, se observó un 47% de cepas EPEC típicas.
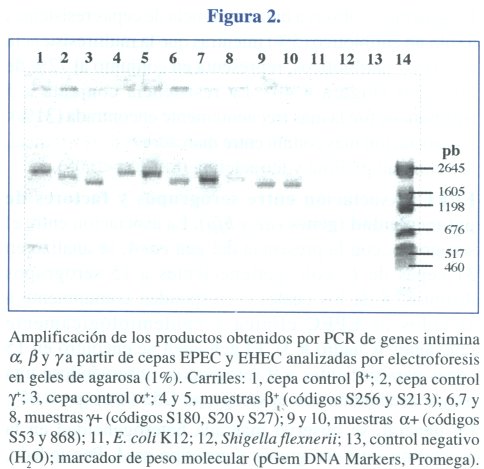
EPEC: Caracterización genética de las intiminas a, b y g. La identificación genética de tres tipos de intimina (a, b y g), se realizó mediante PCR de la región variable del gen eaeA en 72 cepas eae+ que incluyeron a: 15 aislados de 1993 y 57 del período 2001-2002 (Figura # 2).
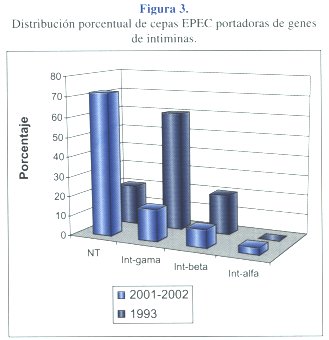
De las cepas analizadas entre el 2001-2002, 3% corresponden al gen de la intimina a, 9% al de la intimina b y 16% al de la intimina g. En el 72% restante, no se observaron productos de amplificación; por lo cual, estas cepas fueron clasificadas como no tipeables (Cuadro # 2).
Se observa que la proporción de cepas no tipeables fue menor entre los aislados de 1993 (20%) con respecto al periodo 2001-2002. El porcentaje de cepas de intimina tipo g fue mayor que el de las otras variantes de intimina identificadas. No se observaron cepas con intimina a en 1993 (Cuadro # 3 y figura #3).
La distribución del tipo de intimina entre las cepas típicas y atípicas aisladas en el 2001-2002, reveló que las intiminas b y g están presentes en cepas de genotipo bfpA+ y bfpA- aunque con mayor predominio en estas ultimas. Las dos cepas de tipo a encontradas, son atípicas.
EPEC: Fermentación de Sorbitol (FS) y Producción de b-D-glucoronidasa (MUG). El 85% de las cepas EPEC analizadas mostraron ser fermentadoras de sorbitol (FS+) y productoras de -D-glucoronidasa (MUG+). Sin embargo, se observaron asimismo fenotipos FS+/MUG- así como FS-/MUG+ en un 14 y 2% respectivamente. No se encontraron cepas FS-/MUG- (Cuadro # 2).
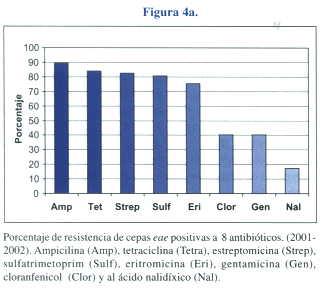
Resistencia a Antibióticos. El 99% de las cepas EPEC presento resistencia al menos a uno de los 8 antibióticos testados (Figura # 4).
Se observó por una parte, un porcentaje significativo (p<0,05) de cepas resistentes a: ampicilina (89%), tetraciclina (84%), estreptomicina (82%), sulfatrimetoprim (81%) y eritromicina (75%). Por otra, la proporción de cepas resistentes a la gentamicina (40%) y cloranfenicol (40%), y al ácido nalidíxico (17%), fue menor pero también significativa (p< 0,05) (Figura # 4a).
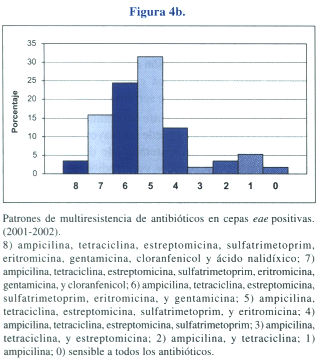
En general, se observa baja frecuencia de cepas resistentes a solo un antibiótico (5%) mientras que la multiresistencia a 5, 6 y 7 antibióticos, representa en conjunto al 72% de las cepas (Figura # 4b). La resistencia conjunta a 5 antibióticos fue la más frecuentemente encontrada (31%). La asociación más común entre marcadores de resistencia, fue entre ampicilina y tetraciclina (82%) (p<0,05).
EPEC: Asociación entre serogrupos y factores de patogenicidad (genes eae y bfp). La asociación entre el serogrupo, con la presencia del gen eaeA, se analizo en 31 cepas de E. Coli, pertenecientes a 15 serogrupos distintos, 8 de los cuales corresponden comúnmente a aislados de EPEC clínica y epidemiológicamente relevantes (Cuadro # 3). El gen eaeA se encontró en 15 cepas pertenecientes a 9 serogrupos, observándose una mayor asociación con el serogrupo O55 (4/6) (p< 0.05).
En general, se observo marcada heterogeneidad en la asociación entre serogrupos y genotipo eaeA. Por un lado, se observaron diferencias respecto a la presencia del gen eaeA entre aislados de un mismo serogrupo. Por otro, se encontraron aislados que corresponden a serogrupos prototipo de EPEC (O125, O126), pero que no portan el gene eaeA, así como serogrupos no relacionados a EPEC que son de genotipo eae+, (serogrupo O28ac comúnmente asociado a EIEC). Asimismo, se observan serogrupos comunes a EPEC pero que pueden estar presentes en otras categorías patogénicas como la cepa 0146 que en otros estudios17 esta relacionada a VTEC/STEC y que sin embargo, en este estudio no porta genes stx. (Cuadro # 3).
Los aislados eae+ de serogrupo definido fueron luego analizados respecto al tipo de intimina (Cuadro 3), observándose que la cepas con intimina g (9/15) corresponden a seis serogrupos diferentes, siendo O55 y O44 los más frecuentemente encontrados. Las cepas portadoras de intimina b se encontraron asociadas a dos serogrupos diferentes, siendo O119 el más común. Dos de las tres cepas no tipificables, fueron del serogrupo O86.
7 cepas EPEC típicas encontradas en este estudio se observaron tanto en cepas con intimina de tipo g (5/7) y b (2/7) y pertenecen a 5 diferentes serogrupos, 4 de los cuales son tradicionales de EPEC, siendo O55 el mas frecuentemente asociado. Las cepas atípicas pertenecen a los serogrupos O1, O44 y O119. Un mismo serogrupo puede estar asociado tanto a una cepa típica como atípica (O55, O86) (Cuadro # 3).
EHEC: Caracterización genotípica y fenotípica. La identificación de EHEC se realizo a través del análisis de los genes stx entre las cepas eae+ mdh+. La prevalencia de EHEC (eae+ stx+) fue de 0.34%. Se encontraron tres cepas clasificadas como EHEC que representan el 5% del total de cepas eae+. Una de éstas pertenece a la categoría de intimina g. Las 2 cepas restantes, fueron clasificadas como no tipeables. Dos de las tres cepas presentaron diferente patrón genotípico respecto a la distribución de toxinas Stx1 y Stx2. En cuanto a la caracterización fenotípica; dos de las tres cepas fueron identificadas por serología como O157 y O6 respectivamente, ambas cepas son FS-/MUG-. La cepa S45 que no presentó serogrupo identificable es FS+/MUG- (Cuadro # 4).
El análisis de la resistencia a antibióticos, mostró que a diferencia de los aislados de EPEC, las cepas EHEC, presentaron sensibilidad a siete antibióticos y solo se observó resistencia a la eritromicina.
Discusión
En los países en desarrollo, las tasas de mortalidad y morbilidad infantil están comúnmente asociadas a las EDA3. Las complicaciones que se derivan de estas enfermedades incluyen la malnutrición e incremento de los costos de atención en salud18. En este contexto, las EDA asociadas a patógenos bacterianos continúan siendo muy frecuentes a nivel mundial19-20.
Las diversas categorías patogénicas de E. Coli Diarreogénica se cuentan entre los patógenos bacterianos más importantes causantes de las EDA13, 21. Debido a que la identificación de DEC, requiere su diferenciación de la flora normal bacteriana, los métodos microbiológicos tradicionales no son útiles, siendo necesaria la aplicación de nuevas herramientas de diagnostico.
En este trabajo, se utilizaron distintos sistemas de PCR para la caracterización del perfil patogénico de las cepas de E. Coli causantes de la lesión A/E (AEEC). La detección por una parte del gen de la intimina (gen eaeA) y por otra, de los genes bfpA y stx permitió la caracterización de EPEC en cepas típicas o atípicas y su distinción de EHEC respectivamente22.
Los marcadores fenotípicos se utilizaron tanto para complementar la caracterización genotípica definida por PCR como para analizar la validez de su uso en el diagnostico.
Identificación y Caracterización de cepas EPEC. Los datos obtenidos en el presente trabajo, muestran que la prevalencia de AEEC fue del 7% con predominancia de EPEC (95%) sobre EHEC entre las cepas eae+ (p<0.05). Se observa también una alta frecuencia de EPEC atípica en relación a EPEC típica (p<0,05). Por lo tanto, las cepas EPEC atípicas tendrían mayor circulación entre la población analizada, reflejando por una parte, la relevancia de este grupo aislado de heces diarreicas de niños que requirieron asistencia hospitalaria y por otra, confirmando el rol del gen eaeA como factor importante de patogenicidad de EPEC. Sin embargo, en vista de la variación del gen bfpA encontrada en cepas EPEC23-24 será necesario reconfirmar estos datos con cebadores dirigidos hacia regiones conservadas comunes a los diferentes alelos bfpA.
La intimina es una proteína de membrana externa involucrada en la patogenicidad de EPEC y EHEC. Sus funciones están relacionadas a la adherencia de la bacteria a la célula eucariota a través de la interacción con la proteína receptora Tir, así como a la estimulación de la respuesta inmune de la mucosa intestinal5. El gen de la intimina forma parte de los genes codificantes para la lesión AE que están localizados en una isla de patogenicidad (PAI) llamada locus de la esfacelación del enterocito (LEE). La región que comprende este locus, es altamente conservada en EPEC y EHEC y contiene 41 y 54 marcos abiertos de lectura respectivamente. Estos genes están localizados en operones policistrónicos donde se encuentra el gen del receptor de la intimina (tir) además de otros asociados al sistema de secreción tipo III y adhesión de proteínas25-27.
La importancia de la intimina en la infección de EPEC y EHEC ha sido documentada en numerosos estudios tanto en cultivos celulares como en voluntarios28-29. Por otra parte, se han encontrado altos niveles de anticuerpos antiintimina en el suero de pacientes infectados con EPEC y EHEC30-32 así como en la leche humana33. Existen evidencias de que anticuerpos contra intimina de EPEC y EHEC podrían tener un efecto protectivo potencial contra la infección por estos patógenos33. Dado, que la infección por EPEC es más común que EHEC, se cree que EPEC conferiría protección inmunitaria de reacción cruzada hacia EHEC, causando la limitación de su circulación en la población.
De forma similar, a lo reportado en este trabajo, otros estudios34-40 han mostrado entre otros, mayor prevalecía de cepas EPEC atípicas en diferentes regiones geográficas.
Sin embargo, se ha reportado mayor prevalencia de cepas EPEC típicas en países en desarrollo, mientras que las atípicas constituyen los aislados mas frecuentes de EPEC en países industrializados41. Esta correlación, no se ajusta a lo observado en el presente estudio, lo que indicaría que existe variedad de patrones epidemiológicos asociados a la infección de EPEC típica y atípica en diferentes áreas geográficas.
En vista que EPEC atípica, no constituye un grupo homogéneo32,42, su rol en las EDA es aun controversial, por lo que será necesario realizar, por una parte, mayores estudios para definir y precisar su perfil de virulencia y por otra, evaluar su presencia en grupos control.
EPEC: Serogrupos y factores de virulencia. Previo al conocimiento de los factores de virulencia y patogenicidad de EPEC, su diagnóstico se basaba en la identificación de E. Coli perteneciente a determinados serotipos clínica y epidemiológicamen-te asociados a las EDA. En algunos reportes, la serología es aún considerada muy útil en el diagnostico presuntivo de EPEC43-44. En este estudio, se observa sin embargo, que la correlación entre los aislados de EPEC con diferentes serogrupos es muy heterogénea y no se distinguen datos consistentes (Cuadro 3). En general se presentan: a) diferencias respecto a la presencia del gen eaeA, entre aislados de un mismo serogrupo característico de EPEC (O44, O55 y O111), b) serogrupos de EPEC considerados tradicionales, que no portan el gen eae+ (serogrupo O26, O125), c) cepas EPEC típicas y atípicas en un mismo serogrupo (O44, O55, O86ac), d) serogrupos EPEC que se observan en otras categorías patogénicas (EHEC y EAEG) y finalmente e) La presencia del gene eaeA en un serogrupo especifico de EIEC.
Estas diferencias podrían estar relacionadas al número de cepas analizadas. Sin embargo, en otros reportes40, 42, 45-59 se ha observado discordancia entre el perfil de patogenicidad de esta categoría (analizado ya sea a través de la presencia de genes específicos o en base a los patrones fenotípicos de adherencia de las cepas) y la pertenencia a serogrupos y serotipos tradicionalmente característicos de EPEC. Asimismo, se han encontrado nuevos serotipos/serogrupos asociados a EPEC60 y presencia de alta diversidad clonal53,59. Estos fenómenos, probablemente relacionados a la diferencia de origen clonal entre cepas de E. Coli de un mismo serotipo, o a la emergencia de cepas con serogrupos previamente no asociados a EPEC, se evidencian en aislados de diferentes regiones geográficas61. Diferentes factores, como la diversidad de cepas circulantes, eventos de transferencia horizontal, estado nutricional del huésped, uso de antibióticos, condiciones sanitarias (acceso al agua potable, hacinamiento de viviendas, contacto con animales, inadecuado manejo de excretas) entre otros, los cuales varían entre una región a otra, podrían afectar a la relación entre determinados genes de patogenicidad y virulencia y un serogrupo o serotipo dado.
El grado de discordancia entre determinados serogrupos y categorías patogénicas puede ser mayor, si como ocurre en nuestro medio, los sistemas de diagnostico, son importados y están basados en estudios de correlación validos para otras regiones. Los resultados obtenidos, tienen importancia clínica y epidemiológica y remarcan la inespecificidad de los métodos basados en la identificación de serogrupo, en la asignación de categorías patogénicas sustentando la necesidad del uso de métodos alternativos de diagnostico de DEC y en particular de EPEC.
Al respecto, será importante analizar la presencia de otros genes de virulencia en las cepas (eae+ y eae-) para caracterizarlas más así como complementar los perfiles de virulencia con ensayos de adherencia in vitro.
EPEC: Tipificación de Intiminas. El análisis de la variación del gen de la intimina, en cepas EPEC aisladas con PDI, en base a los tres tipos predominantes de intimina (α, β y γ) mostró que las cepas no tipeables representan el mayor porcentaje (72%), seguidas de la intimina g e intimina b. La intimina a es poco frecuente. El alto porcentaje de cepas no tipeables refleja la variación del gene de la intimina en la región carboxilo- terminal donde se encuentra el sitio de unión al receptor-la proteína Tir cuyo gen presenta también polimorfismo. Otros estudios recientes, reportan de forma similar a este estudio alta proporción (70%) de intiminas no tipeables62.
Al presente, se han identificado 15 variantes entre tipos y subtipos del gen de la intimina en aislados de humanos y animales de EPEC y EHEC63-70. Existen evidencias de que el tipo de intimina tendría influencia sobre los sitios de colonización, lo que a su vez reflejaría especificidad de tropismo celular o tisular a nivel de la infección71.
En la Figura 3, se distingue la diferencia (p<0.05) en el porcentaje de muestras no tipeables en los períodos 1993 y 2001-2002. Aunque el número total de cepas analizadas es menor en 1993, esta diferencia podría atribuirse a que el gen de la intimina ha adquirido mayor variabilidad a lo largo del tiempo transcurrido y/o a la emergencia de nuevas cepas con variantes diferentes a las analizadas en este estudio.
En cuanto a la intimina g es interesante observar su asociación en casi similar proporción tanto a cepas EPEC típicas como atípicas (Cuadro # 3) que contrasta fuertemente con lo observado en otros estudios donde este tipo de intimina corresponde comúnmente a EHEC o a un set limitado de serotipos (O55:[H7] y O111ac:[H8]) de EPEC atípica41, 72. En este estudio, los 4 aislados eae+ de serogrupo O55 si bien tienen intimina γ, 3 de los cuales son también bfpA+. El serogrupo 0111 pertenece a una cepa atípica. El resto de las cepas con intimina g está distribuido en cuatro serogrupos diferentes que no corresponden a los serogrupos de EPEC epidemiológicamente más comunes.
La presencia de intimina β, puede también estar asociada tanto a cepas típicas como atípicas (Cuadro 3) por lo que en este caso tampoco se observa una correlación entre serogrupos, tipo de intimina y bundlina. Los datos de los aislados del 2001-2002, muestran de forma similar, la distribución de intimina β y g entre cepas típicas como atípicas aunque dada la preponderancia de cepas atípicas la asociación es mayor con esta últimas (Cuadro 2). En conjunto, estos datos, demuestran, la diversidad de cepas EPEC aisladas de heces diarreicas, que evidencian la heterogeneidad entre serogrupos, tipo de intimina y presencia de bundlina. En países en desarrollo, donde E. Coli Diarreogenica es mas frecuente y donde la proporción de infecciones es mayor, tanto por cepas endémicas como por aquellas provenientes de reservorios animales, podrían existir condiciones potenciales para mayor intercambio genético por transferencia horizontal, que explicarían la heterogeneidad encontrada de combinaciones entre factores de patogenicidad.
Estudios filogenéticos sugieren, que el sitio de inserción del locus LEE en el cromosoma bacteriano, está directamente relacionado al origen evolutivo de las cepas EPEC del grupo 1 y 225, 73-74. Las cepas caracterizadas como EPEC típicas con sus respectivos serotipos, pertenecen a la misma rama evolutiva, que contiene el locus LEE insertado dentro del locus selC del cromosoma bacteriano. En EPEC atípica, el LEE es insertado en el locus pheU o en otro sitio75. Dado que la región LEE, es una entidad dinámica en términos de la transferencia de genes, que favorecería la aparición de nuevas cepas patogénicas, será importante determinar en futuros estudios los sitios de inserción de este locus en el cromosoma bacteriano, las variantes de intimina, así como la relación filogenética entre las cepas EPEC encontradas.
EPEC: Caracterización Fenotípica. Si bien las cepas son mayoritariamente del fenotipo FS+/MUG+, existe un porcentaje apreciable de cepas MUG- (14%), que son atípicas. Estas cepas podrían estar filogenéticamente más relacionadas a los aislados de EHEC encontrados que presentan este mismo patrón. Estas observaciones deberán confirmarse en futuros estudios.
Resistencia a antibióticos. El análisis de la resistencia a antibióticos en los aislados de EPEC mostró tres patrones característicos. Por una parte, resalta la alta frecuencia de cepas resistentes a ampicilina, tetraciclina, streptomicina, sulfatri-metoprim y eritromicina en un rango entre 75% a 90% seguida de la resistencia a cloranfenicol y gentamicina alrededor del 40% y finalmente la resistencia al ácido nalidixico en un 17% (Figura # 4a). El primer y segundo patrón, donde se observan frecuencias similares de cepas resistentes para bloques de 5 y 2 antibióticos respectivamente (Figura # 4b), estarían reflejando la resistencia transmisible mediada por plásmidos, transposones o integrones. Este tipo de resistencia estaría sujeta a presión de selección por el uso inadecuado de antibióticos en nuestro medio, para el tratamiento de las EDA y otras enfermedades.
La amplia distribución de cepas multiresistentes en las cepas mas prevalentes de AEEC tiene relevancia epidemiológica. Es importante remarcar los datos encontrados donde los fenotipos mas prevalentes comprenden cepas EPEC multiresistentes a entre 5 y 7 antibióticos y donde casi la totalidad de cepas EPEC 57/58 es resistente a por lo menos un antibiótico. Los patrones de resistencia encontrados estarían reflejando los tipos de antibióticos más frecuentemente usados, la extensión de los mecanismos de transferencia horizontal y la prevalencía de infección de las cepas patógenas en la población.
En estudios de evaluación de la multiresistencia a antibióticos de EPEC y otros enteropatógenos44, 75-86 en otros países, se han encontrado datos similares, aunque en general en menor proporción de cepas multiresistentes.
El incremento de la prevalencía de la resistencia a antibióticos es un problema de salud publica mundial87. La terapia antimicrobiana desde su aplicación en gran escala hace mas de 50 años atrás, ha generado un incremento creciente de organismos resistentes. Virtualmente todas las especies de bacterias patógenas incluyen cepas resistentes a varios antibióticos utilizados para controlarlos. E. Coli como parte de la flora comensal intestinal tiene particularmente un gran potencial para el desarrollo de la resistencia a antimicrobianos y podría constituirse en un reservorio diseminable de genes de multiresistencia a bacterias patógenas o viceversa88. En este contexto, debe también considerarse que gran parte de los factores de patogenicidad y virulencia de DEC, al igual que los genes de multiresistencia a antibioticos, se encuentran asociados a elementos móviles. Se estima que las infecciones causadas por bacterias multiresis-tentes a antibióticos estarían asociadas a mayores tasas de morbilidad y mortalidad89. Este fenómeno derivado del uso inapropiado de antibióticos estaría influenciado por varios factores, como ser la ineficacia de los antibióticos y sus complicaciones en el tratamiento, el incremento de la virulencia microbiana por la asociación entre genes de virulencia y de resistencia a antibióticos y la ineficiente colonización de la microflora intestinal que favorecería la infección de organismos multiresistentes.
Por lo tanto, los datos reportados en este estudio deberán ser considerados en el tratamiento de EDAs y en el desarrollo de programas y estrategias de prevención que enfoquen el uso racional de antibióticos y el control de la emergencia y diseminación de cepas multiresistentes.
Caracterización de cepas EHEC. Las infecciones por EHEC, son un problema de salud pública en países como USA, Canadá, Inglaterra, y Japón90. En el hemisferio sur, EHEC es prevalerte en Chile, Argentina91 y Sudáfrica90. Estos datos contrastan con EPEC que es responsable de un número importante de muertes en niños menores de cinco años en países en vías de desarrollo13.
Los datos obtenidos en este estudio muestran la baja prevalencía de las infecciones de EHEC entre las EDA, que coincide con el patrón epidemiológico observado en otros países7,9,62,83,92-94. Sin embargo, se debe tomar en cuenta, que se consideraron solo a las cepas EHEC portadoras del locus LEE por lo que deberá analizarse en futuros trabajos el rol de las cepas EHEC no asociadas a la lesión A/E.
Las tres cepas EHEC encontradas portan el gen stx2. Varios estudios95 sugieren, que existiría sinergismo entre los genes stx2 y eaeA, asociado a una mayor virulencia de EHEC. Se observan también diferencias en las características fenotípicas con respecto a la fermentación de sorbitol, producción de glucoro-nidasa y serogrupo.
Estos datos reflejarían diferencias de origen clonal y señalarían de forma preliminar que no existe un patrón EHEC circulante predominante como el asociado a la cepa O157H7 (FS-/MUG-).
Es interesante notar en este número reducido de muestras, por una parte la presencia del serogrupo O157 característico de EHEC (en la cepa FS-/ MUG- de intimina no tipeable). Por otra parte, la presencia del serogrupo O6, característico de aislados de ETEC. A nuestro conocimiento, esta cepa representa una nueva variante de EHEC emergente en humanos, que no ha sido reportada previamente en la literatura. Recientemente se ha encontrado a este serogrupo en cepas EHEC de ovinos69.
En conjunto, estos datos deberán tomarse en cuenta al considerar lineamientos de diagnostico de EHEC basados en características fenotípicas (fermentación de sorbitol, producción de b-D-glucoronidasa, serogrupo asociado), pues no serian validos para su uso en nuestro medio. Se remarca nuevamente en esta otra categoría patogénica de DEC, que los patrones de caracterización fenotípica no son comunes ni extrapolables a distintas regiones geográficas.
Los resultados obtenidos en los ensayos de multiresistencia a antibióticos en cepas EHEC, contrastan significativamente (P< 0.05) con los datos de EPEC y podrían asociarse a la baja prevalecía de EHEC encontrada, y a que su circulación estaría reducida en humanos.
Dado que en Bolivia, el uso de antibióticos es mucho más común en húmanos que en animales, se podría inferir en base al patrón de multiresistencia a antibióticos, la emergencia de nuevas cepas a partir de origen animal o humano. Por lo tanto, los contrastes entre los niveles de multiresistencia a antibióticos de EHEC y EPEC reflejarían presiones de selección diferentes determinadas por las frecuencias de infección y circulación de estos patógenos en humanos y/o animales.
El presente estudio constituye un análisis preliminar de la características de dos categorías patogénicas portadoras del locus LEE, presentes en heces de niños con PDI, que servirá de base para futuros estudios epidemiológicos, donde deberá confirmarse su asociación a las EDA.
Agradecimiento
Este trabajo fue realizado en el Instituto de Biología Molecular y Biotecnología (IBMB) con apoyo parcial del Programa de colaboración sueca ASDI-SAREC y constituyó parte de los trabajos de tesis de grado de licenciatura de Samanta Sánchez y Paola Romecín.
Los autores agradecen al personal del Hospital del Niño Ovidio Aliaga y Hospital Materno Infantil. Asimismo a la Dra. Patricia Lada, al Dr. Alberto Benítez y al Sr. Natalio Medrano por la colaboración prestada.
Referencias
1. Snyder J, Merson M. The magnitude of the global problem of acute diarrhoeal disease: a review of active surveillance data. Bull World Health Organ 1982; 60: 605. [ Links ]
2. Bern C, Martines J, Zoysa I, Glas R I. The magnitude of the global problem of diarrhoeal disease: a ten-year update. Bull. World Health Organ 1992; 70: 705-14. [ Links ]
3. Parashar U D, Bresee J, Glass R I. The global burden of diarrhoeal disease in children. Bulletin of the World Health Organization 2003; 81: 236. [ Links ]
4. Encuesta Nacional de Demografía y Salud (ENDSA) 1994. Salud del Niño.
5. Kaper JB, Nataro JP, Mobley LH. Pathogenic Escherichia coli. Nature 2004; 2: 123-40. [ Links ]
6. Levine M, Prado V, Robins-Browne R, Lior H, Kaper J B, Moseley S L, et al. Use of DNA probes and HEp-2 cell adherence assay to detect diarrheagenic Escherichia coli. J. Infect. Dis 1988; 155: 377-89. [ Links ]
7. Albert J, Faruque M, Faruque G, Neogi P, Ansaruzzaman N, Bhuiyan A, et al. Controlled Study of Escherichia coli Diarrheal Infections in Bangladeshi Children. J. Clin. Microbiol 1995. 33: 973-77. [ Links ]
8. González R, Díaz M, Marino M, Cloralt R, Pequeneze M, Pérez-Schael I. Age-specific prevalence of Escherichia coli with localized and aggregative adherence in Venezuelan infants with acute diarrhea. J. Clin. Microbiol 1997; 35: 1103-07. [ Links ]
9. Rosa A, Mariano AT, Pereira AMS, Tibana A, Gomes T, Andrade J. Enteropathogenicity markers in Escherichia coli isolated from infants with acute diarrhoea and healthy controls in Rio de Janeiro, Brazil. J. Med. Microbiol 1998; 47: 781-90. [ Links ]
10. Almeida MT, Silva RM, Donaire LM, Moreira LE, Martinez MB. Enteropathogens associated with acute diarrheal disease in children. J. Pediatr. (Rio J) 1998;74:291-98. [ Links ]
11. Toporovski MS, Mimica IM, Chieffi PP, Paschoalotti MA, Dias AM, Silva CB. Acute diarrhea in children less than 3 years of age: Enteropathogens isolated in patient's stools, compared with a control group. J. Pediatr. (Rio J) 1999; 75:97-104. [ Links ]
12. Souza EC, Martinez MB, Taddei CR, Mukai L, Gilio AE, Racz ML, et al. Etiologic profile of acute diarrhea in children in Sao Paulo. J. Pediatr. (Rio J) 2002; 78: 31-8.
13. Nataro J, Karper J. Diarrheagenic Escherichia coli. J. Microbiol 1998; 11: 147-78. [ Links ]
14. Griffin PM, Tauxe RV. The eppidemiology of infections caused by Escherichia coli O157:H7, other enterohemorrhagic Escherichia coli, and the associated hemoplytic uremic sydrome. J. Clin. Microbiol 1991; 13: 60-8. [ Links ]
15. Karch H, Bielaszewska M, Bietzan M, H Schmidt. Epidemiology and diagnosis of Shiga toxin-producing Escherichia coli infections. Diagnostic Microbiology and Infectious Disease 1999; 34: 229-43.
16. National Committee for Clinical Laboratory Standards. Performance standards for antimicrobial disk susceptibility tests. Approved standard, 7th ed. NCCLS M2-A7 2000; National Committee for Clinical Laboratory Standards, Wayne, Pa. [ Links ]
17. Beutin L, Krause G, Zimmermann S, Kaulfuss S, Gleier K. Characterization of Shiga toxin-producing Escherichia coli strains isolated from human patients in Germany over a 3-year period. J. Clin. Microbiol 2004; 42: 1099-108. [ Links ]
18. World Health Organization. Childhood diarrheal disease and nutrition (CDDN) group -contribution to development of vaccines for children in developing countries. 2001. [ Links ]
19. Guerrant R, Bobak D. Bacterial and protozoal gastroenteritis. N. Engl. J. Med 1991; 325: 327-40. [ Links ]
20. Guerrant R, Why America must care about tropical medicine: threats to global health and security from tropical infectious diseases. Am. J. Trop. Med. Hyg 1998; 59: 3-16. [ Links ]
21. Vaishnavi C, Kaur S. The epidemiological and resistogram patterns of enteropathogenic and enterotoxigenic Escherichia coli isolated from diarrhoeal stools in a north Indian hospital. Trop. Gastroenterol 2003; 24: 70-2. [ Links ]
22. Yatsuyanagi J, Saito S, Miyajima Y, Amano K, Enomoto K. Characterization of atypical enteropathogenic Escherichia coli strains harboring the astA gene that were associated with a waterborne outbreak of diarrhea in Japan. J. Clin. Microbiol 2003; 41:2033-39. [ Links ]
23. Blank TE, Zhong H, Bell A, Whittam TS, Donnenberg MS. Molecular Variation among Type IV Pilin (bfpA) Genes from Diverse Enteropathogenic Escherichia coli Strains. Infection and immunity 2000; 68: 7028-38. [ Links ]
24. Gunzburg S, Tornieporth N, Riley L. Identification of Enteropathogenic Escherichia coli by PCR-Based Detection of the Bundle-Forming Pilus Gene. J. Clin. Microbiol 1995; 33: 1375-77. [ Links ]
25. Frankel A, Phillips L, Rosenbine D, Dondan JB, Kaper J, Knutton S. Enterophatogenic and Enterohaemorrhagic Escherichia coli. Mol. Microbiol. Commission of the European Communitie 1998. 30: 911-21. [ Links ]
26. DeVinney R, Gauthier A, Abe A, Finlay BB. Enteropathogenic Escherichia coli: a pathogen that inserts its own receptor into host cells. Cell. Mol. Life Sci 1999; 55: 961-76. [ Links ]
27. Clarke SC, Haigh RD, Freestone P, Williams PH. Virulence of Enteropathogenic Escherichia coli, a Global Pathogen. Clin. Microbiol. Reviews 2003; 16: 36578. [ Links ]
28. Donnenberg M, Tacket C, James SP. Role of the eaeA gene in experimental enteropathogenic Escherichia coli infection. J. Clin. Invest 1993; 92: 1412-17. [ Links ]
29. Hicks S, Frankel G, Kaper JB. Role of intimin and bundle-forming pili in enteropathogenic Escherichia coli adhesion to pediatric intestinal tissue in vitro. Infect. Immun 1998; 66: 1570-78. [ Links ]
30. Martínez M, Taddei C, Ruiz-Tagle A, Trabulsi LR, Giron JA. Antibody response of children with enteropathogenic Escherichia coli infection to the bundle-forming pilus and locus of enterocyte effacement-encoded virulence determinants. J. Infect. Dis 1999; 179: 269-74. [ Links ]
31. Jenkins C, Chart H, Smith HR. Antibody response of patients infected with verocytotoxin-producing Escherichia coli to protein antigens encoded on the LEE locus. J. Med. Microbiol 2000. 49: 97-101. [ Links ]
32. Karpman D, Bekassy Z, Sjogren AC, Dubois MS, Karmali MA, Mascarenhas M, et al. Antibodies to intimin and Escherichia coli secreted proteins A and B in patients with enterohemorrhagic Escherichia coli infections. Pediatr. Nephrol 2002; 17: 201-11. [ Links ]
33. Parissi-Crivelli A, Parissi-Crivelli J, Giron JA. Recognition of Enteropathogenic Escherichia coli Virulence Determinants by human Colostrum and Serum Antibodies. J. Clin. Microbiol 2000; 38: 2696-2700. [ Links ]
34. Pedroso M, Freymuller E, Travulsi LR, Gomez TA. Attaching-effacing lesions and intracellular penetration in Hale cells and human duodenal mucosa by two Escherichia coli strains not belonging to the classical enteropathogenic Escherichia coli serogroups. Infect. Immun 1993; 61: 1152-56. [ Links ]
35. Pelayo J, Scaletsky I, Smith HR. Virulence properties of atypical EPEC strain. J. Clin. Microbiol 1999; 48: 41-9. [ Links ]
36. Bouzari S, Jafari M, Shokouhi F, Parsi M, Jafari A. Virulence-related DNA sequences and adherence patterns in strains of enteropathogenic Escherichia coli. FEMS Microbiology Letters 2000; 185: 89-3. [ Links ]
37. Galane PM, Le Roux M. Molecular epidemiology of Escherichia coli isolated from young South African children with diarrhoeal diseases. J. Health Popul. Nutr 2001; 19:31-8. [ Links ]
38. Paciorek J. Virulence properties of Escherichia coli faecal strains isolated in Poland from healthy children and strains belonging to serogroups O18, O26, O44, O86, O126 and O127 isolated from children with diarrhoea. Med. Microbiol 2002; 51: 548-71. [ Links ]
39. Yatsuyanagi J, Saito Shioko, Sato Hiroyasu, Miyajima Yoshimichi, Amano Ken-Ichi, Enomoto Katsuhiko. Characterization of Enteropathogenic and Enteroaggregative Escherichia coli Isolated from Diarrheal Outbreaks. J. Clin. Microbiol 2002; 40: 294-97. [ Links ]
40. Afset J, Bergh K, Bevanger L. High prevalence of atypical enterophatogenic Escherichia coli (EPEC) in Norwegian children with diarrhoea. J. Clin. Microbiol 2003; 52: 1015-19. [ Links ]
41. Trabulsi L, Keller R, Gomea T. Typical and Atypical Enteropathogenic Escherichia coli. Emerging Infectious Diseases 2002; 8: 508-13. [ Links ]
42. Dulguer M, Fabbricotti S, Bando SY, Moreira-Filho CA, Fagundes-Neto U, Scaletsky IC. Atypical enteropathogenic Escherichia coli strains: phenotypic and genetic profiling reveals a strong association between enteroaggregative Escherichia coli heat-stable enterotoxin and diarrhea. J. Infect. Dis 2003; 188: 1685-94. [ Links ]
43. Prasannan M, Jesudason M, Kang G, Sridharan G. A study on some phenotypic virulence markers of enteropathogenic Escherichia coli. Indian J. Med. Res 2001;114: 95-8. [ Links ]
44. Mani R, Udgaonkar U, Pawar S. Study of enteropathogenic Escherichia coli(EPEC) diarrhoea in children. Indian J. Pathol. Microbiol 2003: 461:118-20. [ Links ]
45. Smith R, Scotland SM, Stokes N, Rowe B. Examination of strains belonging to enteropathogenic Escherichia coli serogroups for genes encoding EPEC adherence factor and Vero cytotoxins. J. Med. Microbiol 1990; 31: 235-40. [ Links ]
46. Scotland SM, Smith HR, Said B, Willshaw GA, Cheasty T, Rowe B. Identification of enteropathogenic Escherichia coli isolated in Britain as enteroaggregative or as members of a subclass of attaching-and-effacing E. Coli not hybridising with the EPEC adherence-factor probe. J. Med. Microbiol 1991; 35: 278-83. [ Links ]
47. Scotland M, Smith HR, Cheasty T, Said B, Willshaw GA, Stokes N, et al. Enteroaggregative Escherichia coli Strains among Classical Enteropathogenic Escherichia coli O Serogroups. J. Med. Microbiol 1996; 44: 438-43. [ Links ]
48. Giammanco A, Maggio M, Giammanco G, Morelli R, Minelli F, Scheutz F, et al. Characteristics of Escherichia coli strains belonging to enteropathogenic Escherichia coli serogroups isolated in Italy from children with diarrhea. Clin. Microbiol 1996; 34: 689-94. [ Links ]
49. do Valle GR, Gomes TA, Irino K, Trabulsi LR The traditional enteropathogenic Escherichia coli (EPEC) serogroup O125 comprises serotypes which are mainly associated with the category of enteroaggregative E. Coli. FEMS Microbiol Lett 1997; 152:95-100. [ Links ]
50. Yamazaki M, Saito M, Inuzuka K, Shima S, Taniwaki H, Ito. eaeA genes in Escherichia coli derived from Japanese patients with sporadic diarrhea. Kansenshogaku Zasshi 1997; 71: 1059-65. [ Links ]
51. Ortiz A, Ruttler M, Garcia B, Balbi L, Cruzado M, Risler N, et al. Accumulation of actin and adhesion to HEp-2 cells of strains of Escherichia coli isolated from children with diarrhea in Mendoza, Argentina. Rev. Argent. Microbiol 1998; 30:13-9. [ Links ]
52. Sunabe T, Honma Y. Relationship between O-serogroup and presence of pathogenic factor genes in Escherichia coli. Microbiol. Immunol 1998; 42: 845-49. [ Links ]
53. Ansaruzzaman M, Albert MJ, Nahar S, Byun R, Katouli M, Kuhn I, et al. Clonal groups of enteropathogenic Escherichia coli isolated in case-control studies of diarrhoea in Bangladesh. J. Med. Microbiol 2000; 49:177-85. [ Links ]
54. Vidotto MC, Kobayashi RK, Dias AM. Unidentified serogroups of enteropathogenic Escherichia coli (EPEC) associated with diarrhoea in infants in Londrina, Parana, Brazil. J. Med. Microbiol 2000; 49:823-26. [ Links ]
55. Sobieszczanska B, Gryko R. Adherence patterns of Escherichia coli strains isolated from children with diarrhea. Przegl. Epidemiol 2001; 55: 287-97. [ Links ]
56. Elías Samar W, Barros Cristiano G, Moreira Luiz R, Trabulsi TA, Gomes T. Atypical enteropathogenic Escherichia coli. J. Clin. Microbiol 2002; 40: 3540-41. [ Links ]
57. Kobayashi K, Seto K, Yatsuyanagi J, Saito S, Terao M, Kaneko M, et al. Presence of the genes regarding adherence factors of Escherichia coli isolates and a consideration of the procedure for detection of diarrheagenic strain. Kansenshogaku Zasshi 2002; 76:911-20. [ Links ]
58. Piva IC, Pereira AL, Ferraz LR, Silva RS, Vieira AC, Blanco JE, et al. Virulence markers of enteroaggregative Escherichia coli isolated from children and adults with diarrhea in Brasilia, Brazil. J. Clin. Microbiol 2003; 41:1827-32. [ Links ]
59. Kahali S, Sarkar B, Chakraborty S, Macaden R, Deokule JS, Ballal M, et al. Molecular epidemiology of diarrhoeagenic Escherichia coli associated with sporadic cases and outbreaks of diarrhoea between 2000 and 2001 in India. Eur. J. Epidemiol 2004; 19:473-79. [ Links ]
60. Barlow RS, Hirst RG, Norton RE, Ashhurst-Smith C, Bettelheim KA. A novel serotype of enteropathogenic Escherichia coli (EPEC) as a major pathogen in an outbreak of infantile diarrhoea. J. Med. Microbiol 1999; 48: 1123 -125. [ Links ]
61. Peixoto JC, Bando SY, Ordonez JA, Botelho BA, Luiz R, Trabulsi LR, Moreira-Filho CA. Genetic diferences between Escherichia coli O26 strains isolated in Brazil and in other countries. FEMS Microbiology Letters 2001; 196: 239-44. [ Links ]
62. Nunes BE, Saridakis H, Irino K, Sánchez Pelayo J. Genotypicand phenotypic characterization of attaching and effacing (AEEC) isolates from children with and without diarrhoea in Lodrina, Brazil. J. Med. Microbiol 2003; 52: 499-504. [ Links ]
63. AduBobie J, Frankel G, Goncaleves AG, Trabulsi LR, Douce G, Knutton S, et al. Detection of intimin ±, , and ´, four intimin derivates expressed by attaching an effacing microbial pathogen. J. Clin. Microbiol 1998; 36: 662-68. [ Links ]
64. McGraw E, Li J, Selander RK, Whittam TS. Molecular Evolution and Mosaic Structure of ±, and intimin of Pathogenic Escherichia coli. Institute of Molecular Evolutionary Genetic 1999; 1: 12-2. [ Links ]
65. Oswald H, Schmidt HS, Morabito O, Karch, Marches Caprioli A. Typing of intimin genes in human and animal enterohemorrhagic and enteropathogenic Escherichia coli: characterization of a new intimin variant. American Society for Microbiology 2000; 68: 64-70. [ Links ]
66. Zhang W, Kohler B, Oswald D, Beutin L, Karch H, Morabito S, et al. Genetic Diversity of Intimin genes of Attaching and Effacing Escherichia coli Strain. J. Clin. Microbiol 2002; 40: 4486-92. [ Links ]
67. Ramachandran V, Brett K, Hornitzky MA, Dowton M, Bettelheim KA, Walker MJ, et al. Distribution of intimin subtypes among Escherichia coli isolates from ruminant and human sources. J. Clin. Microbiol 2003; 41:5022-32. [ Links ]
68. Jores J, Zehmke K, Eichberg J, Rumer L, Wieler LH. Description of a novel intimin variant (type zeta) in the bovine O84:NM verotoxin-producing Escherichia coli strain 537/89 and the diagnostic value of intimin typing. Exp. Biol. Med 2003; 228: 370-76. [ Links ]
69. Blanco M, Blanco J, Mora A, Rey J, Alonso JM, Hermoso M, et al. Serotypes virulence genes and intimin types of Shiga toxin (Verotoxin)-producing Escherichia coli isolates from healthy sheep in Spain. J. Clin. Microbiol 2003; 41: 1351-356. [ Links ]
70. Blanco M, Blanco J, Mora A, Dahbi G, Alonso MP, González EA, et al. Serotypes, virulence genes and intimin types of Shiga toxin (Verotoxin)-producing Escherichia coli isolates from cattle in Spain: identification of a new intimin variant gene (eae-xi). J. Clin. Microbiol 2004; 42: 645-51. [ Links ]
71. Fitzhenry R, Pickard D, Hartland EL, Reece S, Dougan G, Phillips AD, et al. Intimin type influences the site of human intestinal mucosal colonisation by enterohaemorrhagic Escherichia coli O157:H7 2002; 50: 180-85. [ Links ]
72. Reid S, Betting D, Whittam TS. Molecular Detection and Identification of Intimin Alleles in Pathogenic Escherichia coli by Multiplex PCR. J. Clin. Microbiol 1999; 37: 2719-22. [ Links ]
73. Whittam TS, McGraw E. Clonal analysis of EPEC serogroups. Rev. Microbiol 1996. 27: 7-16. [ Links ]
74. Donnenberg MS, TS Whittam. Pathogenesis and evolution of virulence in enteropathogenic and enterohemorrhagic Escherichia coli. The Journal of Clinical Investigation 2001; 107: 539-48. [ Links ]
75. Wieler LH, MacDaniel TK, Whittam TS, Kaper JB. Insertion site of locus of enterocyte effacement in enteropathogenic and enterohemorrhagic Escherichia coli differs in relation to clonal phylogeny of the strains. FEMS Microbiol. Lett 1997; 156: 49-3. [ Links ]
76. Thoren A, Antibiotic sensitivity of enteropathogenic Escherichia coli to mecillinam, trimethoprim-sulfamethoxazole and other antibiotics. Acta. Pathol. Microbiol. Scand 1980; 88: 265-68. [ Links ]
77. Abbar F, Khalef S, Yonis D. A prospective study of some diarrhoeagenic Escherichia coli in infants with diarrhoea in Mosul, Iraq. Ann. Trop. Paediatr 1991; 11: 99-2. [ Links ]
78. Gomes TA, Rassi V, MacDonald KL, Ramos SR, Trabulsi LR, Vieira MA, et al. Enteropathogens associated with acute diarrheal disease in urban infants in Sao Paulo, Brazil. J. Infect. Dis 1991; 164:331-33. [ Links ]
79. Senerwa D, Mutanda L, Gathuma JM, Olsvik O. Antimicrobial resistance of enteropathogenic Escherichia coli strains from a nosocomial outbreak in Kenya. APMIS 1991; 99: 728-34. [ Links ]
80. Samonis G, Maraki S, Christidou A, Georgiladakis A, Tselentis Y. Bacterial pathogens associated with diarrhoea on the island of Crete. Eur. J. Epidemiol 1997; 13: 831-36. [ Links ]
81. Obi C, Coker A, Epoke J, Ndip RN. Distributional patterns of bacterial diarrhoeagenic agents and antibiograms of isolates from diarrhoeaic and non-diarrhoeaic patients in urban and rural areas of Nigeria. Cent. Afr. J. Med 1998; 44: 223-29. [ Links ]
82. Vila J, Vargas M, Casals C, Urassa H, Mshinda H, Schellemberg D, et al. Antimicrobial Resistance of Diarrheagenic Escherichia coli Isolated from Children under the Age of 5 Years from Ifakara, Tanzania. Antimicrobial Agents and Chemotherapy 1999; 43: 3022-24. [ Links ]
83. Torres ME, Pírez MC, Schelotto F, Varela G, Parodi V, Allende F, et al. Etiology of Children's Diarrhea in Montevideo, Uruguay: Associated Pathogenes and Unusual Isolates. J. Med. Microbiol 2001; 39: 2134-39. [ Links ]
84. Battikhi M. Epidemiological study on Jordanian patients suffering from diarrhoea. New. Microbiol. 2002; 25: 405-12. [ Links ]
85. Wan K, Radu S, Cheah YK, Benjamin PG, Ling CM, Hon SF, Harun A, et al. Antibiotic resistance, plasmid profile and RAPD-PCR analysis of enteropathogenic Escherichia coli (EPEC) clinical isolates. Southeast Asian J. Trop. Med. Public Health 2003; 34: 620-26. [ Links ]
86. Maraki S, Georgiladakis A, Tselentis Y, Samonis G. A 5-year study of the bacterial pathogens associated with acute diarrhoea on the island of Crete, Greece, and their resistance to antibiotics. Eur. J. Epidemiol 2003; 18: 85-90. [ Links ]
87. World Health Organization. Report on infectious diseases 2000: overcoming antimicrobial resistance 2000; http://www.who.int/infectious-disease-report/index.html. [ Links ]
88. Barreta J. Evaluación de la resistencia a antibioticos de cepas de E. Coli de muestras fecales provenientes de niños sanos en edad escolar de la ciudad de La Paz. 2002. Tesis de licenciatura. UMSA. La Paz, Bolivia. [ Links ]
89. Holmberg S, Solomon S, Blake PA. Health and economic impacts of antimicrobial resistance. Rev. Infect. Dis 1987; 9:1065-78. [ Links ]
90. Paton J, Paton A. Pathogenesis and Diagnosis of Shiga Toxin-Producing Escherichia coli Infections. Clinical Microbiology 1998; 11: 450-79. [ Links ]
91. Rivero MA, Padola NL, Etcheverria AI, Parma AE. Enterohemorrhagic Escherichia coli and hemolytic-uremic syndrome in Argentina. Medicina (B Aires) 2004; 64:352-56. [ Links ]
92. Iruka NO, Adebayo L, Steinruck H, Kaper JB. Characterization of Escherichia coli Strains from Cases of Childhood Diarrhea in Provincial Southwestern Nigeria. J. Clin. Microbiol 2000; 38: 7-12. [ Links ]
93. Cortes-Ortiz IA, Rodriguez-Angeles G, Moreno-Escobar EA, Tenorio-Lara JM, Torres-Mazadiego BP, Montiel-Vazquez E. Outbreak caused by Escherichia coli in Chalco, Mexico. Salud Publica Mex 2002; 44:297-2. [ Links ]
94. Regua-Mangia AH, Gomes TA, Vieira MA, Andrade JR, Irino K, Teixeira LM. Frequency and characteristics of diarrhoeagenic Escherichia coli strains isolated from children with and without diarrhoea in Rio de Janeiro, Brazil. J. Infect 2004; 48:161-67. [ Links ]
95. Boerlin P, McEwen SA, Boerlin-Petzold F, Wilson JB, Jhonson RP, Carlton LG. Associations between Virulence Factors of Shiga Toxin-Producing Escherichia coli and Disease in Humans. J. Clin. Microbiol 1999; 37: 487-503. [ Links ]