El HTLV-1 está asociado mayoritariamente a la leucemia/ linfoma de células T adultas y a la paraparesia espástica tropical. A nivel mundial se ha estimado que hay 10 millones de seres humanos infectados con HTLV-1 los cuales se encuentran distribuidos heterogéneamente, presentándose un estado de endemicidad en Sudamérica, principalmente en Brasil, Perú, Chile y Colombia1.
En Perú la detección de esta infección busca identificar anticuerpos anti-HTLV en suero o plasma mediante ensayos inmunoabsorbentes ligados a enzimas (ELISA)2,3, quimioluminiscencia (CLIA) o electroquimioluminiscencia (ECLIA)4, aquellas muestras con resultados reactivos, indeterminados o discordantes, se confirman con inmunofluorescencia indirecta (IFI)5, inmunoblot (IB)6 y/o Western blot (WB)7. IFI HTLV-1 es económica, pero sólo resuelve hasta un 90% de los casos analizados, observándose resultados inespecíficos e indeterminados3,5. Las pruebas de IB/WB arrojan entre 5 a 10% de resultados no concluyentes además su alto costo es la principal limitante para su uso6-7.
Las pruebas moleculares son utilizadas para resolver los casos no concluyentes a las pruebas serológicas confirmatorias; la reacción en cadena de la polimerasa (PCR) presentó buena eficiencia diagnóstica en distintos laboratorios de otros países endémicos8-11.
Otra ventaja a destacar de la PCR frente a las pruebas serológicas es que pueden ser útiles en el diagnóstico del HTLV-1 en recién nacidos ya que, al encontrarse anticuerpos de madres infectadas en los recién nacidos o lactantes, las pruebas serológicas pueden dar falsos positivos12.
A nivel mundial no se dispone de kits comerciables de técnicas moleculares para el diagnóstico cualitativo y cuantitativo de HTLV-1, de allí que cada país desarrolla y estandariza su propia prueba in house8-11, resultando bastante económica en comparación con los IB/WB comerciales6,7.
Brasil y Chile tienen un algoritmo o flujograma normativo que incluye las pruebas moleculares en el diagnóstico del paciente con HTLV-19,13-14, sin embargo, Perú no lo tiene, tal es así que, conociendo esa necesidad, se planteó el siguiente objetivo: estandarizar y evaluar una prueba de PCR múltiplex anidada in house de bajo costo para el diagnóstico de HTLV-1.
Materiales y métodos
La investigación observacional prospectiva de evaluación diagnóstica se desarrolló entre los años 2018 y 2019 en el Instituto Nacional de Salud (INS) de Perú.
Muestras empleadas.
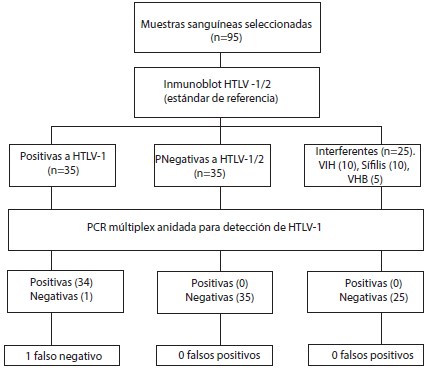
Todas las muestras sanguíneas fueron seleccionadas de manera no probabilística y por conveniencia, las cuales procedieron del Laboratorio de Referencia Nacional Virus de Transmisión Sexual (LRN VTS) del INS. De la hemoteca se seleccionó un total de 95 muestras sanguíneas obtenidas en tubos con anticoagulante EDTA de las cuales 35 fueron positivos al virus HTLV-1, 35 negativos a los virus HTLV 1 y 2 procedente de personas aparentemente sanas y 25 muestras interferentes: Negativas a HTLV 1/2 y positivas a VIH10, Sífilis10 y VHB5 (Figura 1). Para estas 95 muestras, la prueba Gold Estándar fue el IB comercial (Fujirebio) que destaca un 100% de sensibilidad y un 99% de especificidad6. Adicionalmente se usó como controles de Referencia: linfocitos infectados con el virus HTLV-1 (línea celular MT2) y macrófagos sin infectar (línea celular K562)5.
Extracción de ADN
Se utilizó el kit comercial GeneJET™ Whole Blood Genomic DNA Purification Mini Kit (Thermo Fisher Scientific Inc). La cuantificación de ADN se determinó con el Qubit dsDNA HS Assay Kit (Invitrogen) y fueron conservados en -20 °C hasta su utilización.
Estandarización
Se utilizó agua grado biología molecular (Applichem), MgCl2 y buffer de reacción de PCR NovaTaq Hot Star Buffer (Merck Millipore); dNTPs Mix (Invitrogen) y la enzima NovaTaq Hot Start ADN Polymerase (Merck Millipore). Evaluamos distintas concentraciones de MgCl2, primers, dNTPs y Taq polimerasa. Se usó como material de referencia ADN (20 ng/µL) de células MT2 infectadas con HTLV1; se utilizaron primers dirigidos a la región LTR y Pol del genoma de HTLV-1, también se usó primers para el gen de la β-globina como control interno endógeno. Para la reacción final se consideró 25 µL de volumen usando un termociclador Biometra TOne (Analytik Jena); la desnaturalización fue hecha a 95°C por siete minutos, seguida de 35 ciclos (94°C por un minuto, 55 por un minuto, 72°C por dos minutos) una extensión final de 72°C por siete minutos y 4°C indefinido. Las condiciones de la PCR respecto a concentraciones y temperaturas fueron las mismas para ambas rondas, exceptuando que en la segunda ronda se emplearon distintos primers más dos μL del producto amplificado de la ronda inicial (Tabla 1).
Tabla 1. Primers usados en la evaluación y estandarización de la PCR múltiplex anidada para el diagnóstico de HTLV-1
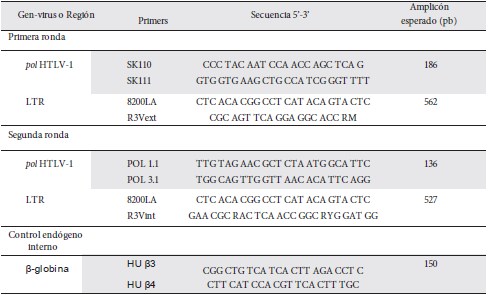
La corrida electroforética se realizó al 2,0% de agarosa usando un peso molecular de 100 pb como marcador de la marca Thermo Scientific. Se visualizaron las bandas usando el fotodocumentador ChemiDoc XRS de Biorad. El criterio de positividad, se definió de la siguiente manera: una muestra fue considerada como positiva si se evidenció la presencia del amplicon de 136 pb o del amplicon de 527 pb, o si hubo la presencia de ambos amplicones; además una muestra fue considerada negativa si no se evidenció la presencia de ninguno de los amplicones de 136 pb o de 527 pb.
Validación del ensayo
Para evaluar el límite de detección se analizaron diluciones seriadas del ADN proviral desde 50 a 0,001 ng/µL extraído del material de referencia (células MT2 infectadas). El límite de detección considerado fue la mínima concentración en la cual se visualizó los amplicones esperados. Se calculó los parámetros de precisión diagnóstica. La repetibilidad y reproducibilidad se evaluaron con tres concentraciones del material de referencia: frente al límite de detección y frente al 20% mayor y 20% menor al límite de detección; bajo las condiciones estandarizadas se realizaron cinco repeticiones para cada una de las tres concentraciones en tres días diferentes (15 repeticiones por cada concentración), con un analista distinto por día.
Resultados
La PCR se estandarizó en los siguientes parámetros: desnaturalización a 95°C, hibridación a 55°C, elongación a 72°C, 35 ciclos de amplificación. Las concentraciones óptimas por reacción fueron: MgCl2 4,0 mM; 0,5 U/μL de Taq polimerasa; 0,2 pmol/μL para Primers de Pol y 0,4 pmol/μL a LTR; 0,2 Mm para dNTPs. El límite de detección de ADN proviral extraído del material referencial (MT2) fue de 0,5 ng/ uL. Asimismo, en todas las muestras sanguíneas sometidas a extracción de ADN se mostró amplificación del control endógeno interno.
Del total de muestras sanguíneas incorporadas al estudio y evaluadas mediante la PCR múltiplex anidada in house para detección de HTLV-1, no resultó ningún falso positivo y sólo se obtuvo un falso negativo (Figura 1).
Fuente: bases de datos de la investigación
Figura 1. Diagrama de flujo de muestras incorporadas al estudio
Los resultados de los parámetros de rendimiento diagnóstico se muestran en la Tabla 2, además, se obtuvo un 100% de especificidad analítica, no encontrándose reactividad cruzada frente a las 25 muestras de tres agentes infecciosos distintos (Sífilis, VIH y VHB).
Tabla 2. Parámetros de rendimiento diagnóstico de la PCR múltiplex anidada para diagnóstico de HTLV-1, considerando al IB como estándar de referencia.
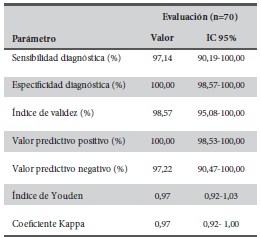
Fuente: bases de datos de la investigación
Otros hallazgos importantes a destacar fue que la repetibilidad y reproducibilidad para concentraciones de 0.4 ng/μL, 0.5 ng/μL, 0.6 ng/μL de ADN y la tasa de detección (intraensayo e interensayo) del material referencial para todas las repeticiones tuvo una concordancia del 100%.
La interpretación del perfil electroforético de la PCR múltiplex anidada HTLV-1 se destaca en la Figura 2.
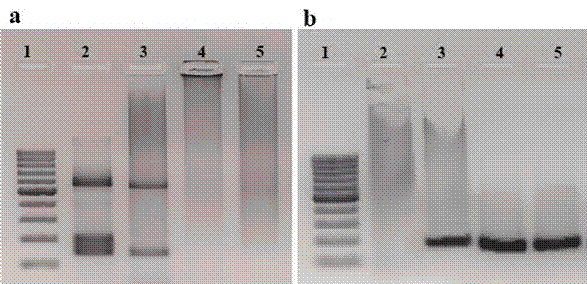
Figura 2. Electroforesis de la PCR múltiplex anidada para el diagnóstico de HTLV-1. a) Amplicones de 527 pb para LTR y 136 pb para pol HTLV-1. Carril 1: marcador de peso molecular 100 pb. Carril 2: control positivo MT2. Carril 3: control positivo muestra. Carril 4: control negativo K562. Carril 5: control negativo muestra. b) Amplificación de control endógeno β-globina de 150 pb. Carril 1: Marcador de peso molecular 100 pb. Carril 2: control blanco. Carril 3: control positivo MT2. Carril 4,5: muestras
Discusión
El flujograma de diagnóstico de HTLV-1 implica la realización de técnicas de diagnóstico serológico (ELISA, CLIA, ECLIA, IFI, IB o WB) no obstante estas metodologías producen una proporción considerable de resultados no concluyentes10, esto implica todo un desafío en países endémicos y sin políticas claras para el manejo, prevención y control de las infecciones por HTLV15, razón por lo cual se ha propuesto el uso de PCR debido a que son menos costosas en comparación de IB o WB2,9. En el presente estudio se estandarizó y evaluó una PCR múltiplex anidada, teniendo como target diagnósticos genes pol y LTR del genoma proviral los cuales son unas de las regiones más utilizadas para detección de ADN proviral de HTLV-110, otros ensayos emplean como genes confirmatorios env, tax, LTR para PCR anidada y pol para PCR cuantitativa11.
En el presente ensayo se determinó como límite de detección 0,5 ng/μL de ADN proviral usando la línea celular MT2 para genes pol y LTR, este límite es óptimo comparado con otra estandarización realizada en Brasil frente a pol y tax pero usando la línea celular C91-PL con un límite de detección 100 ng/μL para PCR convencional y 10 ng/μL para RT-PCR; si se utilizaba la línea celular BBF el límite fue de 1 ng/μL tanto en PCR convencional y RT-PCR10, esto indica que el límite de detección puede estar influenciado por las líneas celulares y por el tipo de PCR validado.
Respecto a la sensibilidad y especificidad encontrada, los resultados son semejantes a los obtenidos por Gonçalves et al.,11 quien reportó una sensibilidad del 97,4% y especificidad del 100% validando un múltiplex RT-PCR para HTLV-1, otras PCR también muestran resultados comparables a los de esta investigación10. Si bien es cierto se han validado PCR para confirmar simultáneamente HTLV-1/2 estas presentan sensibilidad baja (94,6 % para HTLV-1 y 78,6 % para HTLV-2) comparados con nuestro estudio16. Otros parámetros de validación que obtuvieron el 100% fueron repetibilidad, reproducibilidad y vpp. Si bien es cierto que de las 35 muestras positokjhivas testeadas solo 34 fueron positivas por PCR (vpn=97,2), es probable que este único resultado negativo por PCR se explique por la baja carga proviral17 presente en la muestra. Asimismo, el coeficiente kappa y el índice de Youden muestran que esta PCR tiene una fuerza de concordancia casi perfecta comparada al IB y un buen rendimiento como prueba diagnóstica.
Es muy importante mencionar que en este estudio se optó por una PCR múltiplex anidada puesto que, esta metodología comparada a otros tipos de PCR, incrementa tanto la sensibilidad como la especificidad para detectar HTLV-1, ya que en la segunda ronda se vuelve a amplificar el producto de la primera reacción, mientras que los cebadores internos sólo amplifican si en la primera reacción se logró obtener un producto específico18.
Haciendo una estimación preliminar, la presente prueba molecular confirmatoria para la detección de HTLV-1 costaría 21 dólares por ensayo, siendo prácticamente sólo el doble del costo de la prueba in house IFI-HTLV-1 (10,7 dólares por ensayo)5, pero 3,2 veces menor que IB comercial (67 dólares para cada ensayo)6 y 5,4 veces menor que el Western blot comercial (113 dólares por ensayo)7. En consecuencia, esta PCR validada se presenta como una buena opción para Perú, siendo que el diagnóstico molecular de esta enfermedad es más económico especialmente en países endémicos19.
Respecto a las limitaciones del ensayo, éste no cuantifica carga proviral, además al igual que Costa et al.,9 consideramos que, si bien esta metodología puede detectar al virus en muestras con resultados no concluyentes, este tipo de ensayos no eliminan la necesidad de las pruebas serológicas confirmatorias, sino más bien son ensayos complementarios. Otra limitación se encuentra en el número no tan robusto de muestras evaluadas, pues fue difícil la disponibilidad de muestras frescas sanguíneas estudiadas. Una última limitación fue que solo se evaluó la prueba para identificación de HTLV1 y no para HTLV-2, sin embargo, debemos destacar que la proporción de la distribución geográfica de estos virus en Perú es de 98% y 2% respectivamente20.
Finalmente, se concluye que la prueba PCR múltiplex anidada HTLV-1, es una buena alternativa para el diagnóstico fiable de este virus en muestras sanguíneas, presentando un alto rendimiento diagnóstico y debido a su bajo costo se recomienda su implementación en el flujograma del diagnóstico de HTLV-1 en Perú.