El Acinetobacter baumannii es considerado un patógeno nosocomial emergente, causante de infecciones en pacientes hospitalizados sometidos a catéteres, suturas, ventiladores, humidificadores, máquinas de diálisis y otros equipos1,2,3.
La patogenicidad de Acinetobacter baumannii se debe a dos características importantes, supervivencia por tiempos prolongados en condiciones adversas y facilidad de adquirir multirresistencia a los antibióticos. Esta bacteria ha sido causante de brotes hospitalarios en diferentes países, en Estados Unidos el 2003 con el regreso de militares de zonas como Irán y Afganistán se aislaron cepas de A. baumannii multidrogo y pandrogo resistentes1,3.
Datos epidemiológicos demostraron rápidos cambios de sensibilidad a resistente sobre carbapenémicos considerados el tratamiento ideal aumentando la estadía hospitalaria y los costos. Acinetobacter baumannii está asociada a la unidad de terapia intensiva por el uso de dispositivos médicos sobre los cuales se desarrollan biopelículas bacterianas1,4.
La biopelícula es una comunidad de bacterias asociadas irreversiblemente, protegidas por una capa de exopolisacáridos que permite adherirse a una superficie inerte o un tejido. Una biopelícula se establece como una estrategia de supervivencia bacteriana, un mecanismo de protección frente a fluctuaciones medioambientales5,6. De igual manera, Luo et al5 lo describe como un nido con constante intercambio de material genético responsable de nuevos genes asociados a la resistencia antimicrobiana5.
La biopelícula no está restringida a una sola especie bacteriana, por el contrario, varias especies constituyen una biopelícula, de igual manera se asocia a infecciones crónicas difíciles de eliminar en muchos casos provocando infecciones recurrentes6.
Acinetobacter baumannii ha demostrado ser un formador de biopelícula, esta bacteria tiene la facilidad de adherirse a dispositivos médicos y sobrevivir por tiempos prolongados frente a cambios de pH, temperatura y humedad7. Esta comunidad bacteriana facilita el intercambio de nutrientes y eliminación de desechos, permite soportar altas concentraciones de antibióticos por penetración lenta e incompleta, causas metabólicas que interfieren la acción de medicamentos (anaerobiosis, pH, etc.) e intercambio genético que potencia algunos mecanismos de resistencia7,8.
En nuestro país no existen estudios sobre biopelículas y los genes implicados en su producción, los estudios enfocados hasta la fecha se realizaron sobre otras bacterias como Staphylococcus aureus, Escherichia coli y Pseudomona aeruginosa 7,8. A partir de estas investigaciones se identificaron genes ompA, csuE y bap que juegan un papel en la formación de biopelículas, es importante el estudio de estos genes para describir mecanismos que permitan bloquear la producción de biopelícula para mejorar la acción del antimicrobiano7.
El objetivo principal del estudio fue cuantificar la cantidad de biopelícula producido y asociarlo a genes ompA, csuE y bap, identificar el gen asociado a una mayor producción de biopelícula, por último, relacionar la cantidad de biopelícula formada con la resistencia a los carbapenems 9,10,11,12.
Material y métodos
Se analizaron todas las cepas de Acinetobacter baumannii aisladas de dos centros hospitalarios de Cochabamba, Hospital Clínico Viedma (168) y Hospital Materno Infantil Germán Urquidi(HMIU) (23), durante el periodo 2016 - 2017.
Se obtuvo un total de 191 aislamientos de heridas de diferentes sitios anatómicos, hemocultivos, sonda Foley, catéteres, orina, aspirado bronquial, sonda endotraqueal y líquidos biológicos. Se realizó la identificación de Acinetobacter baumannii identificadas por PCR en tiempo real por detección de bla oxa-51, excluyendo aquellos aislamientos de que no presentaran el gen considerado un marcador de especie.
Determinación de la susceptibilidad antibiótica
Realizado en el laboratorio de Bacteriología del Hospital Clínico Viedma utilizando el método de difusión en disco de Kirby-Bauer, bajo criterios establecidos por National Committee for Clinical Laboratory Standards (CLSI), se probaron los siguientes antibióticos: Imipenem y Meropenem (10 ug), Ciprofloxacina (5 ug), Cefotaxima, Ceftazidima y Cefepime (30 ug), Gentamicina y Amikacina (30 ug), Trimetropin/sulfametoxazol (25 ug), Ampicilina sulbactam (10/10 ug), Piperacilina tazobactam (100/10 ug), Aztreonam (30 ug), Minociclina (30 ug), Levofloxacina(5ug).
Todos los antibióticos fueron sometidos a control de calidad interno de discos de sensibilidad a patógenos descritos en el manual M-100 S-10 que regula los puntos de corte de cada sensidisco estudiado, de igual manera el manual M-100 S-10 regula la interpretación de los resultados en sensible, intermedio o resistente de acuerdo a los rangos establecidos por la CLSI.
La susceptibilidad antibiótica se analizó por la técnica de Kirby-Bauer, se observó una alta resistencia en las 23 cepas provenientes del HMIU con un 100% de resistencia a Imipenem y Meropenem, Cefalosporinas, Monobactams, Quinolonas, Amikacina, Gentamicina y Sulfamidas. Para el Hospital Viedma de 168 aislamientos estudiados se encontró resistencia del 91,7% para ceftazidima y Cefepime, 93,4% sulfonamidas. Gentamicina 91%, piperacilina tazobactam 77,1%, amikacina 61,4%, Imipenem 61% y meropenem 61,3%. El resultado más bajo se determinó para colistina 0,6% (Figura 2).
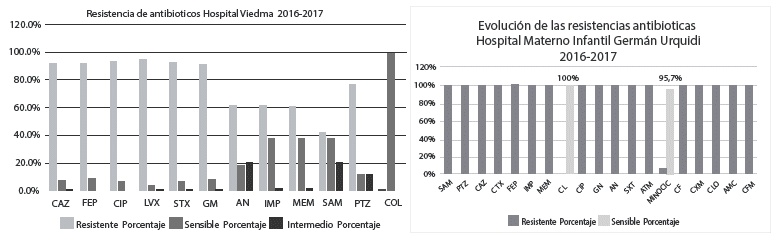
Figura 2: Perfil de resistencia a los antibióticos. A) Hospital Clínico Viedma. B) hospital Materno Infantil Germán Urquidi.
Extracción de ADN
Se realizó el método de hervido o boiling, a partir de cultivos bacterianos en medio Muller Hinton se tomó una asada de colonias de la bacteria, se suspenden en agua calidad biología molecular, posteriormente se somete a 100°C por 15 min, centrifugar a 12 000 rpm, separar el sobrenadante donde se encuentra ADN.
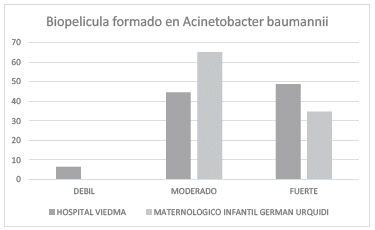
Figura 3: Clasificación del biofilm formado en A. baumannii para ambos centros hospitalarios de Cochabamba.
Identificación génica de Acinetobacter baumannii
La identificación de Acinetobacter baumannii por métodos moleculares se realizó por PCR (reacción en cadena de la polimerasa) en tiempo real se detectó bla oxa-51 denominado gen único o intrínseco identificado solo en esta bacteria a nivel del cromosoma.
Para detectar oxa-51 se llevó a cabo las siguientes condiciones: 1 ciclo inicial de 94°C 4 min, seguidos de 30 ciclos de 94°C 30s, 56°C 30s, 72°C 45s, un ciclo final 72°C 45s y una curva de melting 65°C 0,05s y 95°C 0,05s. Se utilizó el primer descrito en Tabla 1.
Detección de genes implicados en la formación de biofilm
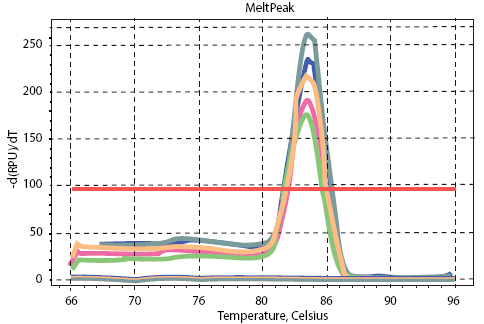
De acuerdo al método descrito por Badmasti y Azizi. Se realizó la identificación de bap, csuE y ompA en aislamiento de A. baumannii por PCR en tiempo real, los resultados obtenidos se observan en las curvas de fusión (Figura 1). Para el gen bap se utilizó los cebadores descrito por Badmasti et al13 que se indica en la Tabla 1, con las siguientes condiciones: un ciclo inicial de 94°C por 4 min, seguidos de 30 ciclos de 94°C por 30s, a 55°C por 30s, a 72°C por 60s, un ciclo final 72°C por 60s y una curva de melting 65°C por 0,05s y 95°C por 0,05s.
Para el gen csuE se utilizó el cebador indicado en la tabla 1, de acuerdo a Azizi et al14 para un amplificado de 168 pb, con las siguientes condiciones: un ciclo inicial de 94°C por 4 min, seguidos de 30 ciclos de 94°C por 30s, a 59°C por 30s, a 72°C por 60s, un ciclo final 72°C 60s y una curva de melting 65°C por 0,05s y 95°C por 0,05s.
Para el gen ompA se utilizó el cebador descrito por Smani et al10 que se indica en la Tabla 1, con las siguientes condiciones: 1 ciclo inicial de 94°C 4 min, seguidos de 30 ciclos de 94°C por 30s, a 56°C por 30s, a 72°C por 60s, un ciclo final 72°C por 60s y una curva de melting 65°Cpor 0,05s y 95°C por 0.05s.
Cuantificación de biofilm
Se disuelve una asada de colonia de A. baumannii en LB Broth con 0,25% por 24 horas, se realiza una dilución 1:50 en un nuevo medio LB Broth, depositar 200 ul de la dilución en pocillos de Elisa estériles e incubar por 24 horas, posteriormente desechar el sobrenadante y realizar 3 lavados con PBS, depositar 200 ul de cristal violeta 1% para teñir el biopelículas formado por 25 minutos, lavar 3 veces con PBS, resuspender con 200 ul de alcohol acetona (80:20) el cristal violeta fijada al biopelículas y leer al 630 nm (Figura 4).
Los resultados de densidad obtenidos se clasificaron en no formadores (DO≤Doc), débiles (Doc < DO ≤ 2 Doc), moderados (2 Doc < DO < 4DOc) y fuertes (> 4DOc).
Obteniéndose la densidad óptica de corte (Doc) de acuerdo a la siguiendo la fórmula Doc = DO +(3 * DS) 8,14.
El protocolo de investigación fue aprobado por el Comité de Ética del HMIGU y Hospital Clínico Viedma.
Resultados
Todas las cepas estudiadas fueron formadores de biopelículas, moderados 65,2% y 34,8% fuertes para el Maternológico German Urquidi, en cuanto al Hospital Viedma se registró 6,5% (11/168) débiles, 44,6% (75/168) moderados y 48,8% (82/168) formadores fuertes de biopelículas. Como se observa en la Figura 3.
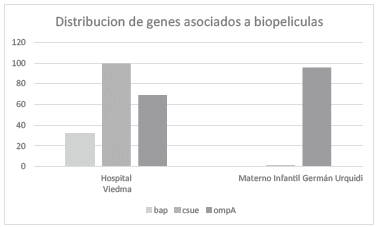
En cuanto al gen bap ninguna cepa del HMIGUlo presenta, para el hospital Viedma existe presencia en un 31,5% (53/168), como se observa en la figura 4. El gen bap no presenta relación con la cantidad de biopelícula formada de acuerdo al análisis estadístico.
El gen csuE es el gen de mayor presencia en el Hospital Viedma 99,4% (167/168) contrario al Maternológico Infantil 1,34% (1/23). Se ha demostrado que este gen no presenta asociación con la cantidad de biopelícula, pero si se encuentra en todos los aislamientos formadores de biopelículas (Figura 4).
El gen ompA se identificó en el 95,6% (22/23) para el HMIGU y 69% (116/168) para el Hospital Viedma, para este centro hospitalario se encontró una asociación significativa entre la cantidad de biopelícula y la presencia de este gen, resultado contrario al Materno Infantil (Figura 4).
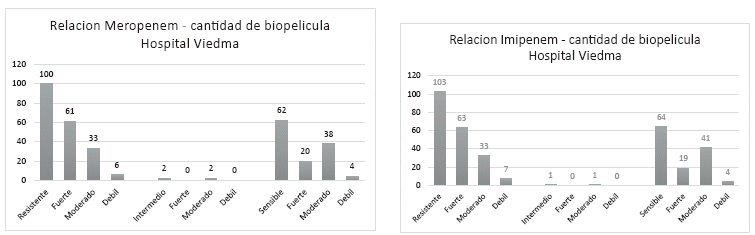
De igual manera el análisis estadístico de la resistencia de Imipenem y Meropenem frente a la cantidad de biofilm formado mostró una asociación significativa, formadores fuertes de biofilm influyen en la resistencia a ambos carbapenems (Figura 5).
Discusión
Este estudio permitió establecer un protocolo para cuantificar biopelículas y relacionarla con la cantidad formada, los genes implicados en su formación y la resistencia a los antibióticos. Se estandarizó la detección de genes bla oxa-51, bap, ompA y csuE por PCR en tiempo real a partir de aislamientos de Acinetobacter baumannii del Hospital Viedma y Materno Infantil Germán Urquidi.
La identificación de A. baumannii por reacción en cadena de la polimerasa (PCR) se basa en la presencia de bla OXA- 51 como demostró Turton et al 19 que puso a prueba la presencia de este gen frente a otras especies del género Acinetobacter y otras bacterias Gram negativas similares como, Stenotrophomona, Pseudomona, Moraxella y Burkhordelia, obteniendo excelentes resultados.
El alto porcentaje de resistencia a los carbapenémicos para el Maternológico (100%) y el Hospital Viedma (61% y 61,3%) son similares a los obtenidos por Vanegas et al3 que identifica a Sudamérica como el continente con más resistencia frente a Europa (25,7%) y Estados Unidos (12,1%), en Argentina se muestra resistencia de 84.9%, Brasil 71,4%, Chile 50%, Chávez et al15 describió en Colombia resistencias de 96,2% y 100% para Meropenem e Imipenem respectivamente, Ciello et al16 en Brasil describió un 66,1% similar a otros estudios.
En Bolivia el 2008 Fernández et al17 determinó resistencia 39,71% para carbapenems, similares resultados obtuvieron Sevillano et al17 en 2008-2009 con 35% de resistencia al Imipenem, en 2014 Parra et al18 describió resistencia de 38% y 39% para Imipenem y Meropenem, resultados diferentes a nuestro estudio. Sin embargo, el 2015 Fernández et al17 demostró aumento significante en la resistencia de los carbapenémicos a 95,1% y se encontró un aislamiento con resistencia a Colistina lo que demostró el comienzo de alta resistencia.
El enfoque principal de este trabajo radica en la biopelícula producida por A. baumannii, se estudió la cantidad y los genes asociados a su formación, resistencia asociada a Imipenem y Meropenem.
Gómez et al 20 el 2012 clasificó la cantidad de biopelícula de varias bacterias Gram negativas, de acuerdo a la técnica descrita por Stephanovic et al8, siendo A. baumannii una bacteria no formadora de biopelícula (60%), diferente a nuestro estudio donde todas las cepas de A. baumannii son formadores de biopelícula, más del 90% entre moderados y fuertes.
La asociación de CsuE y la cantidad de biopelícula formada no presentan relación de acuerdo a los resultados de chi cuadrado, este gen está implicado en la formación de pilis. La adherencia del pili a la superficie es el primer paso para la formación de biopelícula y no se relaciona con la cantidad o espesor formado, lo que explica la amplia distribución y que todas las cepas estudiadas sean formadoras de biopelícula sin importar la cantidad, Breij et al21 demostró que su mutación afecta la formación de pilis, produciendo pilis más cortos y delgados afectando la formación de biopelícula.
En A. baumannii el gen OmpA presenta una distribución extensa, Liu et al22 el 2013-2014 describió un 100% de presencia un resultado similar a Badmasti et al13, similares resultados al HMIGU (95%) y menor cantidad para el Hospital Viedma (69%).
Ma et al23 en 2008 describió el efecto de la delección de ompA en E. coli produciendo la disminución de la cantidad de biopelícula de 10 a 20 veces. El resultado obtenido en el estudio para A. baumannii de manera similar demostró para el Hospital Viedma que existe la asociación de este gen con la cantidad de biopelícula formada.
En cuanto al gen bap identificado por primera vez en Staphylococcus aureus presenta estudios como Cucarella et al 24 quien describió la importancia de este gen en la formación de biopelícula. Badmasti et al 13 describió una distribución del 30% del gen bap en cepas de Acinetobacter baumannii, un resultado diferente describió Luo et al 5 84% del gen bap en A. baumannii.
El estudio de biopelícula en A. baumannii es de gran interés por la resistencia a los antibióticos que afrontamos en la actualidad, sin embargo, en la actualidad existen pocos estudios dedicados a la cuantificación de biopelícula en esta bacteria.
Srinivasa et al 25 demostró la correlación entre la cantidad de biopelícula con la multirresistencia a Imipenem y Meropenem, un resultado similar al que obtuvimos demostrando que cepas formadoras fuertes de biopelícula se asocian con la resistencia a carbapenems en cepas de Acinetobacter baumannii.