Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Gaceta Médica Boliviana
Print version ISSN 1012-2966On-line version ISSN 2227-3662
Gac Med Bol vol.29 no.2 Cochabamba 2006
TRABAJO ORIGINAL
DETECCION Y DIFERENCIACION DE ENTAMOEBA HISTOLYTICA / ENTAMOEBA DISPAR MEDIANTE REACCION EN CADENA DE LA POLIMERASA (PCR) USANDO DNA EXTRAIDO DE QUISTES PRESENTES EN MUESTRAS DE HECES FECALES HUMANAS
* Carmen Patricia Montaño Almendras,** Jean Pierre Herveg,* Mercedes Iriarte
* Laboratorio de Microbiología UMSS
** Universidad Catolica de Lovaina, CIUF. Laboratorio de Inmuparasitologia chagas
RESUMEN
Está establecido que existen dos especies distintas de amebas que originalmente fueron conocidas como Entamoeba Histolytica. Ellas son E. dispar (forma no patogénica) y E. histolytica (forma patogénica). La diferencia entre estos dos organismos es de gran importancia clínica desde que son morfológicamente indistinguibles. El diagnóstico diferencial de estas dos especies es esencial para la decisión del tratamiento y la salud pública. Se puede usar un método rápido de extracción de DNA directamente de especímenes suspendidos en formalina éter. La extracción de ADN fue usada para la identificación de las especies existentes en las herramientas por reacción de cadena polimerasa (PCR). Un total de 75 muestras recolectadas aleatoriamente fueron analizadas. Despues de la confirmación por PCR: 7/75 muestras resultaron positivas para E. histolytica, 60/75 muestras resultaron positivas para E. dispar y 8/75 resultaron negativas porque no amplificaron, pudiendo tratarse de otras amebas como E. hartmani. E. iodoamoeba, E. coli, etc. Con estos resultados podemos darnos cuenta de la magnitud de falsos positivos que se generan con los exámenes microscópicos. Estas observaciones implican que el uso del DNA extraído directamente a partir de concentración de quistes para amplificación por PCR, es una herramienta útil para obtener un diagnóstico sensitivo y preciso que puede ser aplicado incluso en epidemiología.
Palabras claves: Entamoeba histolytica, Entamoeba dispar, reacción en cadena de polimerasa.
ABSTRACT
It has been established that two distinct species exist within what was originally known as Entamoeba histolytica. These are E. dispar (nonpathogenic form) and E. histolytica (pathogenic form). Differentiation of these two organisms is of great clinical importance since they are morphologically indistinguishable. Differential diagnosis of this two species is essential for treatment decision and public health knowledge. A simple and rapid DNA-extraction method that can be used directly on formalin-ether stool specimens. The extracted DNA was used for the identification of the species existing in the stools by polymerase chain reaction (PCR). A total of 75 randomly collected stool sample were analyzed. The samples analyzed by microscopic were 75, after confirmation by PCR: 7/75 samples resulting positive for E. histolytica, 60/75 samples resulting positive for E. dispar and 8/75 resulting negative because didn't amplify be able to be other amoebas like E. hartmani, E. iodoamoeba, E. coli, etc. With these results we can realize the magnitude of false positive that are generated with the microscopic exams. These observations imply that the use of the DNA extracted directly of the concentrate of cysts for PCR amplification is a useful tool for obtaining a sensitive and accurate diagnosis that can be applied even in epidemiology.
Key words: Entamoeba histolytica, Entamoeba dispar, polymerase chain reaction (PCR)
INTRODUCCION
Estrictamente debe denominarse amebiasis a la enfermedad producida en seres humanos por amebas de la especie Entamoeba histolytica. Esta definición excluye de forma implícita otras situaciones que pueden ser confundidas con esta entidad. En primer lugar, no debe considerarse amebiasis las infecciones por otras especies de amebas parasitarias (por ejemplo. Entamoeba coli, Entamoeba polecki, Entamoeba hartmanni. Iodamoeba butschlii), ya que no ocasionan enfermedad (Clark, et al., 2000). Sin embargo, su detección en heces es un dato de interés, ya que indica el consumo de agua o alimentos contaminados. Por otro lado, las amebas de vida libre como Naegleria fowleri, varias especies de Acanthamoeba y Balamuthia mandrillaris dan lugar a cuadros clínicos muy diferentes de la amebiasis, con una patogenia y una clínica específica (meningoencefalitis primaria amebiana, encefalitis granulomatosa amebiana y queratitis por Acanthamoeba) (Tanyuksel, et al., 2003).
Así, la primera identificación de E. histolytica fue realizada por Fedor Losch en el año 1875, aunque la primera descripción taxonómica formal de E. histolytica fue realizada por Fritz Schaudinn en 1903 (Clark, et al., 2000). En 1925, Emile Brumpt propugnó la existencia de dos especies similares morfológicamente pero diferentes desde el punto de vista clínico: E. histolytica, capaz de ocasionar enfermedad en seres humanos y E. dispar sin capacidad patógena (Clark, et al., 2000). Desde entonces y hasta hace menos de una década, se ignoró esta interpretación, aunque fueron acumulándose un gran número de datos (epidemiológicos, patogénicos, clínicos, diagnósticos y terapéuticos) atribuidos a la infección por E. histolytica (Tannich et al .,1991). Sin embargo, en 1993, la consideración de varios tipos de datos llevó a una nueva descripción de la existencia de ambas especies de amebas y, por lo tanto, a la necesidad de reinterpretar los datos disponibles y realizar nuevos estudios.
Recientemente investigadores del instituto científico (TIGR) en Rock Ville, Maryland y del Instituto Wellcome Trust Sanger en el Reino Unido (2005) completaron la secuencia de Entamoeba histolytica. El genoma de la ameba tiene 92 genes bacterianos. Probablemente por haber fagocitado bacterias intestinales durante toda su vida evolutiva en el tracto digestivo. La mayoría de estos genes están involucrados en vías metabólicas y presumiblemente le sirva a la ameba para usar vías metabólicas bacterianas que le permitan adaptarse a las bajas concentraciones de oxígeno del intestino, así podría prescindir de las organelas que tienen el resto de las eucariontes, también vieron que presentaba una complejidad genómica inesperada, con casi 10.000 genes (Loftus B, et al. 2005).
Los genes de E. histolytica que codifican la subunidad pequeña del ARNr son más abundantes que otros. Estos genes se encuentran en un plasmido circular (no en el núcleo): y tienen un tamaño de 25 kb y presentan cerca de 200 copias por célula (Ramachandran. S.et al., 1993).
El objetivo de este trabajo fue la detección y diferenciación genética de Entamoeba histolytica con Entamoeba dispar, mediante reacción en cadena de la polimerasa (PCR) usando DNA extraído directamente de los quistes presentes en muestras de heces fecales humanas.
MATERIALES Y METODOS
La recolección de muestras de heces fecales se realizó durante 3 meses, de Marzo a Mayo de 2005, de diferentes Laboratorios de Cochabamba: el Laboratorio de parasitología de la Caja Petrolera, Laboratorio de parasitología de la Unidad de Saneamiento Ambiental, Laboratorio de parasitología y análisis clínico del Hospital Viedma y el laboratorio de la Escuela Técnica de Salud Boliviano Japonés. Posteriormente, se realizó un nuevo examen parasitológico directo en el Laboratorio de Microbiología del Centro de Aguas y Saneamiento Ambiental de la Universidad Mayor de San Simón.
La recolección de muestras para este estudio se hizo sin tomar en cuenta la edad ni el sexo de las personas.
Diagnóstico parasitológico
La identificación morfológica del parásito fue realizada por dos técnicas coproparasitológicas: 1. examen parasitológico directo y 2. Por la técnica de Ritchie modificada para la concentración de quistes (Botero et al . 2003). Posteriormente este concentrado fue alicotado en tubos eppendorff y guardado a -20°C hasta su uso.
Extracción del DNA
Para la extracción del DNA, se realizó una centrifugación de 200 μl del concentrado, luego se incubó a 56ºC por 2 horas con Dodesil sulfato de Sodio (SDS) y Proteinasa K. El pellet fue lavado con 1 ml del buffer fosfato salino. Después de la centrifugación el pellet fue resuspendido y centrifugado en 500μl de PBS+ Polivinilpirolidon al 2% (PVPP) (Sigma). Posteriormente fue nuevamente resuspendido en 500 μl de PBS+ Polivinilpirolidon al 2% y se llevó a ebullición a 100°C durante 10 min. Luego se incubó a 56°C por 2 horas con Dodesil Sulfato de Sodio (SDS) y Proteinasa K, seguidamente la extracción del DNA total de los quistes se realizó con el kit comercial Quiamp DNA Blood Mini Kit. La concentración de ADN de las muestras se midió por espectrofotometría y se almacenaron a -20ºC hasta su uso.
Diseño de cebadores
Para buscar el par de cebadores que sean complementarios a esta secuencia codificante además de que contengan un sitio de restricción, se utilizó el BLAST (Basic Local Alignment Search Tool)(http://www.ncbi.nlm.nih.gov/BLAST/), y (http://frodo.wi-mit.edu/cgibin/primer3/primer3_WWW.CGI).
Así se eligieron los siguientes cebadores:

Cuadro N° 1. Cebadores Sens y Antisens diseñados.
Estandarización de la Reacción en Cadena de la Polimerasa.
Después de sucesivas pruebas para la estandarización de la PCR se definieron los siguientes parámetros: Se aplicó la variante hot Start PCR, la temperatura hot Start para la desnaturalización inicial fue de 94ºC por 2 minutos en el primer ciclo, seguida de 39 ciclos, en las cuales se encuentran ciclos de desnaturalización de 94°C por 1 minuto, hibridación de los cebadores a una temperatura de 64°C por 1 minuto y la extensión a 72°C durante 1 minuto.
Amplificación
La reacción de amplificación se realizó en tubos eppendorf estériles que contenían: 5 μl de ADN (100 ng), l0mM de desoxinucleótido trifosfato (dNTPs), 10 U de Taq polimerasa, 3 μl de cada uno de los cebadores, 2.5 μ1 de la solución tampón de amplificación 10X, Agua destilada libre de nucleasas (Promega Madison W U.S.A). Todas estas cantidades fueron calculadas para 25 μl de la solución total. Los productos amplificados fueron mantenidos a 4°C hasta su uso.
La eficacia de la reacción de amplificación se comprobaba mediante electroforesis horizontal (Mini-Sub-Cell. GT System, BioRad), en gel de agarosa al 2% en tampón TAE 1X (TAE pH 8,5: tris base 5,4%. Acido acético; 0,5 M EDTA- Sigma), a un voltaje constante de 50 V durante 1 hora. En cada análisis electroforético fue incluido un patrón o marcador molecular 1000 pb (Promega G210A), con el objeto de comprobar el tamaño y la posible existencia de más de un fragmento amplificado.
El gel fue teñido con bromuro de etidio (Sigma) (0,5 μg/ml p/v) y visualizado con luz ultravioleta.
Hidrólisis Enzimática del Fragmento Amplificado de Entamoeba histolytica.
Una vez obtenidos los productos amplificados mediante la PCR se prosiguió con la hidrólisis de los mismos con el fin de diferenciar las secuencias específicas amplificadas para E. histolytica y para E. dispar. La hidrólisis de estos productos amplificados se llevó a cabo con la enzima endonucleasa Bg1 II que reconoce sitios con secuencias AGATCT, esta región de corte solo se encuentra en los productos amplificados de E. histolytica y no así en los productos amplificados de E. dispar, así podemos diferenciar a estos dos parásitos protozoarios.
El procedimiento seguido fue el siguiente: Los productos amplificados fueron incubados con 1 μl de Bgl II (Promega, R 6081), 10 U/μl. Para esto se usó 10μl del producto amplificado, 2 μl del tampón D, 7 μl de agua destilada libre de nucleasas, y fue incubado a 37ºC por 2 horas.
Separación Electroforética de los productos PCR e hidrolizados.
Para la identificación de los productos de la reacción en cadena de la polimerasa y los tratados con la endonucleasa de restricción Bgl II, se realizó una electroforesis horizontal junto con el marcador de peso molecular (PM), de acuerdo al siguiente procedimiento:
- Preparación del gel: Un volumen de agarosa (Promega Corporation), al 2% en tampón TAE 1X con bromuro de etidio (Sigma), fue depositado sobre la bandeja portageles, colocada sobre una superficie nivelada, y antes de la gelificación se inserta el peine para formar los correspondientes pocillos.
- Preparación de las muestras:
Los productos PCR y los productos amplificados hidrolizados con la enzima Bgl II. Fueron diluidos (5:1, v/v) con Azul de Bromofenol y depositados en los pocilios del gel que estaba sumergido en tampón TAE 1X, en el interior de la cubeta de electroforesis.
- Separación electroforética y visualización:
Las muestras fueron sometidas a migración electroforética con un voltaje constante de 50 V durante 1 hora, momento en que el azul de bromofenol había alcanzado el extremo del gel, tras lo cual, el gel fue depositado sobre el transiluminador de luz ultravioleta. El tamaño de los fragmentos de ADN fueron estimados por las distancias de migración del marcador de peso molecular 1 Kb y la imagen fue fotografiada utilizando una cámara digital.
Análisis e Interpretación
El tamaño del producto amplificado sin restricción es de 288 pb, que pertenece a la secuencia codificante de la subunidad pequeña de ARN ribosómico (SSU rRNA) para E. histolytica y para E. dispar. Cuando se realiza el corte con la Enzima de restricción Bgl II se obtienen dos fragmentos de 98 pb y de 198 pb respectivamente para E. histolytica. Cabe recalcar que el corte no se realiza en el producto amplificado de E. dispar porque esta enzima de restricción no reconoce ninguna región de esta secuencia.
RESULTADOS Y DISCUSION
De las 108 muestras de heces fecales recolectadas para este estudio, solamente en 80 se encontraron quistes E. histolytica y/o E. dispar, mediante la técnica de Ritchie modificada 78/108 muestras que contenían quistes de E. histolytica y/o E. dispar.
El alineamiento genómico de E. histolytica y E. dispar reveló un 2% de diferencia, presentando un 98% de similitud a nivel de la secuencia codificante para la subunidad pequeña del RNAr. Estos parásitos también muestran un rango similar de organización y biología celular.
Especificidad de los Cebadores
Para analizar la especificidad de los cebadores se trabajó en forma paralela con DNA de Entamoeba invadens y DNA de E. histolytica en diferentes diluciones, no habiéndose presentado diferencias significativas en cuanto a esta variable. Como se ve en la figura Nº 2 carril 5 y 6, los cebadores no amplificaron para E. invadens, pero hubo una buena aplificación para Entamoeba histolytica. Con esta prueba y con el tamaño del producto amplificado obtenido, nos aseguramos que los cebadores diseñados sensSSU y antisSSU son específicos para E. histolytica (Figura 1).
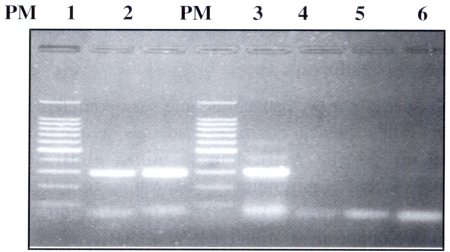
| Figura Nº 1. Prueba de especificidad de los cebadores sensSSU y antisSSU. Estos cebadores amplifican la secuencia codificante de la subunidad pequeña del ARNr iSSU ARNr). EL producto amplificado tiene un tamaño de 288 pb. PM: Marcador de peso molecular, carril I: control positivo DNA E. histolytica dilución 1/100, carril 2: control positivo DNA E. histolytica dilución 1/75. PM: marcador de peso molecular, carril 3: control positivo DNA E. histolytica dilución 1/50, carril 4: control negativo, carril 5: DNA E. invadens . carril 6: DNA E. invadens. |
El uso de cebadores que reconocen regiones específicas de la subunidad pequeña del RNAr ha sido muy útil, mostrando una mayor sensibilidad en relación a otros cebadores que reconocen otros sitios específicos del genoma de E. histolytica y E. dispar (Mirelman D.,et al. 1997).
Amplificación del DNA extraído a partir de muestras de heces fecales.
Para la amplificación de las muestras se usaron los cebadores diseñados: sensSSU y antisSSU: específicos para E. histolytica y E. dispar respectivamente. El fragmento amplificado tiene un tamaño de 288 pb (Figuras N° 2 y N° 3). Se analizaron 75 muestras.
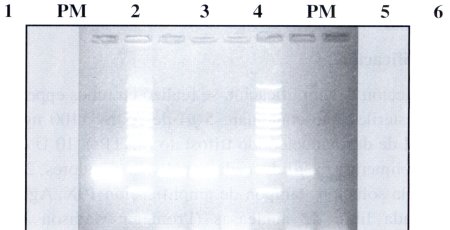
| Figura Nº 2. Resultados PCR. Productos amplificados obtenidos con los cebadores sensSSU y antisSSU: carril I: control positivo, PM marcador de peso molecular, carril 2: muestra A. carril 3: muestra B. carril 4: muestra C. PM: marcador de peso molecular, carril 5: muestra D, carril 6: control negativo. |
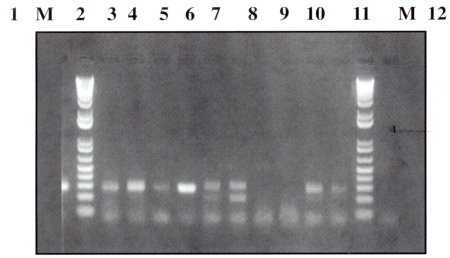
| Figura Nº 3. Resultados PCR Productor amplificadas obtenidos con los cebadores sensSSU y antisSSU: carril I: control positiv.; PM: marcador de peso molecular, carril 2: muestra F. carril 3: muestra G, carril 4: muestra H. carril 5: muestra I, carril 6: mustia J. carril 7: muestra K, carril 8: muestra L, carril 9: muestra M, carril 10: muestra N, carril 11: muestra N, PM: marcador de peso molecular, carril 12: control negativo. |
Como vemos en la figura Nº 4. los carriles 2, 3, 4, 5, 6, 7. 10 y 11 resultaron positivos para E. histolytica y/o E. dispar, mientras que en los carriles 8 y 9 no se produjo amplificación, por tanto no se trata de E. histolytica ni E. dispar. Entonces podemos decir que durante el examen microscópico se produjeron equivocaciones, pudiendo tratarse de otras amebas como Entamoeba coli, Entamoeba hartmanni, etc.
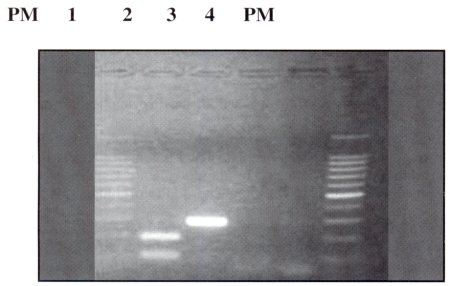
| Figura Nº 4. Resultados de la hidrólisis con la enzima Bgl II : PM: marcador de peso molecular, carril 1: DNA de F. histolytica hidrolizada, carril 2: DNA de F. histolytica sin hidrólisis: carril 3: control negativo, carril 4: control negativo sometido a hidrolisis, PM: marcador de peso molecular I kb ( promega). |
Patrones obtenidos tras la restricción enzimática con Bgl II.
La hidrólisis de los Productos amplificados se hicieron con el objetivo de diferenciar E. histolytica y E. dispar, la hidrólisis solo se produce en los productos amplificados de E. histolytica y no así en los productos amplificados de E. dispar, obteniéndose dos fragmentos uno de 190 pb y el otro de 98 pb (Figura Nº4).
Evaluación del total de casos infectados con E. histolytica o E. dispar.
Las muestras positivas para E. histolytica y/o E. dispar mediante microscopía fueron 75, después de la confirmación mediante reacción en cadena de la polimerasa (PCR) basandose en la amplificación específica de la secuencia codificante de la subunidad pequeña del ARNr (SSU rRNA), se obtuvo los siguientes resultados: - 7/75 muestras resultaron positivas para E. histolytica, 60/75 resultaron positivos para E. dispar y 8/75 muestras resultaron negativas porque no amplificaron, pudiendo tratarse de otras amebas como E. hartmani, E. iodoamoeba, E. coli, etc. Con estos resultados podemos darnos cuenta de la magnitud de falsos positivos que se generan con los exámenes microscópicos (Cuadro Nº 2).
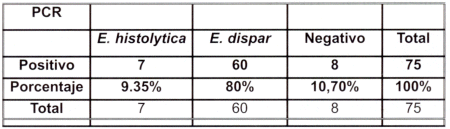
| Cuadro Nº 2. Comparación de los resultados microscópicos con la reacción en cadena de la polimerasa (PCR). El resultado microscópico de las muestras con quistes de E. histolytica y/o E. dispar es de n= 75. |
DISCUSION
Estos resultados muestran que las infecciones por E. dispar tienen mayor porcentaje en relación a las infecciones por E. histolytica. Estos datos concuerdan con información encontrada en estudios epidemiológicos realizados mediante PCR, en el norte de Filipinas, donde también encontraron un mayor número de personas infectadas por el parásito comensal E. dispar. (Rivera W., et al. 1998). Por lo tanto se sugiere que la PCR debería ser usada como test de diagnóstico para diferenciar a estas dos especies y de esta manera contribuir a la toma de decisiones del médico para el tratamiento del paciente infectado con E. histolytica o E. dispar, evitando de esta manera medicar a las personas que no necesitan tratamiento, conociendo además que el tratamiento es costoso y tóxico.
En este estudio se demuestra la confiabilidad y la sensibilidad de la PCR, usando DNA extraído directamente a partir de muestras de heces fecales, esta confiabilidad de la PCR también ha sido reportada por muchos estudios (Rivera W. et al. 1996.: Srivastava S., et al. 2005.; Mijak P., et al. 2000). La confianza en solamente el examen microscópico a menudo es engañoso, un tamaño pequeño y variabilidad en los estados del ciclo de vida hace que el diagnóstico sea dificultoso en relación a la infección por Entamoebas de diferentes especies. Estudios comparativos donde se usaron el ELISA y la PCR para realizar la diferenciación de E. histolytica y E. dispar indican que la PCR fue mas útil, sensible y especifica que el ELISA (Rivera W., et al. 1998.; Gonin R, et al. 2003.; Mirelman D., et al ,1997). Desde que E. dispar tiene el mismo nicho ecológico, ciclo celular y mecanismos de transmisión que E. histolytica, también es importante el estudio de este organismo (Ramos F, et al., 2005). Hay evidencias de que E. dispar es capaz de producir lesiones intestinales en animales (Espinosa C. et al. 1997), destruyendo la monocapa de las células epiteliales "invitro "(Furst. et al,2002.; Pinheiro.et al.2005).
CONCLUSIONES
Los resultados descritos anteriormente nos han permitido extraer las siguientes conclusiones: La aplicación de herramientas informáticas tales como BLAST (Basic Local Alignment Search Tool), FASTA de Pud Med y FRODO, nos permitió ver la existencia de un 98% de similitud entre ambas secuencias genómicas (E. histolytica y E. dispar). El par de cebadores diseñados sensSSU y antisSSU son específicos para ambos parásitos (E. histolytica y E. dispar) y amplifican la secuencia codificante para la subunidad pequeña del RNAr (SSU-RNAr), el tamaño del producto amplificado es de 288 pb.
La hidrólisis enzimatica con la enzima de restricción Bgl II nos permitió diferenciar la cepa patógena (E. histolytica) de la no patógena (E. dispar). La enzima Bgl II reconoce un sitio específico del fragmento amplificado de E. histolytica y corta el fragmento en dos: resultando un fragmento grande de 198 pb y otra pequeña de 98 pb.
Este estudio nos muestra una clara utilidad de la PCR para diferenciar E. dispar y E. histolytica directamente a partir de las heces fecales. En beneficio adicional puede ser usado para una monitoreo y eficaz tratamiento de los pacientes, el cual es usualmente limitado por pruebas serológicas por la persistencia de la respuesta de anticuerpos aun después de sucesivos tratamientos.
Agradecimientos
Agradecemos al Laboratorio del Centro de Aguas y Saneamiento Ambiental, y al Laboratorio de Inmuno-parasitología Chagas.
BIBLIOGRAFIA
1. Botero D, Restrepo M. 1998. Amebiasis. Parasitosis Humanas Cuarta edición. Medellín: Corporación Investigaciones Biológicas. 30-58. [ Links ]
2. Clark CG, Espinosa Castellano M, Bhattaeharya A. 2000. Entamoeba histolytica: An overview of the biology of the organism. En: Ravdin JI. editor Amebosis. Londres: Imperial College Press. 37-45. [ Links ]
3. Espinosa Castellano M.Castañon G, Martínez Palomo A.. 1997. In vivo pathogenesis of Entamoeba dispar. Arch. Med. Res. 28: 204-206.
4.Furst C, Gomes M.A.Tafuri W.L. Silva E. 2002. Biological aspects of a Brazilian strain of Entamoeba dispar. Pathologica. 94: 22-27.
5. Gonin P. and Trudel Louise. 2003. Detecction and differentiation of Entamoeba histolytica and Entamoeba dispar Isolates in Clinical Samples by PCR and Enzyme-Linked Immunosorbent Assay. Journal of Clinical Microbiology. 41: 237-241.
6. Loftus Brendan, Anderson lain, Robo Davie, Alsmark M, Samuelson , Amedeo Paolo, Roncaglia Paola, Mate Berrimar, Hirt Roberto, Mann J. Barbara, Nozaki Tomo, Suh Bernrd, Cecilia De Mihai, Egbert Tannich, Matthias Leippe, Margit Hofer. 24 de febrero 2005. El genoma del Parásito protista Entamoeba histolytica. Naturaleza 433: 865-868.
7. Mijak Przemyslaw. Kur José, Pietkiewocz Halina, Kotlowski Andrzej.Nahorski Waclaw, Szostakostakowska Beata. 2000. Molecular Differentiation of entamoeba histolytiea and Entamoeba dispar from Stool and Culture Sample Obtained from PolishCitizens infected in Tropics and in Poland. Acta protozoologia. 39: 217-224.
8. Mirelman D. Nuchamowitz Y. Stolarsky T, 1997. Comparison of enzymc-linkcd immunosorbent assay-based kits and PCR amplification of rRN A genes for simultaneous detection of Entamoeba histolytica and E. dispar. J Clin Microbiol 5: 2405- 2407.
9. Pinheiro Sandra M, Maciel Rogérico F. Moráis Marcos A. Acá, Ivanize S, Carvalho Luiz B. Coimbra Maria R. 2005. Genetic charaterization of Entamoeha dispar isolates in Northeast Brazil. Science direct 70: 35-40.
10. Ramachandran S. Bhattaeharya A. Bhattaeharya S. 1993. Nucleotide sequenee analysis of the rRN A transcription unit of a pathogenic Entamoeba histolytica strain HM-1: IMSS. Oxfort University Press. Nucleic Acids Research. 21: 2011.
11. Ramos F, Garda G. Valadez A. Moran P, González E. Gómez A, Melendro E. ValenzuelaO.XiménezC. 2005. análisis of polymorphism as a toll for study of geographic distribution. Molecular and Biochemical parasitology. 141:175- 177.
12. Rivera Wilndell L, Tachibana Hiroshhi. Mary rose Angnes Silva Tahat, harki Uemura Hiroji Kanbara. 1996. Differentiation of Entamoeba histolytiea and E. dispar DNA from cyst present in stool specimens by polymerase chain reaction: its field application in the Philippines. Parasiton Res 82: 585-589.
13. Rivera Windell L. Tachibana Hiroshi, Kanbara. 1998. Fiel study or the distribution of Entamoeba histolytica and Entamoeba dispar in the northern Philippines as Detected by the polymerase Chain Reaction University Nagasaki Japan. Departamento de Protozoologia. 59: 916-921.
14. Srinvastava Shweta, Bhattaeharya Sudha, Paul Jaishree Paul. 2005 Species and strain especific probes derived from repetitive DNA for distinguishing Entamoeba histolytica and Entamoeba dispar. Experimental parasitology. 110: 303-308.
15. Tannich E, Burchard GD. 1991. Differentiation of pathogenie from nonpathogenic Entamoeba histolytica by restriction fragment analysis of a single gene amplified in vitro. J Clin Microbiol. 2: 250-255.
16.Tanyuksel Mehmet. Petri William A. 2003. Laboratory Diagnosis of Amebiasis. Clinical Microbiolouv Reviews. 713-729.