Introducción
La resistencia a los antibióticos es la capacidad de los microorganismos para resistir los efectos de los antibióticos, una capacidad que las bacterias adquieren durante la infección (1).
La Organización Mundial de la Salud (OMS) estima que para 2050, la resistencia bacteriana causará 10 millones de muertes (2). Como parte de un plan de acción mundial sobre la resistencia a los antimicrobianos, propone la creación de una red de laboratorios dedicados a conservar las cepas y optimizar el uso de los antimicrobianos (1).
Dentro de las infecciones comunes que son causadas por bacterias multirresistentes están las infecciones del tracto urinario (ITU). La mayoría de estas infecciones son causadas por miembros de la familia Enterobacteriaceae, principalmente por Escherichia coli con el 69-90 % de los casos, seguida por Klebsiella spp. (4). Estas bacterias forman parte del microbiota intestinal, sus factores de virulencia les permiten adherirse, colonizar y migrar al tracto urinario (6).
La aparición y rápida propagación de microorganismos resistentes a las cefalosporinas de tercera generación (cefotaxima, ceftazidima y ceftriaxona), como Escherichia coli (E. coli) y Klebsiella pneumoniae (K. pneumoniae), se considera un problema de salud pública a nivel mundial. La resistencia a ellos se debe principalmente a la producción de betalactamasas de espectro extendido (BLEE). Las BLEE son enzimas transportadas por elementos genéticos móviles que a su vez contienen genes de resistencia a otras clases de antibióticos (aminoglucósidos, fluoroquinolonas y trimetoprim sulfametoxazol) (7).
Las betalactamasas de espectro extendido (BLEE) y betalactamasas de espectro ampliado (BLEA), son enzimas producidas por bacilos Gram negativos como las enterobacterias, comúnmente producidas por Escherichia coli y Klebsiella pneumoniae. Estas enzimas son capaces de inactivar las penicilinas y cefalosporinas de primera y segunda generación (8).
Debido a lo expuesto, la presente investigación tiene como objetivo determinar perfil de resistencia antimicrobiana de Escherichia coli y Klebsiella pneumoniae en pacientes que acudieron al Hospital del Norte, Cochabamba - Bolivia.
Metodología
Se trata de un estudio descriptivo, no experimental, de corte transversal. La población estuvo conformada por 134 pacientes que ingresaron con diagnóstico de infecciones urinarias y otras infecciones al Hospital del Norte, ubicado al norte de la ciudad de Cochabamba, Bolivia. Se incluyeron muestras de orina, esputo, punta de catéter, aspirado traqueal, secreción vaginal, líquido abdominal, absceso, secreción faríngea y secreción escrotal, provenientes de pacientes de ambos sexos y que fueron atendidos entre los meses de diciembre 2022 - abril 2023. Los criterios de inclusión fueron: Pacientes con orden médico, que tengan datos completos (sexo, edad), muestras que estén identificadas (Orina, esputo, secreción de herida, etc.), además de ser identificadas según el método de recolección de la muestra en el caso de orina (chorro medio, por sonda o punción suprapúbica) y cultivos con desarrollo de colonias pertenecientes a E. coli y K. pneumoniae. Sin embargo, se excluyeron a: Muestras no identificadas, visiblemente contaminadas, envases no adecuados, mal tapados o sin tapa; pacientes que estén recibiendo algún tipo de tratamiento con antibióticos y cultivos con otros tipos de bacterias.
Para el aislamiento e identificación de microorganismos (E. coli y K. pneumoniae) se emplearon medios de cultivos como el Agar Sangre, Mac Conkey, Eosina azul de metileno (EMB), pruebas bioquímicas como el agar Hierro-triple azúcar (TSI), Lisina hierro (LIA), Motilidad-indol-ornitina (MIO), UREA y CITRATO.
La sensibilidad y resistencia a los antibióticos in vitro se determinó mediante el método de placa de difusión en agar. Se emplearon antibióticos como la amoxicilina/ácido clavulánico, cefazolina, cefoxitina, ceftazidima, imipenem, meropenem, amikacina, nitrofurantoina, ciprofloxacina, ácido nalidíxico, levofloxacina, ampicilina, aztreonam, cefotaxima, cefepime, gentamicina y tetraciclina.
Se empleó como instrumento el cuaderno de registros de laboratorio y una ficha de recolección de datos elaborada para la investigación. Los datos obtenidos se almacenaron en Microsoft Excel. Se utilizó el software estadístico SPSS v.23 para el procesamiento estadístico. Con el cual se determinó la frecuencia de los microorganismos de Escherichia coli y Klebsiella pneumoniae. Se realizó la tabla cruzada para la relación de los microorganismos con el tipo de muestra, el sexo y grupo etario. Se tomaron en cuenta los principios éticos de investigación correspondientes a los estándares de Helsinki, el estudio fue autorizado por la institución y se aseguró la confidencialidad mediante códigos específicos,la información del paciente.
Resultados
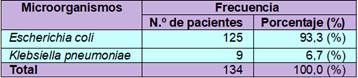
La tabla 1 muestra los dos microorganismos aislados de pacientes que ingresaron al Hospital del Norte. Se realizó el cultivo de un total de 134 muestras de pacientes que ingresaron al hospital del Norte. Los dos microorganismos que se aislaron con mayor prevalencia fue Escherichia coli con 93,3 %, seguido por Klebsiella pneumoniae con 6,7 %.
Tabla 1 Frecuencia de infecciones por Escherichia coli y Klebsiella pneumoniae en pacientes que ingresaron al Hospital del Norte
Fuente: elaboración propia
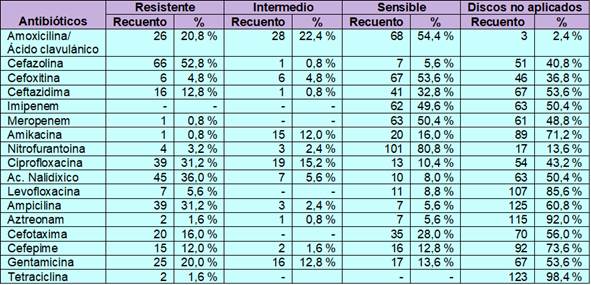
La tabla 2 muestra el perfil de resistencia de Escherichia coli. Se evidencia mayor resistencia a cefazolina 52,8 %, ácido nalidíxico 36 %, ciprofloxacina 31,2 %, ampicilina 31,2 %, gentamicina 20,0 %. Por otro lado, se observa mayor sensibilidad a nitrofurantoína con 80,8 %, seguidos por amoxicilina/ácido clavulánico 54,4 %, cefoxitina 53,6 %, meropenem 50,4 %, imipenem 49,6 %.
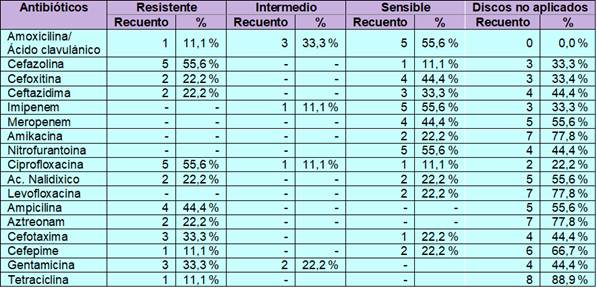
La tabla 3 muestra el perfil de resistencia de Klebsiella pneumoniae. Se evidencia mayor resistencia a cefazolina 55,6 %, ciprofloxacina 55,6 %, ampicilina 44,4 %, cefotaxima 33,3 %, gentamicina 33,3 %. Por otro lado, se observa sensibilidad a amoxicilina/ácido clavulánico 55,6 %, imipenem 55,6 %, nitrofurantoína 55,6 %, cefoxitina 22,2 % y meropenem 44,4 %.
En la tabla 4 muestra los mecanismos de resistencia de Escherichia coli y Klebsiella pneumoniae. El 27,6 % de Escherichia coli produce betalactamasas de espectro extendido (BLEE), el 6,8 % produce betalactamasas de espectro ampliado (BLEA) y 0,7 % producen AMPc. Por otro lado, Klebsiella pneumoniae el 3,0 % produce betalactamasas de espectro extendido (BLEE), el 0,7 % produce betalactamasas de espectro ampliado (BLEA) y el 0,7 % produce AMPc.
Tabla 4 Mecanismos de resistencia de Escherichia coli y Klebsiella pneumoniae
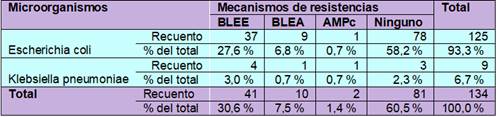
Fuente: elaboración propia
La tabla 5. Se observa los dos microorganismos aislados según el tipo de muestra. Se aislaron Escherichia coli con mayor frecuencia en muestras de orina con 82,1 %, seguidos por líquido abdominal 6,0 %, absceso 2,2 %, secreción vaginal 1,5 %, punta de catéter 0,7 % y secreción escrotal 0,7 %. En cuanto a Klebsiella pneumoniae se aisló con mayor frecuencia en muestras de orina 3,7 %, esputo 1,5 %, Aspirado traqueal 0,7 % y secreción faríngea 0,7 %. La prueba de chi-cuadrado de Pearson mostró un valor de P= 0,000 (P>0,05 a un intervalo de confianza de 95 %), lo que indica que sí existe una asociación o relación significativa entre la frecuencia de estos microorganismos y el tipo de muestra.
Tabla 5 Escherichia coli y Klebsiella pneumoniae aislados según el tipo de muestra
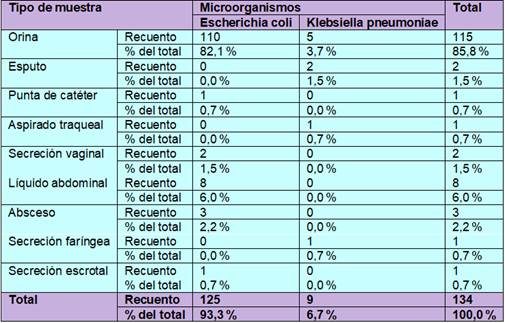
Fuente: elaboración propia
La tabla 6 muestra los dos microorganismos aislados según el sexo de la población de estudio. Se aisló con mayor frecuencia a Escherichia coli en el sexo femenino con el 70,2 % y se aisló 23,1 % en el sexo masculino. En cuanto a Klebsiella pneumoniae se aisló con mayor frecuencia en el sexo masculino con 4,5 % y el 2,2 % en el sexo femenino. La prueba de chi-cuadrado de Pearson mostró un valor de P= 0,007 (P>0,05 a un intervalo de confianza de 95 %), lo que indica que sí existe una asociación o relación significativa entre la frecuencia de estos microorganismos y el sexo.
Tabla 6 Frecuencia de Escherichia coli y Klebsiella pneumoniae según el sexo de los pacientes
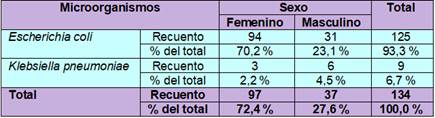
Fuente: Elaboración propia
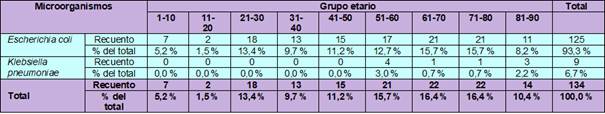
La Tabla 7 se observa los dos microorganismos aislados según el grupo etario de los pacientes. Se encontró con mayor frecuencia a Escherichia coli con 15,7 % entre las edades de 61-70 años y 71-80 años, de igual manera, seguidos de 21-30 años con 13,4 %, 51-60 años con 12,7 %, 41-50 años con 11,2 %, 31-40 años con 9,7 %, 81-90 años con 8,2 %, 1-10 años con 5,2 % y 11-20 años con 1,5 %. En cuanto a Klebsiella pneumoniae se encontró con mayor frecuencia entre los 51-60 años con 3,0 %, seguidos de los 81-90 años con 2,2 % y un 0,7 % entre los 61-70 años, al igual entre los 71-80 años. La prueba de chi-cuadrado de Pearson mostró un valor de P= 0,076 (P>0,05 a un intervalo de confianza de 95 %), lo que indica que no existe una asociación o relación significativa entre la frecuencia de estos microorganismos y el grupo etario.
Discusión
En la presente investigación se analizaron 134 muestras en el laboratorio de microbiología del Hospital del Norte. Los microorganismos que se aislaron con mayor prevalencia fue Escherichia coli con 93,3 %, seguido por Klebsiella pneumoniae con 6,7 %. Estos hallazgos concuerdan con los resultados obtenidos por Carriel et al. (9), quienes aislaron con mayor prevalencia a E. coli con 76,0 %, K. pneumoniae con 5,8 % y otras bacterias patógenas 18,2 %. Al igual que en el estudio realizado por Vidal et al. (10) E. coli fue la bacteria más aislada en un 85,3 % y K. pneumoniae en un 3,1 %, el 11,6 % pertenece a otras bacterias aisladas. Sin embargo, en el estudio realizado por Romero et al. (11), aislaron con mayor prevalencia K. pneumoniae con 37 %, seguido por E. coli con 33 % y el 30 % pertenece a distintos tipos de bacterias.
En cuanto a la resistencia a los diferentes antibióticos, Escherichia coli presentó resistencia a cefazolina 52,8 %%, ácido nalidíxico 36 %, ciprofloxacina 31,2 %, ampicilina 31,2 %, amoxicilina/ácido clavulánico 20,8 %, gentamicina 20,0 %. Y presentó sensibilidad a nitrofurantoína con 80,8 %, amoxicilina/ácido clavulánico 54,4 %, cefoxitina 53,6 %, meropenem 50,4 %, imipenem 49,6 %. La Klebsiella pneumoniae presentó mayor resistencia a cefazolina 55,6 %, ciprofloxacina 55,6 %, ampicilina 44,4 %, cefotaxima 33,3 %, gentamicina 33,3 %. Estos hallazgos concuerdan con el estudio realizado por Cabrera et al. (12). En el cual, Escherichia coli mostró resistencia superior al 60 % a los antibióticos, ácido nalidíxico, cefotaxima, trimetoprim - sulfametoxazol y ceftazidima. La nitrofurantoína 88,8 % y la amikacina presentaron 83,8 % de efectividad. Se observó altos valores de resistencia a ceftazidima, trimetoprim - sulfametoxazol y ácido nalidíxico para Klebsiella pneumoniae. Y amikacina presentó niveles de sensibilidad de 71 %. Por otro lado, en un estudio realizado por Cepero et al. (13)(1) Tanto para E. coli como K. pneumoniae mostraron resistencia a ampicilina/sulbactam, aztreonam, cefotaxima, cefepime, trimetoprim/sulfametoxazol, ciprofloxacina, tetraciclina y gentamicina. Y mostraron sensibilidad a piperacilina/tazobactam, meropenem, cloranfenicol y amikacina, con excepción de la K. pneumoniae que mostró baja sensibilidad a la amikacina.
Escherichia coli presenta mecanismos de resistencia de tipo betalactamasas de espectro extendido (BLEE) 27,6 %, betalactamasas de espectro ampliado (BLEA) 6.7 % y AMPc 0,7 %. Por otro lado, Klebsiella pneumoniae produce (BLEE) 3,0 % y (BLEA) 0,7 % y un 0,7 % de AMPc. Al igual que en el estudio realizado por Castellanos et al. (14) los mecanismos de resistencia que se encontró con mayor frecuencia para E. coli es del tipo BLEE 51,7 % seguido del tipo AMPc 36,3 %, para la Alevilla solo encontró un tipo de mecanismo, el de tipo BLEE 41,3 %.
Los resultados obtenidos por Diaz - Velásquez et al. (6) se asemejan a los resultados obtenidos en esta investigación, donde las infecciones ocasionadas por E. coli fueron más prevalentes en el sexo femenino, con 70,1 % respecto al sexo masculino con 23,1 % y en cuanto al grupo etario, se encontró con mayor frecuencia a Escherichia coli con 15,7 % en el grupo etario de 61-70 años y 71-80 años, seguido de 21-30 años con 13,4 %, 41-50 años con 11,2 % y 81-90 años con 8,2 %.
En conclusión, los microorganismos aislados en pacientes que acudieron al Hospital del Norte durante diciembre 2022 - abril 2023 presentaron una multirresistencia a los antibióticos, en el caso de Escherichia coli presentó resistencia a cefazolina 52,8 %, ácido nalidíxico 36 %, ciprofloxacina 31,2 %, ampicilina 31,2 %, gentamicina 20,0 %. En cuanto a la Klebsiella pneumoniae presentó resistencia a cefazolina 55,6 %, ciprofloxacina 55,6 %, ampicilina 44,4 %, cefotaxima 33,3 %, gentamicina 33,3 %.












 uBio
uBio